RNA: differenze tra le versioni
m Bot: accenti |
Amplio da en.wiki con note (WIP) |
||
| Riga 1: | Riga 1: | ||
{{ |
{{WIP|Adert}} |
||
[[File:Pre-mRNA-1ysv-tubes.png|thumb|right|A hairpin loop from a pre-mRNA. Highlighted are the [[nucleobase]]s (green) and the ribose-phosphate backbone (blue). Note that this is a single strand of RNA that folds back upon itself.]] |
|||
{{F|acidi nucleici|dicembre 2012}} |
|||
[[File:RNA Struktur.png|thumb|upright=1.8|Struttura del RNA]] |
|||
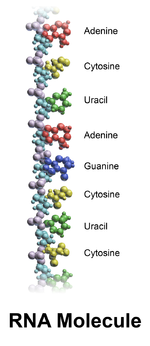
L''''acido ribonucleico''' ('''RNA''') è una [[molecola]] [[polimero|polimerica]] implicata in vari ruoli biologici di [[codice genetico|codifica]], [[Sintesi proteica|decodifica]], [[RNA interference|regolazione]] e l'[[Splicing|espressione]] dei [[gene|geni]]. L'RNA e il [[DNA]] sono [[acidi nucleici]], e, insieme a [[proteine]] e [[carboidrati]], costituiscono le tre principali [[macromolecole]] essenziali per tutte le forme di vita conosciute. Come il DNA, l'RNA è assemblato come una catena di [[nucleotidi]], ma a differenza del DNA è più frequente in natura come un singolo filamento ripiegata su se stesso, piuttosto che un doppio filamento accoppiato. Gli oOrganismi cellulari utilizzano l'[[RNA messaggero]] (mRNA) per trasmettere le informazioni genetiche (mediante le lettere G, U, A, e C per indicare la [[basi azotate]] [[guanina]], [[uracile]], [[adenina]] e [[citosina]]) che dirige la sintesi di proteine specifiche. Molti [[virus (biologia)|virus]] codificano le loro informazioni genetiche utilizzando un [[genoma]] a RNA. |
|||
L''''acido ribonucleico''' ('''RNA''' o '''ARN''') è un [[macromolecola|polimero]] organico, risultante dalla [[polimerizzazione]] di [[ribonucleotide|ribonucleotidi]]. |
|||
Alcune molecole di RNA svolgono un ruolo attivo all'interno delle cellule al fine di [[catalizzatore|catalizzare]] le reazioni biologiche, di controllo dell'[[espressione genica]] o per percepire e comunicare le risposte a segnali cellulari. Uno di questi processi attivi è la [[sintesi proteica]], una funzione universale per cui molecole di mRNA dirigono l'assemblaggio delle proteine nei [[ribosomi]]. Questo processo utilizza le [[molecola|molecole]] di RNA di trasferimento (tRNA) per fornire gli [[amminoacidi]] al ribosoma, dove l'[[RNA ribosomiale]] (rRNA) collega insieme gli amminoacidi per formare le proteine. |
|||
==Descrizione== |
|||
== Confronto con il DNA == |
|||
Chimicamente l'RNA è molto simile al [[DNA]]. Anch'esso è una catena polinucleotidica contenente quattro nucleotidi diversi. Le molecole di RNA differiscono da quelle di DNA perché: |
|||
[[File:Blausen 0773 RNA.png|thumb|left|150px|Bases in an RNA molecule.]] |
|||
[[File:50S-subunit of the ribosome 3CC2.png|thumb|Three-dimensional representation of the [[50S]] ribosomal subunit. RNA is in ochre, protein in blue. The active site is in the middle (red).]] |
|||
La struttura chimica dell'RNA è molto simile a quella del [[DNA]], ma differisce in tre aspetti principali: |
|||
* contengono lo zucchero [[ribosio]] (con un gruppo OH legato al carbonio 2') anziché il [[deossiribosio]] (da qui il nome); |
|||
* una delle [[Base (chimica)|basi]], la [[timina]] (T), è sostituita dall'[[uracile]] (U). In questo caso è l'[[uracile]] la base complementare all'[[adenina]], mentre la [[guanina]] è sempre complementare alla [[citosina]]; |
|||
* Diversamente dal DNA che è a doppio filamento, l'RNA è una molecola a singolo filamento<ref>{{cite web | url=http://learn.genetics.utah.edu/content/molecules/rnamolecule/ | title =RNA: The Versatile Molecule | publisher =[[University of Utah]] | year =2015}}</ref> in molti dei suoi ruoli biologici e ha una catena molto più breve di nucleotidi.<ref>{{cite web | url=http://www.chem.ucla.edu/harding/notes/notes_14C_nucacids.pdf | title =Nucleotides and Nucleic Acids | publisher =[[University of California, Los Angeles]]}}</ref> Tuttavia, l'RNA può, per l'appaiamento delle basi complementari, forma doppie eliche intraelicoidali, come nel caso del tRNA. |
|||
Le molecole di RNA vengono sintetizzate attraverso un processo, conosciuto come [[trascrizione del DNA]], dove un filamento di [[DNA]] viene ricopiato nel corrispondente filamento di RNA. |
|||
* Mentre DNA contiene [[desossiribosio]], l'RNA contiene [[ribosio]]<ref>{{cite book | url=https://books.google.com/books?id=7-UKCgAAQBAJ&pg=PT386&lpg=PT386&dq=dna+contains+deoxyribose+rna+ribose&source=bl&ots=QfJaaVzXYG&sig=PUA6T_MyLGQOEUMRgVsV_qgNpCg&hl=en&sa=X&ved=0CEcQ6AEwCDgKahUKEwiBl-KBgMfHAhXJVh4KHaVECHI#v=onepage&q=dna%20contains%20deoxyribose%20rna%20ribose&f=false | title =Analysis of Chromosomes | author =R.N. Shukla | isbn =9789384568177}}</ref> (nel desossiribosio non vi è un [[gruppo ossidrile]] attaccato all'[[anello pentoso]] in posizione 2'). Questi gruppi ossidrilici rendono l'RNA meno stabile del DNA perché è più incline all'[[idrolisi]]. |
|||
* La base complementare al DNA dell'[[adenina]] è la [[timina]], mentre nell'RNA è l'[[uracile]], che è una forma [[metilazione|metilata]] di timina.<ref name=Biochemistry/> |
|||
Come il DNA, l'RNA è biologicamente più attivo, tra l'[[mRNA]], il [[tRNA]], l'[[rRNA]], l'[[snRNA]] e altri [[RNA non codificante|RNA non codificanti]], contengono sequenze auto-complementari che permettono a parti dell'RNA di ripiegarsi<ref>{{Cite journal|author=I. Tinoco|author2=C. Bustamante|last-author-amp=yes|title=How RNA folds| |
|||
journal=J. Mol. Biol.|volume=293|pages=271–281|date=1999|doi=10.1006/jmbi.1999.3001|issue=2|pmid=10550208|postscript=.}}</ref> e collegarsi andando a formando doppie eliche. L'analisi di questi RNA ha rivelato che essi sono altamente strutturati. A differenza del DNA, le loro strutture non sono costituite da lunghe doppie eliche, ma piuttosto da raccolte di eliche riunite in strutture simili a proteine. In questo modo, l'RNA può realizzare la [[catalisi]] chimica, come gli [[enzimi]].<ref>{{cite journal|author=Higgs PG|title=RNA secondary structure: physical and computational aspects|journal=Quarterly Reviews of Biophysics|date=2000|volume=33|pages=199–253|doi=10.1017/S0033583500003620|pmid = 11191843|issue=3 }}</ref> Ad esempio, la determinazione della struttura del [[ribosoma]], un enzima che catalizza la formazione di un [[legame peptidico]], ha rivelato che il suo [[sito attivo]] è composto interamente di RNA.<ref name=ribosome_activity>{{cite journal|author=Nissen P|author2=Hansen J|author3=Ban N|author4=Moore PB|author5=Steitz TA|title=The structural basis of ribosome activity in peptide bond synthesis|journal=Science|date=2000|volume=289|issue=5481|pages=920–30|doi=10.1126/science.289.5481.920|pmid = 10937990|bibcode = 2000Sci...289..920N }}</ref> |
|||
== Struttura == |
|||
[[File:Piwi-siRNA-basepairing.png|thumb|right|230px|Watson-Crick base pairs in a [[siRNA]] (hydrogen atoms are not shown)]] |
|||
Ogni nucleotide dell'RNA contiene uno zucchero [[ribosio]], con il carbonio numerato da 1' a 5'. Una base è collegato alla posizione 1', in generale, l'[[adenina]] (A), la [[citosina]] (C), la [[guanina]] (G) o l'[[uracile]] (U). L'[[adenina]] e la [[guanina]] sono [[purine]], la [[citosina]] e l'[[uracile]] sono [[pirimidine]]. Un [[gruppo fosfato]] è attaccato alla posizione 3' di un ribosio e alla posizione 5' del successivo. I gruppi fosfato hanno una carica negativa a un [[pH]] fisiologico, rendendo l'RNA una molecola carica (polianione). Le basi formano [[legame idrogeno|legami idrogeno]] tra la [[citosina]] e la [[guanina]], tra l'[[adenina]] e l'[[uracile]] e fra la [[guanina]] e l'[[uracile]].<ref name="pmid15561141"/> Tuttavia, altre interazioni sono possibili, come ad esempio un gruppo di basi adenina legate tra loro,<ref>{{cite book|title=RNA biochemistry and biotechnology|author=Barciszewski J|author2=Frederic B|author3=Clark C|date=1999|pages=73–87|publisher=Springer|isbn=0-7923-5862-7|oclc=52403776}}</ref> o un tetragiro GNRA che possiede una coppia di base di guanina-adenina.<ref name="pmid15561141">{{cite journal|author = Lee JC|author2 = Gutell RR|title = Diversity of base-pair conformations and their occurrence in rRNA structure and RNA structural motifs|journal = J. Mol. Biol.|volume = 344|issue = 5|pages = 1225–49|date = 2004|doi = 10.1016/j.jmb.2004.09.072|pmid=15561141}}</ref> |
|||
[[File:RNA chemical structure.GIF|thumb|left|Chemical structure of RNA]] |
|||
Una caratteristica strutturale importante dell'RNA che lo distingue dal DNA è la presenza di un [[gruppo idrossile]] in posizione 2' dello zucchero ribosio. La presenza di questo gruppo funzionale provoca l'adozione di un elica a forma di A rispetto alla forma B più comunemente osservata nel DNA.<ref>{{cite journal|author=Salazar M|author2=Fedoroff OY|author3=Miller JM|author4=Ribeiro NS|author5=Reid BR|title=The DNA strand in DNAoRNA hybrid duplexes is neither B-form nor A-form in solution|journal=Biochemistry|date=1992|volume=32|issue=16|pages=4207–15|pmid=7682844|doi=10.1021/bi00067a007}}</ref> Questo si traduce in un solco maggiore molto profondo e stretto e un solco minore superficiale e largo.<ref>{{cite journal|author=Hermann T|author2=Patel DJ|title=RNA bulges as architectural and recognition motifs|journal=Structure|date=2000|volume=8|issue=3|pages=R47–R54|doi=10.1016/S0969-2126(00)00110-6|pmid = 10745015 }}</ref> Una seconda conseguenza della presenza del gruppo 2'-idrossile è che nelle regioni conformazionalmente flessibili di una molecola di RNA (cioè non coinvolte nella formazione di una doppia elica), può attaccare chimicamente il legame fosfodiestere adiacente e fendere la dorsale.<ref>{{cite journal|author=Mikkola S|author2=Stenman E|author3=Nurmi K|author4=Yousefi-Salakdeh E|author5=Strömberg R|author6=Lönnberg H|title=The mechanism of the metal ion promoted cleavage of RNA phosphodiester bonds involves a general acid catalysis by the metal aquo ion on the departure of the leaving group|journal=Perkin transactions 2|date=1999|pages=1619–26|doi=10.1039/a903691a|issue=8}}</ref> |
|||
[[File:Ciliate telomerase RNA.JPG|thumb|[[Secondary structure]] of a [[telomerase RNA]].]] |
|||
L'RNA è trascritto con solo quattro basi (adenina, citosina, guanina e uracile),<ref>{{cite book|title=Clinical gene analysis and manipulation: Tools, techniques and troubleshooting|author=Jankowski JAZ|author2=Polak JM|date=1996|page=14|publisher=Cambridge University Press|isbn=0-521-47896-0|oclc=33838261}}</ref> ma queste basi e zuccheri possono essere modificati in numerosi modi, come gli RNA maturi. La [[pseudouridina]] (Ψ), in cui il legame tra uracile e ribosio passa da un legame C-N ad un legame C-C, e la [[ribotimidina]] (T) si trovano in vari luoghi (i più importanti nel ciclo TΨC di tRNA ).<ref>{{cite journal|author=Yu Q|author2=Morrow CD|title=Identification of critical elements in the tRNA acceptor stem and TΨC loop necessary for human immunodeficiency virus type 1 infectivity|journal=J Virol|date=2001|volume=75|issue=10|pages=4902–6|doi=10.1128/JVI.75.10.4902-4906.2001|pmid = 11312362|pmc=114245 }}</ref> Un altro notevole base modificata è ipoxantina, una base adenina deaminato cui nucleosidici è chiamato inosina (I). Inosina gioca un ruolo chiave nella ipotesi oscillazione del codice genetico.<ref>{{cite journal|author=Elliott MS|author2=Trewyn RW|title=Inosine biosynthesis in transfer RNA by an enzymatic insertion of hypoxanthine|journal=J. Biol. Chem.|date=1983|volume=259|issue=4|pages=2407–10|pmid=6365911}}</ref> |
|||
Vi sono più di 100 altri nucleosidi modificati presenti in natura.<ref>{{cite journal|last=Cantara|first=WA|author2=Crain, PF |author3=Rozenski, J |author4=McCloskey, JA |author5=Harris, KA |author6=Zhang, X |author7=Vendeix, FA |author8=Fabris, D |author9= Agris, PF |title=The RNA Modification Database, RNAMDB: 2011 update|journal=Nucleic Acids Research|date=January 2011|volume=39|issue=Database issue|pages=D195–201|pmid=21071406|doi=10.1093/nar/gkq1028|pmc=3013656}}</ref> La più grande diversità strutturale di modifiche può essere trovato nel tRNA,<ref>{{cite book|title=TRNA: Structure, biosynthesis, and function|author=Söll D|author2=RajBhandary U|date=1995|page=165|publisher=ASM Press|isbn=1-55581-073-X|oclc=183036381 30663724}}</ref> mentre la pseudouridina e i nucleosidi spesso presenti nell'rRNA sono i più comuni.<ref>{{cite journal|author=Kiss T|title=Small nucleolar RNA-guided post-transcriptional modification of cellular RNAs|journal=The EMBO Journal|date=2001|volume=20|pages=3617–22|doi=10.1093/emboj/20.14.3617|pmid = 11447102|pmc=125535|issue=14}}</ref> I ruoli specifici di molte di queste modifiche dell'RNA non sono pienamente comprese. Tuttavia, è da notare che, nell'RNA ribosomiale molte delle modificazioni post-trascrizionali si verificano nelle regioni altamente funzionali, come il centro della peptidil-transferasi e l'interfaccia subunità, che implica che sono importanti per la funzione normale.<ref>{{cite journal|author=King TH|author2= Liu B|author3=McCully RR|author4=Fournier MJ|title=Ribosome structure and activity are altered in cells lacking snoRNPs that form pseudouridines in the peptidyl transferase center|journal=Molecular Cell|date=2002|volume=11|issue=2|pages=425–35|doi=10.1016/S1097-2765(03)00040-6|pmid = 12620230}}</ref> |
|||
La forma funzionale di molecole a singolo filamento di RNA, come le proteine, richiede spesso una [[struttura terziaria]] specifica. L'impalcatura di questa struttura è fornita da elementi strutturali secondari che sono [[legami idrogeno]] all'interno della [[molecola]]. Questo porta a diversi "domini" riconoscibili della struttura secondaria.<ref>{{cite journal|author=Mathews DH|author2=Disney MD|author3=Childs JL|author4=Schroeder SJ|author5=Zuker M|author6=Turner DH|title=Incorporating chemical modification constraints into a dynamic programming algorithm for prediction of RNA secondary structure|journal=Proc. Natl. Acad. Sci. USA|date=2004|volume=101|issue=19|pages=7287–92|doi=10.1073/pnas.0401799101|pmid = 15123812|pmc=409911|bibcode=2004PNAS..101.7287M}}</ref> Dal momento che l'RNA è carico, sono necessari ioni metallici, come [[magnesio|Mg<sup>2+</sup>]] per stabilizzare molte strutture secondarie e terziarie.<ref>{{cite journal|author=Tan ZJ|author2=Chen SJ|title=Salt dependence of nucleic acid hairpin stability|journal=Biophys. J.|volume=95|pages=738–52|date=2008|pmid=18424500|doi=10.1529/biophysj.108.131524|issue=2|pmc=2440479|bibcode = 2008BpJ....95..738T }}</ref> |
|||
== Sintesi == |
|||
La [[sintesi]] dell'RNA è solitamente catalizzata da un enzima, l'[[RNA-polimerasi]], utilizzando il DNA come stampo, un processo noto come [[trascrizione]]. La trascrizione inizia con il legame dell'enzima ad una [[Promotore (biologia)|sequenza promotrice]] nel DNA (di solito "a monte" di un gene). Il DNA a doppia elica viene svolto dall'attività [[elicasi ]]dell'enzima. L'enzima procede poi lungo il filamento stampo nella direzione da 3' a 5', sintetizzando una molecola di RNA complementare nell'allungamento che si verifica da 5' a 3'. La sequenza di DNA determina anche il luogo dove si verifica la cessazione della sintesi di RNA.<ref>{{cite journal|author=Nudler E|author2=Gottesman ME|title=Transcription termination and anti-termination in E. coli|journal=Genes to Cells|date=2002|volume=7|pages=755–68|doi=10.1046/j.1365-2443.2002.00563.x|pmid = 12167155|issue=8 }}</ref> |
|||
La prima trascrizione dell'RNA è spesso modificata da enzimi dopo la trascrizione. Ad esempio, un [[poliadenilazione]] e una [[rivestimento in 5']] vengono aggiunti a un [[pre-mRNA]] [[eucariote]] e gli [[Introne|introni]] sono rimossi dal [[spliceosoma]]. |
|||
Vi sono anche una serie di [[RNA polimerasi RNA-dipendente]] che utilizzano l'RNA come stampo per la sintesi di un nuovo filamento di RNA. Per esempio, un numero di [[virus (biologia)|virus]] a RNA (come il ''[[poliovirus]]'') utilizzano questo tipo di enzima per replicare il loro materiale genetico.<ref>{{cite journal|author=Jeffrey L Hansen|author2=Alexander M Long|author3=Steve C Schultz|title=Structure of the RNA-dependent RNA polymerase of poliovirus|journal=Structure|date=1997|volume=5|issue=8|pages=1109–22|doi=10.1016/S0969-2126(97)00261-X|pmid = 9309225 }}</ref> Inoltre, l'RNA polimerasi RNA-dipendente è parte del percorso di [[RNA interference|interferenza dell'RNA]] in molti organismi.<ref>{{cite journal|author=Ahlquist P|title=RNA-Dependent RNA Polymerases, Viruses, and RNA Silencing|journal=Science|date=2002|volume=296|issue=5571|pages=1270–73|doi=10.1126/science.1069132|pmid = 12016304 |bibcode = 2002Sci...296.1270A }}</ref> |
|||
== Tipi di RNA == |
|||
Vi sono tre tipi di RNA comuni a tutti gli organismi [[cellula]]ri: |
Vi sono tre tipi di RNA comuni a tutti gli organismi [[cellula]]ri: |
||
| Riga 25: | Riga 55: | ||
* [[snRNA]] (piccolo RNA nucleare) necessario per la maturazione dell'HnRna. |
* [[snRNA]] (piccolo RNA nucleare) necessario per la maturazione dell'HnRna. |
||
La sintesi dell'RNA è molto simile a quella del DNA. La [[RNA polimerasi]] non richiede però un innesco. La trascrizione può iniziare solo presso una sequenza detta promotore e termina in presenza di altre sequenze particolari. |
La sintesi dell'RNA è molto simile a quella del DNA. La [[RNA polimerasi]] non richiede però un innesco. La trascrizione può iniziare solo presso una sequenza detta promotore e termina in presenza di altre sequenze particolari. È stata avanzata l'ipotesi che l'RNA abbia assunto un ruolo chiave negli organismi primitivi prima del DNA (RNA world o [[Ipotesi del mondo a RNA|mondo a RNA]]). A favore di tale ipotesi c'è la capacità catalitica di alcune molecole di RNA (''[[ribozima|ribozimi]]''). Sull'mRNA viene trascritta l'informazione genetica che poi verrà utilizzata per svariati usi. |
||
=== Panoramica === |
|||
È stata avanzata l'ipotesi che l'RNA abbia assunto un ruolo chiave negli organismi primitivi prima del DNA (RNA world o [[Ipotesi del mondo a RNA|mondo a RNA]]). A favore di tale ipotesi c'è la capacità catalitica di alcune molecole di RNA (''[[ribozima|ribozimi]]''). |
|||
L'[[RNA messaggero]] (mRNA) è l'RNA che trasporta le informazioni dal DNA al [[ribosoma]], i siti della [[sintesi proteica]] (traduzione) nella [[cellula]]. La sequenza codificante dell'mRNA determina la [[sequenza aminoacidica]] della [[proteina]] che viene prodotta.<ref name=The_Cell/> Tuttavia, molti RNA non possiedono codice per le proteine (circa il 97% della produzione trascrizionale non codifica proteine negli eucarioti<ref>{{cite journal|author=Mattick JS|author2=Gagen MJ|title=The evolution of controlled multitasked gene networks: the role of introns and other noncoding RNAs in the development of complex organisms|journal=Mol. Biol. Evol.|volume=18|issue=9|pages=1611–30|date=1 September 2001|pmid=11504843|url=http://mbe.oxfordjournals.org/cgi/pmidlookup?view=long&pmid=11504843|doi=10.1093/oxfordjournals.molbev.a003951 }}</ref><ref>{{cite journal|date=2001|title=Noncoding RNAs: the architects of eukaryotic complexity|journal=EMBO Reports|volume=2|pages=986–91|url=http://www.nature.com/embor/journal/v2/n11/full/embor291.html|doi=10.1093/embo-reports/kve230|pmid=11713189|last1=Mattick|first1=JS|issue=11|pmc=1084129}}</ref><ref>{{cite journal|author=Mattick JS|title=Challenging the dogma: the hidden layer of non-protein-coding RNAs in complex organisms|journal=BioEssays : News and Reviews in Molecular, Cellular and Developmental Biology|volume=25|issue=10|pages=930–9|date=October 2003|pmid=14505360|doi=10.1002/bies.10332|url=http://www.imb-jena.de/jcb/journal_club/mattick2003.pdf}}</ref><ref>{{cite journal|author=Mattick JS |title=The hidden genetic program of complex organisms |journal=Scientific American |volume=291 |issue=4 |pages=60–7 |date=October 2004 |pmid=15487671 |doi=10.1038/scientificamerican1004-60 |url=http://www.sciam.com/article.cfm?articleID=00045BB6-5D49-1150-902F83414B7F4945 |deadurl=yes |archiveurl=https://web.archive.org/20150208155941/http://www.sciam.com/article.cfm?articleID=00045BB6-5D49-1150-902F83414B7F4945 |archivedate=February 8, 2015 }}</ref>). |
|||
Sull'mRNA viene trascritta l'informazione genetica che poi verrà utilizzata per svariati usi. |
|||
Questi cosiddetti RNA non codificanti ("ncRNA") possono essere codificati dai propri geni (geni RNA), ma possono anche derivare da [[introne|introni]] di mRNA.<ref name=transcriptome/> Gli esempi più importanti di RNA non codificanti sono l'[[RNA di trasferimento]] (tRNA) e l'[[RNA ribosomiale]] (rRNA), entrambi i quali sono coinvolti nel processo di traduzione.<ref>{{cite journal|author=Rossi JJ|title=Ribozyme diagnostics comes of age|journal=Chemistry & Biology|date=2004|volume=11|issue=7|pages=894–95|doi=10.1016/j.chembiol.2004.07.002|pmid=15271347}}</ref> Vi sono anche RNA non codificanti coinvolti nella [[regolazione genica]], nel processo dell'RNA e altri ruoli. Alcuni RNA sono in grado di [[catalisi|catalizzare]] le [[reazioni chimiche]], come il taglio e [[Ligasi|legatura]] altre molecole di RNA[32] e la catalisi di formazione del [[legame peptidico]] nel ribosoma;<ref name=ribosome_activity/> questi sono conosciuti come [[ribozima|ribozimi]]. |
|||
== Sintesi == |
|||
=== Nella traduzione === |
|||
Questo processo è molto diverso tra gli ''eucarioti'' e i ''procarioti.'' |
|||
Negli eucarioti la sintesi avviene nel nucleo attraverso tre diversi enzimi detti [[RNA polimerasi]] (nei procarioti ce n'è solo una). Sono parzialmente diverse tra loro, infatti hanno alcune subunità in comune e altre uniche per la loro [[specie]]. Ciò è anche dovuto al fatto che esse producono tre tipi diversi di RNA. |
|||
L'[[RNA messaggero]] (mRNA) trasporta le informazioni di una sequenza proteica ai ribosomi, i luoghi della cellula deputati alla [[sintesi proteica]]. L'informazione è codificata in in modo che ad ogni tre nucleotidi ([[codone]]) corrispondano un [[amminoacido.]] Nelle cellule eucariotiche, una volta che il precursore mRNA (pre-mRNA) è stata trascritto dal DNA, si trasforma per diventare mRNA. In questa fase vengon rimosse le sezioni non codificanti del pre-mRNA. L'mRNA viene poi spostato dal [[nucelo cellulare|nucleo]] al [[citoplasma]], dove arriva ai ribosomi e quindi viene tradotto nella corrispondente proteina grazie all'aiuto del [[tRNA]]. Nelle cellule procariote, che non hanno nucleo e compartimenti nel citoplasma, l'mRNA può legarsi ai ribosomi mentre viene trascritto dal DNA. Dopo un certo periodo di tempo il messaggio degrada nelle sue componenti nucleotidi con l'assistenza della [[ribonucleasi]].<ref name=The_Cell/> |
|||
Subunità centrali: |
|||
rpb1, rpb2, rpb3, sono richiesti per l'attività enzimatica; |
|||
L'[[RNA di trasferimento]] (tRNA) è una piccola catena di RNA composta da circa 80 nucleotidi che trasferisce uno specifico aminoacido ad una catena [[polipeptide|polipeptidica]] crescente nel sito ribosomiale della sintesi proteica durante la traduzione. Esso possiede siti per l'attacco di aminoacidi e una regione [[anticodone]] per il riconoscimento del codone che si lega ad una sequenza specifica sulla catena dell'RNA messaggero attraverso [[legame idrogeno|legami idrogeno]].<ref name=transcriptome/> |
|||
Subunità comuni: |
|||
rpb5, rpb6, rpb8, rpb10, rpb12 processo trascrizionale; |
|||
L'[[RNA ribosomiale]] (rRNA) è il componente [[catalizzatore|catalitico]] dei ribosomi. Negli eucarioti, i ribosomi contengono quattro diverse molecole di rRNA: 18S, 5.8S, 28S e 5S rRNA; tre di esse sono sintetizzate nel [[nucleolo]]. Nel citoplasma, RNA ribosomiale e le proteine si combinano per formare un nucleoproteina chiamato ribosoma. Il ribosoma lega l'mRNA ed effettua la sintesi proteica. Diversi ribosomi possono essere collegati ad un singolo mRNA in qualsiasi momento.<ref name=The_Cell>{{cite book|title=The Cell: A Molecular Approach|edition=3rd|author=Cooper GC|author2=Hausman RE|date=2004|pages=261–76, 297, 339–44|publisher=Sinauer|isbn=0-87893-214-3|oclc=174924833 52121379 52359301 56050609}}</ref> Quasi tutti gli RNA che si trovano in una tipica cellula eucariotica sono rRNA. |
|||
subunità non essenziali: |
|||
rpb4, rpb9 non sono richieste per attività trascrizionale; |
|||
rpb7, rpb11,sono richieste per l'attività polimerasica; |
|||
L'[[RNA transfer-messaggero]] (tmRNA) si trova in molti [[batteri]] e [[plastidi]]. Si occupa di marcare le proteine codificate dall'mRNA che mancano di codoni di stop per la degradazione e impedisce il blocco del ribosoma.<ref>{{cite journal|author=Gueneau de Novoa P, Williams KP|title=The tmRNA website: reductive evolution of tmRNA in plastids and other endosymbionts|journal=Nucleic Acids Res|volume=32|issue=Database issue|pages=D104–8|date=2004|pmid=14681369|doi=10.1093/nar/gkh102|pmc=308836|last2=Williams}}</ref> |
|||
* La RNA polimerasi I produce gli rRNA 5,8 S, 18 S, 28 S nel nucleolo |
|||
* La RNA polimerasi II produce mRNA e piccoli RNA stabili che servono a formare gli [[snRNP]] nel nucleo |
|||
* La RNA polimerasi III produce piccoli RNA stabili, tRNA e l'RNA 5 S sempre nel nucleo. |
|||
=== RNA regolatori === |
|||
La RNA polimerasi più conosciuta è la RNA polimerasi II perché produce l'mRNA, che servirà poi ai ribosomi per sintetizzare le proteine. |
|||
Diversi tipi di RNA sono in grado di sottoregolare l'[[espressione genica]] per essere complementari ad una parte di un mRNA o il gene ddl DNA.<ref name=Carthew2009>{{cite journal |last = Carthew| first = RW | author2 = Sontheimer, EJ.|title = Origins and Mechanisms of miRNAs and siRNAs.| journal = Cell |pmid = 19239886|url = http://www.ncbi.nlm.nih.gov/pmc/articles/PMC2675692/|pmc = 2675692 | doi = 10.1016/j.cell.2009.01.035 | accessdate = 2009-02-20 }}</ref><ref name=Liang2013>{{cite journal |last = Liang| first = Kung-Hao | author2 = Yeh, Chau-Ting|title = A gene expression restriction network mediated by sense and antisense Alu sequences located on protein-coding messenger RNAs.| journal = BMC Genomics |pmid = 23663499|url = http://www.biomedcentral.com/1471-2164/14/325 |pmc = 3655826 | doi = 10.1186/1471-2164-14-325 | accessdate = 2013-05-11 }}</ref> I [[microRNA]] (miRNA; 21-22 nt). Si trovano negli eucarioti e agiscono tramite l'[[RNA interference]] (RNAi), dove un complesso effettore di miRNA e enzimi in grado di scindere l'mRNA complementare, blocca la traduzione dell'mRNA vengano tradotti o accelerara la sua degradazione.<ref>{{cite journal|author=Wu L|author2=Belasco JG|title=Let me count the ways: mechanisms of gene regulation by miRNAs and siRNAs|journal=Mol. Cell|volume=29|issue=1|pages=1–7|date=January 2008|pmid=18206964|doi=10.1016/j.molcel.2007.12.010}}</ref><ref>{{cite journal|author=Matzke MA|author2=Matzke AJM|title=Planting the seeds of a new paradigm|journal=PLoS Biology|date=2004|volume=2|issue=5|doi=10.1371/journal.pbio.0020133|pages = e133|pmid = 15138502|pmc=406394}}</ref> |
|||
Mentre ''[[Short interfering RNA]]'' (siRNA, 20-25 nt) vengono spesso prodotti in seguito alla rottura di RNA virale, vi sono anche fonti endogene di siRNA.<ref>{{cite journal|author=Vazquez F|author2=Vaucheret H|author3=Rajagopalan R|author4=Lepers C|author5=Gasciolli V|author6=Mallory AC|author7=Hilbert J|author8=Bartel DP|author9=Crété P|title=Endogenous ''trans''-acting siRNAs regulate the accumulation of ''Arabidopsis'' mRNAs|journal=Molecular Cell|date=2004|volume=16|issue=1|pages= 69–79|doi=10.1016/j.molcel.2004.09.028|pmid = 15469823}}</ref><ref>{{cite journal|author=Watanabe T|title=Endogenous siRNAs from naturally formed dsRNAs regulate transcripts in mouse oocytes|journal=Nature|volume=453|issue=7194|pages=539–43|date=May 2008|pmid=18404146|doi=10.1038/nature06908|last12=Sakaki|first12=Y|last13=Sasaki|first13=H|bibcode = 2008Natur.453..539W |name-list-format=vanc|author2= Totoki Y|author3= Toyoda A|displayauthors=3|author4=Kaneda M|last5=Kuramochi-Miyagawa|first5=Satomi|last6=Obata|first6=Yayoi|last7=Chiba|first7=Hatsune|last8=Kohara|first8=Yuji|last9=Kono|first9=Tomohiro|last10=Nakano|first10=Toru|last11=Surani|first11=M. Azim}}</ref> I siRNA agiscono attraverso l'interferenza dell'RNA in modo simile ai miRNA. Alcuni miRNA e siRNA possono causare la [[Metilazione del DNA|metilazione]] dei geni ''target'', diminuendo o aumentando in tal modo la trascrizione di questi geni.<ref>{{cite journal|author=Sontheimer EJ|author2=Carthew RW|title=Silence from within: endogenous siRNAs and miRNAs|journal=Cell|volume=122|issue=1|pages=9–12|date=July 2005|pmid=16009127|doi=10.1016/j.cell.2005.06.030}}</ref><ref>{{cite journal|author=Doran G|title=RNAi – Is one suffix sufficient?|journal=Journal of RNAi and Gene Silencing|date=2007|volume=3|issue=1|pages=217–19|url=http://libpubmedia.co.uk/RNAiJ-Issues/Issue-5/Doran.htm}}</ref><ref>{{cite journal|author=Pushparaj PN|author2=Aarthi JJ|author3=Kumar SD|author4=Manikandan J|title=RNAi and RNAa — The Yin and Yang of RNAome|journal=Bioinformation|volume=2|issue=6|pages=235–7|date=2008|pmid=18317570|pmc=2258431|doi=10.6026/97320630002235}}</ref> Gli animali possiedono i ''[[piwi-interacting RNA]]'' (piRNA; 29-30 nt) che sono attivi nella linea germinale delle cellule e sono ritenuti essere una difesa contro i [[trasposoni]] e svolgere un ruolo nella [[gametogenesi]].<ref name=fruitfly_piRNA>{{cite journal|author=Horwich MD|author2=Li C|author3=Matranga C|author4=Vagin V|author5=Farley G|author6=Wang P|author7=Zamore PD|title=The ''Drosophila'' RNA methyltransferase, DmHen1, modifies germline piRNAs and single-stranded siRNAs in RISC|journal=Current Biology|date=2007|volume=17|pages=1265–72|doi=10.1016/j.cub.2007.06.030|pmid = 17604629|issue=14 }}</ref><ref>{{cite journal|author=Girard A|author2=Sachidanandam R|author3=Hannon GJ|author4=Carmell MA|title=A germline-specific class of small RNAs binds mammalian Piwi proteins|journal=Nature|date=2006|volume=442|pages=199–202|doi=10.1038/nature04917|pmid = 16751776|issue=7099|bibcode = 2006Natur.442..199G }}</ref> |
|||
L'rRNA 18 S più 35 proteine compresse andrà a formare la subunità ribosomiale minore. |
|||
Gli rRNA 5 S, 5,8 S e 28 S più 50 proteine andranno a formare la subunità ribosomiale maggiore. |
|||
Molti [[procarioti]] hanno l'RNA [[CRISPR]], un sistema di regolamentazione simile all'interferenza dell'RNA.<ref>{{cite journal|author=Horvath P|author2=Barrangou R|title=CRISPR/Cas, the Immune System of Bacteria and Archaea|journal=Science|volume=327|pages=167–70|date=2010|pmid=20056882|doi=10.1126/science.1179555|url=http://www.sciencemag.org/cgi/content/abstract/327/5962/167|issue=5962|bibcode = 2010Sci...327..167H }}</ref> Gli [[RNA antisenso]] sono diffusi, molti regolano un [[gene]], ma alcuni sono attivatori della trascrizione.<ref>{{cite journal|author=Wagner EG|author2=Altuvia S|author3=Romby P|title=Antisense RNAs in bacteria and their genetic elements|journal=Adv Genet|date=2002|volume=46|pages=361–98|pmid=11931231|doi=10.1016/S0065-2660(02)46013-0|series=Advances in Genetics|isbn=9780120176465}}</ref> Un modo in cui l'[[RNA antisenso]] può agire è legandosi ad un mRNA, formando un RNA a doppio filamento che viene degradato enzimaticamente.<ref>{{cite book|author=Gilbert SF|title=Developmental Biology|edition=7th|publisher=Sinauer|isbn=0-87893-258-5|pages=101–3|date=2003|oclc=154656422 154663147 174530692 177000492 177316159 51544170 54743254 59197768 61404850 66754122}}</ref> Vi sono molti lunghi RNA non codificanti che regolano i geni negli eucarioti,<ref>{{cite journal|author=Amaral PP|author2=Mattick JS|title=Noncoding RNA in development|journal=Mammalian genome : official journal of the International Mammalian Genome Society|volume= 19|issue= 7–8|pages= 454–92|date=October 2008|pmid=18839252|doi=10.1007/s00335-008-9136-7}}</ref> uno di questi è l'Xist, che si trova nel [[cromosoma X]] nei mammiferi di sesso femminile e serve per inattivarlo.<ref>{{cite journal|author=Heard E|author2=Mongelard F|author3=Arnaud D|author4=Chureau C|author5=Vourc'h C|author6=Avner P|title=Human ''XIST'' yeast artificial chromosome transgenes show partial X inactivation center function in mouse embryonic stem cells|journal=Proc. Natl. Acad. Sci. USA|date=1999|volume=96|issue=12|pages=6841–46|pmid=10359800|doi = 10.1073/pnas.96.12.6841|pmc=22003|bibcode = 1999PNAS...96.6841H }}</ref> |
|||
=== Anomalie dell'RNA === |
|||
=== Nell'elaborazione === |
|||
Molti RNA sono coinvolti nel modificare altri RNA. Gli [[introni]] sono "[[Splicing|montati]]" in pre-mRNA grazie agli [[spliceosomi]] che contengono diversi piccoli [[RNA nucleari]] (snRNA),<ref name=Biochemistry/> o gli introni possono essere ribozimi che vengono collegati da loro stessi.<ref>{{cite journal|author=Steitz TA|author2=Steitz JA|title=A general two-metal-ion mechanism for catalytic RNA|journal=Proc. Natl. Acad. Sci. U.S.A.|volume=90|issue=14|pages=6498–502|date=1993|pmid=8341661|doi = 10.1073/pnas.90.14.6498|pmc=46959|bibcode = 1993PNAS...90.6498S }}</ref> L'RNA può anche essere alterato da avere i suoi nucleotidi modificati con altri nucleotidi di [[adenina|A]], [[citosina|C]], [[guanina|G]] e [[uracile|U]]. Negli [[eucarioti]], le modifiche dei nucleotidi di RNA sono in genere diretti da [[Small nucleolar RNA|piccoli RNA nucleolari]] (snoRNA; 60-300 nt),<ref name=transcriptome>{{cite book|title=Mining the transcriptome – methods and applications|url=http://kth.diva-portal.org/smash/get/diva2:10803/FULLTEXT01|author=Wirta W|date=2006|isbn=91-7178-436-5|publisher=School of Biotechnology, Royal Institute of Technology|location=Stockholm|oclc=185406288}}</ref> riscontrati nel [[nucleolo]] e nei [[corpi di Cajal]]. Gli enzimi quindi eseguono la modifica del nucleotide. rRNA e tRNA sono ampiamente modificati, ma anche gli snRNAs e l'mRNA possono anche essere bersaglio di modifiche di base.<ref>{{cite journal|author=Xie J|author2=Zhang M|author3=Zhou T|author4= Hua X|author5=Tang L|author6=Wu W|title=Sno/scaRNAbase: a curated database for small nucleolar RNAs and cajal body-specific RNAs|journal=Nucleic Acids Res|volume=35|pages=D183–7|date=2007|pmid=17099227|doi=10.1093/nar/gkl873|issue=Database issue|pmc=1669756}}</ref><ref>{{cite journal|author=Omer AD|author2=Ziesche S|author3=Decatur WA|author4=Fournier MJ|author5=Dennis PP|title=RNA-modifying machines in archaea|journal=Molecular Microbiology|date=2003|volume=48|issue=3|pages=617–29|doi=10.1046/j.1365-2958.2003.03483.x|pmid = 12694609}}</ref> L'RNA può anche essere demetilato.<ref>{{cite journal|author = Cavaillé J|author2 = Nicoloso M|author3 = Bachellerie JP|title = Targeted ribose methylation of RNA in vivo directed by tailored antisense RNA guides|journal = Nature|volume = 383|issue = 6602|pages = 732–5|date = 1996|pmid = 8878486|doi = 10.1038/383732a0 |bibcode = 1996Natur.383..732C }}</ref><ref>{{cite journal|author = Kiss-László Z|author2 = Henry Y|author3 = Bachellerie JP|author4 = Caizergues-Ferrer M|author5 = Kiss T|title = Site-specific ribose methylation of preribosomal RNA: a novel function for small nucleolar RNAs|journal = Cell|volume = 85|issue = 7|pages = 1077–88|date = 1996|pmid = 8674114|doi = 10.1016/S0092-8674(00)81308-2 }}</ref> |
|||
=== Genoma RNA === |
|||
Come il DNA, l'RNA può trasportare le informazioni genetiche. I virus a RNA possiedono genomi composti da RNA che codificano un numero di proteine. Il genoma virale viene replicato da alcune di queste proteine, mentre altre proteine proteggono il genoma quando la particella virale si sposta in una nuova cellula ospite. I [[viroidi]] sono un altro gruppo di agenti patogeni, ma sono costituiti esclusivamente da RNA, non codificare qualsiasi proteine e vengono replicati dalla polimerasi di una cellula ospite.<ref>{{cite journal|author=Daròs JA|author2=Elena SF|author3=Flores R|title=Viroids: an Ariadne's thread into the RNA labyrinth|journal=EMBO Rep|volume=7|issue=6|pages=593–8|date=2006|pmid=16741503|doi=10.1038/sj.embor.7400706|pmc=1479586}}</ref> |
|||
=== Nella trascrizione inversa === |
|||
I [[Retroviridae|virus a trascrizione inversa]] sono in grado di replicare i loro genomi attraverso una [[trascrizione inversa]] di copie di DNA dal loro RNA; queste copie di DNA vengono trascritte a nuovo RNA. I [[retrotrasposoni]] si diffondono anche copiando DNA e RNA da uno all'altro<ref>{{cite journal|author=Kalendar R|author2=Vicient CM|author3=Peleg O|author4=Anamthawat-Jonsson K|author5=Bolshoy A|author6=Schulman AH|title=Large retrotransposon derivatives: abundant, conserved but nonautonomous retroelements of barley and related genomes|journal=Genetics|volume=166|issue=3|pages = 1437–50|date=2004|pmid=15082561|doi = 10.1534/genetics.166.3.1437|pmc=1470764}}</ref> e la [[telomerasi]] contiene un RNA che viene utilizzato come modello per realizzare le estremità dei [[cromosoma|cromosomi]] eucariotici.<ref>{{cite journal|author=Podlevsky JD|author2=Bley CJ|author3=Omana RV|author4=Qi X|author5=Chen JJ|title=The telomerase database|journal=Nucleic Acids Res|volume=36|issue=Database issue|pages=D339–43|date=2008|pmid=18073191|doi=10.1093/nar/gkm700|pmc=2238860}}</ref> |
|||
=== RNA a doppio filamento === |
|||
L'RNA a doppio filamento (dsRNA) è l'RNA con due filamenti complementari, simile al DNA che può essere trovato in tutte le cellule. Il dsRNA costituisce il materiale genetico di alcuni virus (virus RNA a doppio filamento). L'RNA a doppio filamento, come l'RNA virale o il [[Short interfering RNA|siRNA]] possono innescare l'[[RNA interference]]interferenza RNA]] negli [[eucarioti]], così come risposta dell'[[interferone]] nei [[vertebrati]].<ref>{{Cite journal | last1 = Blevins | first1 = T. | last2 = Rajeswaran | first2 = R. | last3 = Shivaprasad | first3 = PV. | last4 = Beknazariants | first4 = D. | last5 = Si-Ammour | first5 = A. | last6 = Park | first6 = HS. | last7 = Vazquez | first7 = F. | last8 = Robertson | first8 = D. | last9 = Meins | first9 = F. | title = Four plant Dicers mediate viral small RNA biogenesis and DNA virus induced silencing | journal = Nucleic Acids Res | volume = 34 | issue = 21 | pages = 6233–46 | date= 2006 | doi = 10.1093/nar/gkl886 | PMID = 17090584 | pmc=1669714}} |
|||
</ref><ref>{{cite journal|author=Jana S|author2=Chakraborty C|author3=Nandi S|author4=Deb JK|title=RNA interference: potential therapeutic targets|journal=Appl. Microbiol. Biotechnol.|volume=65|issue=6|pages=649–57|date=2004|pmid=15372214|doi=10.1007/s00253-004-1732-1}}</ref><ref>{{cite journal|author=Schultz U|author2=Kaspers B|author3=Staeheli P|title=The interferon system of non-mammalian vertebrates|journal=Dev. Comp. Immunol.|volume=28|issue=5|pages=499–508|date=2004|pmid=15062646|doi=10.1016/j.dci.2003.09.009}}</ref><ref>{{Cite journal |
|||
| last1 = Whitehead | first1 = K. A. |
|||
| last2 = Dahlman | first2 = J. E. |
|||
| last3 = Langer | first3 = R. S. |
|||
| last4 = Anderson | first4 = D. G. |
|||
| title = Silencing or Stimulation? SiRNA Delivery and the Immune System |
|||
| doi = 10.1146/annurev-chembioeng-061010-114133 |
|||
| journal = Annual Review of Chemical and Biomolecular Engineering |
|||
| volume = 2 |
|||
| pages = 77–96 |
|||
| year = 2011 |
|||
| pmid = 22432611 |
|||
| pmc = |
|||
}}</ref> |
|||
== Anomalie dell'RNA == |
|||
Durante il corso degli anni i ricercatori si sono accorti che gran parte dell'RNA sintetizzato dalle polimerasi veniva scartato e solo una piccola parte veniva inviata sotto forma di mRNA per la [[sintesi proteica]]. Infatti per una proteina media di circa 400 amminoacidi (quindi 1200 nucleotidi) venivano sintetizzati anche più del doppio dei nucleotidi realmente necessari. |
Durante il corso degli anni i ricercatori si sono accorti che gran parte dell'RNA sintetizzato dalle polimerasi veniva scartato e solo una piccola parte veniva inviata sotto forma di mRNA per la [[sintesi proteica]]. Infatti per una proteina media di circa 400 amminoacidi (quindi 1200 nucleotidi) venivano sintetizzati anche più del doppio dei nucleotidi realmente necessari. |
||
Ciò è dovuto al fatto che nel [[DNA]] esistono delle sequenze non più codificanti, che servivano alla cellula quando non era ancora specializzata. Queste sequenze vengono comunque trascritte dalla polimerasi e vengono dette [[introni]], quelle che invece vengono copiate e codificanti, esoni. Questo fatto implica che prima della traduzione, esse andranno tagliate; in un processo denominato [[Splicing]] |
Ciò è dovuto al fatto che nel [[DNA]] esistono delle sequenze non più codificanti, che servivano alla cellula quando non era ancora specializzata. Queste sequenze vengono comunque trascritte dalla polimerasi e vengono dette [[introni]], quelle che invece vengono copiate e codificanti, esoni. Questo fatto implica che prima della traduzione, esse andranno tagliate; in un processo denominato [[Splicing]] |
||
== Scoperte fondamentali in biologia dell'RNA == |
|||
=== L'RNA transfer (tRNA) === |
|||
[[File:R Holley.jpg|thumb|210px|Robert W. Holley, left, poses with his research team.]] |
|||
{{Vedi anche|tRNA}} |
|||
L'RNA transfer è costituito da un filamento di RNA di circa 80 [[base azotata|basi azotate]] ripiegato a formare tre lobi, che ricordano la caratteristica forma di un trifoglio. Un lobo riconosce tramite un [[anticodone]] la tripletta sull'RNA messaggero corrispondente all'[[amminoacido]] trasportato dal transfer; un altro riconosce l'[[enzima]] che attacca l'amminoacido al transfer; un terzo è il sito di riconoscimento del ribosoma. Trasferisce ai ribosomi i vari amminoacidi, che uniti formano fra loro un [[legame peptidico]] per poter formare le proteine. |
|||
=== L'RNA messaggero (mRNA) === |
|||
{{Vedi anche|mRNA}} |
|||
L'RNA si occupa di trasportare le informazioni codificate nel DNA al [[citoplasma]]. L'mRNA si presenta sotto forma di filamento sul quale sono presenti triplette di nucleotidi (detti codoni). Il processo di formazione di un nuovo filamento è detto trascrizione. La sequenza base del filamento di mRNA è complementare a quella del filamento del DNA dal quale è stato copiato, e non identica. Quando l'mRNA ha trasmesso l'informazione si scompone nei nucleotidi che lo componevano. |
|||
La ricerche effettuate sull'RNA hanno portato a molte importanti scoperte biologiche e conferito numerosi [[premi Nobel]]. Gli [[acidi nucleici]] furono scoperti nel 1868 da [[Friedrich Miescher]], che egli chiamò materiale 'nucleino' in quanto li riscontrò nel [[nucleo cellulare|nucleo]].<ref>{{cite journal|author=Dahm R|title=Friedrich Miescher and the discovery of DNA|journal=Developmental Biology|date=2005|volume=278|issue=2|pages=274–88|pmid=15680349|doi = 10.1016/j.ydbio.2004.11.028}}</ref> Successivamente si è scoperto che le [[cellule procariote]], che non hanno un [[nucleo]], li contengono anch'essi. Il ruolo dell'RNA nella [[sintesi proteica]] era già sospettato nel 1939.<ref>{{cite journal|journal=Nature|author=Caspersson T|author2=Schultz J|title=Pentose nucleotides in the cytoplasm of growing tissues|date=1939|volume=143|doi=10.1038/143602c0|pages=602–3|issue=3623|bibcode=1939Natur.143..602C}}</ref> [[Severo Ochoa]] nel 1959 vinse il [[Premio Nobel per la Medicina]] (insieme a [[Arthur Kornberg]]) dopo aver scoperto un [[enzima[[ che può [[sintesi|sintetizzare]] l'RNA in laboratorio.<ref>{{cite web|author=Ochoa S|title=Enzymatic synthesis of ribonucleic acid|work=Nobel Lecture|date=1959|url=http://nobelprize.org/nobel_prizes/medicine/laureates/1959/ochoa-lecture.pdf}}</ref> Tuttavia, l'enzima scoperto da Ochoa ([[polinucleotide fosforilasi]]) fu poi dimostrato essere responsabile della degradazione dell'RNA e non la sintesi dell'RNA. Nel 1956 [[Alex Rich]] e [[David Davies]] ibridarono due filoni separati di RNA per formare il primo RNA la cui struttura potesse essere valutata con la [[cristallografia a raggi X]].<ref>{{cite journal | author=Rich A | author2=Davies, D|title=A New Two-Stranded Helical Structure: Polyadenylic Acid and Polyuridylic Acid|journal=Journal of the American Chemical Society|date=1956|volume=78|issue=14|doi=10.1021/ja01595a086|pages=3548–3549}}</ref> |
|||
==== Elaborazione dell'mRNA ==== |
|||
{{vedi anche|Maturazione dell'mRNA}} |
|||
La '''maturazione dell'mRNA''' (o '''maturazione dell'RNA''') è una serie di processi chimici che trasformano una molecola di [[pre-mRNA]] (''trascritto primario'') in [[mRNA]]. |
|||
La sequenza dei 77 nucleotidi del [[tRNA]] di [[lievito]] fu scoperto da [[Robert W. Holley]] nel 1965,<ref>{{cite journal|author=Holley RW|title=Structure of a ribonucleic acid|journal=Science|date=1965|volume=147|issue=3664|pages=1462–65|doi=10.1126/science.147.3664.1462|pmid = 14263761 |bibcode = 1965Sci...147.1462H|last2=Apgar|first2=J.|last3=Everett|first3=G. A.|last4=Madison|first4=J. T.|last5=Marquisee|first5=M.|last6=Merrill|first6=S. H.|last7=Penswick|first7=J. R.|last8=Zamir|first8=A. |name-list-format=vanc|displayauthors=1}}</ref> tale scoperta gli valse nel 1968 del Premio Nobel per la Medicina (in comune con [[Har Gobind Khorana]] e [[Marshall Nirenberg]]). Nel 1967, [[Carl Woese]] ipotizzò che l'RNA potesse essere un catalizzatore e suggerì che le prime forme di vita (molecole autoreplicanti) avrebbe potuto utilizzare l'RNA sia per trasportare le informazioni genetiche che per catalizzare le [[reazioni biochimice]]: l'[[ipotesi del mondo a RNA]].<ref>{{cite web|title=Common sequence structure properties and stable regions in RNA secondary structures |author=Siebert S |date=2006 |page=1 |url=http://deposit.ddb.de/cgi-bin/dokserv?idn=982323891&dok_var=d1&dok_ext=pdf&filename=982323891.pdf |work=Dissertation, Albert-Ludwigs-Universität, Freiburg im Breisgau |deadurl=yes |archiveurl=https://web.archive.org/20120309212648/http://deposit.ddb.de/cgi-bin/dokserv?idn=982323891&dok_var=d1&dok_ext=pdf&filename=982323891.pdf |archivedate=March 9, 2012 }}</ref><ref>{{cite journal|title=The origin of the genetic code: amino acids as cofactors in an RNA world|author=Szathmáry E|journal=Trends Genet|date=1999|volume=15|issue=6|pages=223–9|doi=10.1016/S0168-9525(99)01730-8|pmid = 10354582 }}</ref> |
|||
La maturazione dell'[[mRNA]] differisce molto tra gli [[eucarioti]] e i [[procarioti]]. L'mRNA procariotico è già maturo dopo la trascrizione e non richiede di essere controllato, tranne che in alcuni casi. Il pre-RNA eucariotico invece richiede diversi passaggi. |
|||
Durante i primi anni 1970, furono scoperti i [[retrovirus]] e la [[trascrittasi inversa]], che mostrò per la prima volta come gli enzimi possano copiare l'RNA in DNA (l'opposto del percorso abituale per la trasmissione delle informazioni genetiche). Per questo lavoro, [[David Baltimore]], [[Renato Dulbecco]] e [[Howard Temin]] ricevettero il premio Nobel nel 1975. L'anno seguente, [[Walter Fiers]] e il suo gruppo determinarono la prima sequenza nucleotidica del genoma completo di un virus a RNA, il [[batteriofago MS2]].<ref>{{cite journal|author=Fiers W|title=Complete nucleotide-sequence of bacteriophage MS2-RNA: primary and secondary structure of replicase gene|journal=Nature|date=1976|volume=260|pages=500–7|pmid=1264203|doi = 10.1038/260500a0|first12=M|issue=5551|bibcode=1976Natur.260..500F|last2=Ysebaert|first2=R.|last3=Duerinck|first3=F.|last4=Haegeman|first4=G.|last5=Iserentant|first5=D.|last6=Merregaert|first6=J.|last7=Min Jou|first7=W.|last8=Molemans|first8=F.|last9=Raeymaekers|first9=A.|last10=Van Den Berghe|first10=A.|last11=Volckaert|first11=G.|last12=Ysebaert|name-list-format=vanc|displayauthors=1}}</ref> |
|||
Le tappe della maturazione sono: |
|||
* [[Splicing]] |
|||
* [[Capping]] |
|||
* [[Poliadenilazione]] |
|||
Nel 1977, gli [[introni]] e lo ''[[splicing]]'' dell'RNA furono entrambi scoperti nei virus dei mammiferi e nei geni cellulari, con un conseguente Nobel a [[Philip Sharp]] e [[Richard Roberts]] nel 1993. Le prime molecole di RNA catalitici ([[ribozimi]]) furono individuati nei primi anni 1980, portando ad un Nobel premio a [[Thomas Cech]] e [[Sidney Altman]] nel 1989. Nel 1990, fu trovata nella ''[[petunia]]'' i geni che possono silenziare altri geni simili della pianta, un processo ora noto come [[RNA interference|interferenza dell'RNA]].<ref>{{cite journal|author=Napoli C|author2=Lemieux C|author3=Jorgensen R|title=Introduction of a chimeric chalcone synthase gene into petunia results in reversible co-suppression of homologous genes in trans|journal=Plant Cell|date=1990|volume=2|issue=4|pages=279–89|pmid=12354959|doi = 10.1105/tpc.2.4.279|pmc=159885}}</ref><ref>{{cite journal|author=Dafny-Yelin M|author2=Chung SM|author3=Frankman EL|author4=Tzfira T|title=pSAT RNA interference vectors: a modular series for multiple gene down-regulation in plants|journal=Plant Physiol.|volume=145|issue=4|pages=1272–81|date=December 2007|pmid=17766396|pmc=2151715|doi=10.1104/pp.107.106062}}</ref> |
|||
Molecole di [[snRNA]] prendono parte alla ''maturazione''. |
|||
Più o meno nello stesso periodo, RNA lunghi 22 nt, chiamati oggi [[microRNA]], furono scoperti per avere un ruolo nello [[Biologia dello sviluppo|sviluppo]] della ''[[Caenorhabditis elegans]]''.<ref>{{cite journal|author=Ruvkun G|title=Glimpses of a tiny RNA world|journal=Science|date=2001|volume=294|issue=5543|pages=797–99|doi=10.1126/science.1066315|pmid = 11679654}}</ref> Studi sull'interferenza dell'RNA fruttò un premio Nobel per [[Andrew Fire]] e [[Craig Mello]] nel 2006 e un altro Nobel fu assegnato per studi sulla trascrizione dell'RNA a [[Roger Kornberg]] nello stesso anno. La scoperta di geni RNA regolatori ha portato a tentativi di sviluppare farmaci realizzati con RNA, come i [[siRNA]], per silenziare i geni.<ref>{{cite journal|author=Fichou Y|author2=Férec C|title=The potential of oligonucleotides for therapeutic applications|journal=Trends in Biotechnology|date=2006|volume=24|issue=12|pages=563–70|doi=10.1016/j.tibtech.2006.10.003|pmid = 17045686}}</ref> |
|||
=== L'RNA ribosomiale (rRNA) === |
|||
{{Vedi anche|rRNA}} |
|||
L'RNA ribosomiale nel citoplasma, forma con le proteine particolari organuli, i [[ribosoma|ribosomi]], sui quali avviene la [[sintesi proteica]]. |
|||
==Note== |
|||
Il meccanismo utilizzato per la conversione dell'informazione da sequenze di nucleotidi in sequenze di amminoacidi è conosciuto come [[codice genetico]]; il processo di trasferimento di informazione dagli mRNA alle proteine costituisce la cosiddetta "traduzione" dell'informazione genetica. |
|||
{{references|auto}} |
|||
Il codice genetico consiste nell'esistenza di una corrispondenza tra unità di messaggio, costituite a sequenze di tre nucleotidi (triplette o codoni) contenute negli mRNA, e uno dei venti amminoacidi che costituiscono le proteine. |
|||
== Bibliografia == |
== Bibliografia == |
||
Versione delle 15:58, 13 ott 2015

L'acido ribonucleico (RNA) è una molecola polimerica implicata in vari ruoli biologici di codifica, decodifica, regolazione e l'espressione dei geni. L'RNA e il DNA sono acidi nucleici, e, insieme a proteine e carboidrati, costituiscono le tre principali macromolecole essenziali per tutte le forme di vita conosciute. Come il DNA, l'RNA è assemblato come una catena di nucleotidi, ma a differenza del DNA è più frequente in natura come un singolo filamento ripiegata su se stesso, piuttosto che un doppio filamento accoppiato. Gli oOrganismi cellulari utilizzano l'RNA messaggero (mRNA) per trasmettere le informazioni genetiche (mediante le lettere G, U, A, e C per indicare la basi azotate guanina, uracile, adenina e citosina) che dirige la sintesi di proteine specifiche. Molti virus codificano le loro informazioni genetiche utilizzando un genoma a RNA.
Alcune molecole di RNA svolgono un ruolo attivo all'interno delle cellule al fine di catalizzare le reazioni biologiche, di controllo dell'espressione genica o per percepire e comunicare le risposte a segnali cellulari. Uno di questi processi attivi è la sintesi proteica, una funzione universale per cui molecole di mRNA dirigono l'assemblaggio delle proteine nei ribosomi. Questo processo utilizza le molecole di RNA di trasferimento (tRNA) per fornire gli amminoacidi al ribosoma, dove l'RNA ribosomiale (rRNA) collega insieme gli amminoacidi per formare le proteine.
Confronto con il DNA

La struttura chimica dell'RNA è molto simile a quella del DNA, ma differisce in tre aspetti principali:
- Diversamente dal DNA che è a doppio filamento, l'RNA è una molecola a singolo filamento[1] in molti dei suoi ruoli biologici e ha una catena molto più breve di nucleotidi.[2] Tuttavia, l'RNA può, per l'appaiamento delle basi complementari, forma doppie eliche intraelicoidali, come nel caso del tRNA.
- Mentre DNA contiene desossiribosio, l'RNA contiene ribosio[3] (nel desossiribosio non vi è un gruppo ossidrile attaccato all'anello pentoso in posizione 2'). Questi gruppi ossidrilici rendono l'RNA meno stabile del DNA perché è più incline all'idrolisi.
- La base complementare al DNA dell'adenina è la timina, mentre nell'RNA è l'uracile, che è una forma metilata di timina.[4]
Come il DNA, l'RNA è biologicamente più attivo, tra l'mRNA, il tRNA, l'rRNA, l'snRNA e altri RNA non codificanti, contengono sequenze auto-complementari che permettono a parti dell'RNA di ripiegarsi[5] e collegarsi andando a formando doppie eliche. L'analisi di questi RNA ha rivelato che essi sono altamente strutturati. A differenza del DNA, le loro strutture non sono costituite da lunghe doppie eliche, ma piuttosto da raccolte di eliche riunite in strutture simili a proteine. In questo modo, l'RNA può realizzare la catalisi chimica, come gli enzimi.[6] Ad esempio, la determinazione della struttura del ribosoma, un enzima che catalizza la formazione di un legame peptidico, ha rivelato che il suo sito attivo è composto interamente di RNA.[7]
Struttura
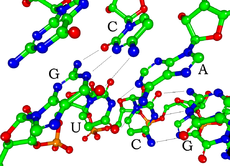
Ogni nucleotide dell'RNA contiene uno zucchero ribosio, con il carbonio numerato da 1' a 5'. Una base è collegato alla posizione 1', in generale, l'adenina (A), la citosina (C), la guanina (G) o l'uracile (U). L'adenina e la guanina sono purine, la citosina e l'uracile sono pirimidine. Un gruppo fosfato è attaccato alla posizione 3' di un ribosio e alla posizione 5' del successivo. I gruppi fosfato hanno una carica negativa a un pH fisiologico, rendendo l'RNA una molecola carica (polianione). Le basi formano legami idrogeno tra la citosina e la guanina, tra l'adenina e l'uracile e fra la guanina e l'uracile.[8] Tuttavia, altre interazioni sono possibili, come ad esempio un gruppo di basi adenina legate tra loro,[9] o un tetragiro GNRA che possiede una coppia di base di guanina-adenina.[8]

Una caratteristica strutturale importante dell'RNA che lo distingue dal DNA è la presenza di un gruppo idrossile in posizione 2' dello zucchero ribosio. La presenza di questo gruppo funzionale provoca l'adozione di un elica a forma di A rispetto alla forma B più comunemente osservata nel DNA.[10] Questo si traduce in un solco maggiore molto profondo e stretto e un solco minore superficiale e largo.[11] Una seconda conseguenza della presenza del gruppo 2'-idrossile è che nelle regioni conformazionalmente flessibili di una molecola di RNA (cioè non coinvolte nella formazione di una doppia elica), può attaccare chimicamente il legame fosfodiestere adiacente e fendere la dorsale.[12]
L'RNA è trascritto con solo quattro basi (adenina, citosina, guanina e uracile),[13] ma queste basi e zuccheri possono essere modificati in numerosi modi, come gli RNA maturi. La pseudouridina (Ψ), in cui il legame tra uracile e ribosio passa da un legame C-N ad un legame C-C, e la ribotimidina (T) si trovano in vari luoghi (i più importanti nel ciclo TΨC di tRNA ).[14] Un altro notevole base modificata è ipoxantina, una base adenina deaminato cui nucleosidici è chiamato inosina (I). Inosina gioca un ruolo chiave nella ipotesi oscillazione del codice genetico.[15]
Vi sono più di 100 altri nucleosidi modificati presenti in natura.[16] La più grande diversità strutturale di modifiche può essere trovato nel tRNA,[17] mentre la pseudouridina e i nucleosidi spesso presenti nell'rRNA sono i più comuni.[18] I ruoli specifici di molte di queste modifiche dell'RNA non sono pienamente comprese. Tuttavia, è da notare che, nell'RNA ribosomiale molte delle modificazioni post-trascrizionali si verificano nelle regioni altamente funzionali, come il centro della peptidil-transferasi e l'interfaccia subunità, che implica che sono importanti per la funzione normale.[19]
La forma funzionale di molecole a singolo filamento di RNA, come le proteine, richiede spesso una struttura terziaria specifica. L'impalcatura di questa struttura è fornita da elementi strutturali secondari che sono legami idrogeno all'interno della molecola. Questo porta a diversi "domini" riconoscibili della struttura secondaria.[20] Dal momento che l'RNA è carico, sono necessari ioni metallici, come Mg2+ per stabilizzare molte strutture secondarie e terziarie.[21]
Sintesi
La sintesi dell'RNA è solitamente catalizzata da un enzima, l'RNA-polimerasi, utilizzando il DNA come stampo, un processo noto come trascrizione. La trascrizione inizia con il legame dell'enzima ad una sequenza promotrice nel DNA (di solito "a monte" di un gene). Il DNA a doppia elica viene svolto dall'attività elicasi dell'enzima. L'enzima procede poi lungo il filamento stampo nella direzione da 3' a 5', sintetizzando una molecola di RNA complementare nell'allungamento che si verifica da 5' a 3'. La sequenza di DNA determina anche il luogo dove si verifica la cessazione della sintesi di RNA.[22]
La prima trascrizione dell'RNA è spesso modificata da enzimi dopo la trascrizione. Ad esempio, un poliadenilazione e una rivestimento in 5' vengono aggiunti a un pre-mRNA eucariote e gli introni sono rimossi dal spliceosoma.
Vi sono anche una serie di RNA polimerasi RNA-dipendente che utilizzano l'RNA come stampo per la sintesi di un nuovo filamento di RNA. Per esempio, un numero di virus a RNA (come il poliovirus) utilizzano questo tipo di enzima per replicare il loro materiale genetico.[23] Inoltre, l'RNA polimerasi RNA-dipendente è parte del percorso di interferenza dell'RNA in molti organismi.[24]
Tipi di RNA
Vi sono tre tipi di RNA comuni a tutti gli organismi cellulari:
- mRNA (RNA messaggero) che contiene l'informazione per la sintesi delle proteine;
- rRNA (RNA ribosomiale), che entra nella struttura dei ribosomi;
- tRNA (RNA transfer) necessario per la traduzione nei ribosomi.
Negli eucarioti abbiamo anche:
- hnRNA (RNA eterogeneo nucleare) tipo di molecole di cui fa parte il pre-mRNA;
- snRNA (piccolo RNA nucleare) necessario per la maturazione dell'HnRna.
La sintesi dell'RNA è molto simile a quella del DNA. La RNA polimerasi non richiede però un innesco. La trascrizione può iniziare solo presso una sequenza detta promotore e termina in presenza di altre sequenze particolari. È stata avanzata l'ipotesi che l'RNA abbia assunto un ruolo chiave negli organismi primitivi prima del DNA (RNA world o mondo a RNA). A favore di tale ipotesi c'è la capacità catalitica di alcune molecole di RNA (ribozimi). Sull'mRNA viene trascritta l'informazione genetica che poi verrà utilizzata per svariati usi.
Panoramica
L'RNA messaggero (mRNA) è l'RNA che trasporta le informazioni dal DNA al ribosoma, i siti della sintesi proteica (traduzione) nella cellula. La sequenza codificante dell'mRNA determina la sequenza aminoacidica della proteina che viene prodotta.[25] Tuttavia, molti RNA non possiedono codice per le proteine (circa il 97% della produzione trascrizionale non codifica proteine negli eucarioti[26][27][28][29]).
Questi cosiddetti RNA non codificanti ("ncRNA") possono essere codificati dai propri geni (geni RNA), ma possono anche derivare da introni di mRNA.[30] Gli esempi più importanti di RNA non codificanti sono l'RNA di trasferimento (tRNA) e l'RNA ribosomiale (rRNA), entrambi i quali sono coinvolti nel processo di traduzione.[31] Vi sono anche RNA non codificanti coinvolti nella regolazione genica, nel processo dell'RNA e altri ruoli. Alcuni RNA sono in grado di catalizzare le reazioni chimiche, come il taglio e legatura altre molecole di RNA[32] e la catalisi di formazione del legame peptidico nel ribosoma;[7] questi sono conosciuti come ribozimi.
Nella traduzione
L'RNA messaggero (mRNA) trasporta le informazioni di una sequenza proteica ai ribosomi, i luoghi della cellula deputati alla sintesi proteica. L'informazione è codificata in in modo che ad ogni tre nucleotidi (codone) corrispondano un amminoacido. Nelle cellule eucariotiche, una volta che il precursore mRNA (pre-mRNA) è stata trascritto dal DNA, si trasforma per diventare mRNA. In questa fase vengon rimosse le sezioni non codificanti del pre-mRNA. L'mRNA viene poi spostato dal nucleo al citoplasma, dove arriva ai ribosomi e quindi viene tradotto nella corrispondente proteina grazie all'aiuto del tRNA. Nelle cellule procariote, che non hanno nucleo e compartimenti nel citoplasma, l'mRNA può legarsi ai ribosomi mentre viene trascritto dal DNA. Dopo un certo periodo di tempo il messaggio degrada nelle sue componenti nucleotidi con l'assistenza della ribonucleasi.[25]
L'RNA di trasferimento (tRNA) è una piccola catena di RNA composta da circa 80 nucleotidi che trasferisce uno specifico aminoacido ad una catena polipeptidica crescente nel sito ribosomiale della sintesi proteica durante la traduzione. Esso possiede siti per l'attacco di aminoacidi e una regione anticodone per il riconoscimento del codone che si lega ad una sequenza specifica sulla catena dell'RNA messaggero attraverso legami idrogeno.[30]
L'RNA ribosomiale (rRNA) è il componente catalitico dei ribosomi. Negli eucarioti, i ribosomi contengono quattro diverse molecole di rRNA: 18S, 5.8S, 28S e 5S rRNA; tre di esse sono sintetizzate nel nucleolo. Nel citoplasma, RNA ribosomiale e le proteine si combinano per formare un nucleoproteina chiamato ribosoma. Il ribosoma lega l'mRNA ed effettua la sintesi proteica. Diversi ribosomi possono essere collegati ad un singolo mRNA in qualsiasi momento.[25] Quasi tutti gli RNA che si trovano in una tipica cellula eucariotica sono rRNA.
L'RNA transfer-messaggero (tmRNA) si trova in molti batteri e plastidi. Si occupa di marcare le proteine codificate dall'mRNA che mancano di codoni di stop per la degradazione e impedisce il blocco del ribosoma.[32]
RNA regolatori
Diversi tipi di RNA sono in grado di sottoregolare l'espressione genica per essere complementari ad una parte di un mRNA o il gene ddl DNA.[33][34] I microRNA (miRNA; 21-22 nt). Si trovano negli eucarioti e agiscono tramite l'RNA interference (RNAi), dove un complesso effettore di miRNA e enzimi in grado di scindere l'mRNA complementare, blocca la traduzione dell'mRNA vengano tradotti o accelerara la sua degradazione.[35][36]
Mentre Short interfering RNA (siRNA, 20-25 nt) vengono spesso prodotti in seguito alla rottura di RNA virale, vi sono anche fonti endogene di siRNA.[37][38] I siRNA agiscono attraverso l'interferenza dell'RNA in modo simile ai miRNA. Alcuni miRNA e siRNA possono causare la metilazione dei geni target, diminuendo o aumentando in tal modo la trascrizione di questi geni.[39][40][41] Gli animali possiedono i piwi-interacting RNA (piRNA; 29-30 nt) che sono attivi nella linea germinale delle cellule e sono ritenuti essere una difesa contro i trasposoni e svolgere un ruolo nella gametogenesi.[42][43]
Molti procarioti hanno l'RNA CRISPR, un sistema di regolamentazione simile all'interferenza dell'RNA.[44] Gli RNA antisenso sono diffusi, molti regolano un gene, ma alcuni sono attivatori della trascrizione.[45] Un modo in cui l'RNA antisenso può agire è legandosi ad un mRNA, formando un RNA a doppio filamento che viene degradato enzimaticamente.[46] Vi sono molti lunghi RNA non codificanti che regolano i geni negli eucarioti,[47] uno di questi è l'Xist, che si trova nel cromosoma X nei mammiferi di sesso femminile e serve per inattivarlo.[48]
Nell'elaborazione
Molti RNA sono coinvolti nel modificare altri RNA. Gli introni sono "montati" in pre-mRNA grazie agli spliceosomi che contengono diversi piccoli RNA nucleari (snRNA),[4] o gli introni possono essere ribozimi che vengono collegati da loro stessi.[49] L'RNA può anche essere alterato da avere i suoi nucleotidi modificati con altri nucleotidi di A, C, G e U. Negli eucarioti, le modifiche dei nucleotidi di RNA sono in genere diretti da piccoli RNA nucleolari (snoRNA; 60-300 nt),[30] riscontrati nel nucleolo e nei corpi di Cajal. Gli enzimi quindi eseguono la modifica del nucleotide. rRNA e tRNA sono ampiamente modificati, ma anche gli snRNAs e l'mRNA possono anche essere bersaglio di modifiche di base.[50][51] L'RNA può anche essere demetilato.[52][53]
Genoma RNA
Come il DNA, l'RNA può trasportare le informazioni genetiche. I virus a RNA possiedono genomi composti da RNA che codificano un numero di proteine. Il genoma virale viene replicato da alcune di queste proteine, mentre altre proteine proteggono il genoma quando la particella virale si sposta in una nuova cellula ospite. I viroidi sono un altro gruppo di agenti patogeni, ma sono costituiti esclusivamente da RNA, non codificare qualsiasi proteine e vengono replicati dalla polimerasi di una cellula ospite.[54]
Nella trascrizione inversa
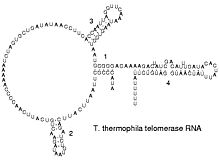
I virus a trascrizione inversa sono in grado di replicare i loro genomi attraverso una trascrizione inversa di copie di DNA dal loro RNA; queste copie di DNA vengono trascritte a nuovo RNA. I retrotrasposoni si diffondono anche copiando DNA e RNA da uno all'altro[55] e la telomerasi contiene un RNA che viene utilizzato come modello per realizzare le estremità dei cromosomi eucariotici.[56]
RNA a doppio filamento
L'RNA a doppio filamento (dsRNA) è l'RNA con due filamenti complementari, simile al DNA che può essere trovato in tutte le cellule. Il dsRNA costituisce il materiale genetico di alcuni virus (virus RNA a doppio filamento). L'RNA a doppio filamento, come l'RNA virale o il siRNA possono innescare l'RNA interferenceinterferenza RNA]] negli eucarioti, così come risposta dell'interferone nei vertebrati.[57][58][59][60]
Anomalie dell'RNA
Durante il corso degli anni i ricercatori si sono accorti che gran parte dell'RNA sintetizzato dalle polimerasi veniva scartato e solo una piccola parte veniva inviata sotto forma di mRNA per la sintesi proteica. Infatti per una proteina media di circa 400 amminoacidi (quindi 1200 nucleotidi) venivano sintetizzati anche più del doppio dei nucleotidi realmente necessari.
Ciò è dovuto al fatto che nel DNA esistono delle sequenze non più codificanti, che servivano alla cellula quando non era ancora specializzata. Queste sequenze vengono comunque trascritte dalla polimerasi e vengono dette introni, quelle che invece vengono copiate e codificanti, esoni. Questo fatto implica che prima della traduzione, esse andranno tagliate; in un processo denominato Splicing
Scoperte fondamentali in biologia dell'RNA

La ricerche effettuate sull'RNA hanno portato a molte importanti scoperte biologiche e conferito numerosi premi Nobel. Gli acidi nucleici furono scoperti nel 1868 da Friedrich Miescher, che egli chiamò materiale 'nucleino' in quanto li riscontrò nel nucleo.[61] Successivamente si è scoperto che le cellule procariote, che non hanno un nucleo, li contengono anch'essi. Il ruolo dell'RNA nella sintesi proteica era già sospettato nel 1939.[62] Severo Ochoa nel 1959 vinse il Premio Nobel per la Medicina (insieme a Arthur Kornberg) dopo aver scoperto un [[enzima[[ che può sintetizzare l'RNA in laboratorio.[63] Tuttavia, l'enzima scoperto da Ochoa (polinucleotide fosforilasi) fu poi dimostrato essere responsabile della degradazione dell'RNA e non la sintesi dell'RNA. Nel 1956 Alex Rich e David Davies ibridarono due filoni separati di RNA per formare il primo RNA la cui struttura potesse essere valutata con la cristallografia a raggi X.[64]
La sequenza dei 77 nucleotidi del tRNA di lievito fu scoperto da Robert W. Holley nel 1965,[65] tale scoperta gli valse nel 1968 del Premio Nobel per la Medicina (in comune con Har Gobind Khorana e Marshall Nirenberg). Nel 1967, Carl Woese ipotizzò che l'RNA potesse essere un catalizzatore e suggerì che le prime forme di vita (molecole autoreplicanti) avrebbe potuto utilizzare l'RNA sia per trasportare le informazioni genetiche che per catalizzare le reazioni biochimice: l'ipotesi del mondo a RNA.[66][67]
Durante i primi anni 1970, furono scoperti i retrovirus e la trascrittasi inversa, che mostrò per la prima volta come gli enzimi possano copiare l'RNA in DNA (l'opposto del percorso abituale per la trasmissione delle informazioni genetiche). Per questo lavoro, David Baltimore, Renato Dulbecco e Howard Temin ricevettero il premio Nobel nel 1975. L'anno seguente, Walter Fiers e il suo gruppo determinarono la prima sequenza nucleotidica del genoma completo di un virus a RNA, il batteriofago MS2.[68]
Nel 1977, gli introni e lo splicing dell'RNA furono entrambi scoperti nei virus dei mammiferi e nei geni cellulari, con un conseguente Nobel a Philip Sharp e Richard Roberts nel 1993. Le prime molecole di RNA catalitici (ribozimi) furono individuati nei primi anni 1980, portando ad un Nobel premio a Thomas Cech e Sidney Altman nel 1989. Nel 1990, fu trovata nella petunia i geni che possono silenziare altri geni simili della pianta, un processo ora noto come interferenza dell'RNA.[69][70]
Più o meno nello stesso periodo, RNA lunghi 22 nt, chiamati oggi microRNA, furono scoperti per avere un ruolo nello sviluppo della Caenorhabditis elegans.[71] Studi sull'interferenza dell'RNA fruttò un premio Nobel per Andrew Fire e Craig Mello nel 2006 e un altro Nobel fu assegnato per studi sulla trascrizione dell'RNA a Roger Kornberg nello stesso anno. La scoperta di geni RNA regolatori ha portato a tentativi di sviluppare farmaci realizzati con RNA, come i siRNA, per silenziare i geni.[72]
Note
- ^ RNA: The Versatile Molecule, su learn.genetics.utah.edu, University of Utah, 2015.
- ^ Nucleotides and Nucleic Acids (PDF), su chem.ucla.edu, University of California, Los Angeles.
- ^ R.N. Shukla, Analysis of Chromosomes, ISBN 9789384568177.
- ^ a b Errore nelle note: Errore nell'uso del marcatore
<ref>: non è stato indicato alcun testo per il marcatoreBiochemistry - ^ I. Tinoco, How RNA folds, in J. Mol. Biol., vol. 293, n. 2, 1999, pp. 271–281, DOI:10.1006/jmbi.1999.3001.
- ^ Higgs PG, RNA secondary structure: physical and computational aspects, in Quarterly Reviews of Biophysics, vol. 33, n. 3, 2000, pp. 199–253, DOI:10.1017/S0033583500003620.
- ^ a b Nissen P, The structural basis of ribosome activity in peptide bond synthesis, in Science, vol. 289, n. 5481, 2000, pp. 920–30, DOI:10.1126/science.289.5481.920.
- ^ a b Lee JC, Diversity of base-pair conformations and their occurrence in rRNA structure and RNA structural motifs, in J. Mol. Biol., vol. 344, n. 5, 2004, pp. 1225–49, DOI:10.1016/j.jmb.2004.09.072.
- ^ Barciszewski J, RNA biochemistry and biotechnology, Springer, 1999, pp. 73–87, ISBN 0-7923-5862-7.
- ^ Salazar M, The DNA strand in DNAoRNA hybrid duplexes is neither B-form nor A-form in solution, in Biochemistry, vol. 32, n. 16, 1992, pp. 4207–15, DOI:10.1021/bi00067a007.
- ^ Hermann T, RNA bulges as architectural and recognition motifs, in Structure, vol. 8, n. 3, 2000, pp. R47–R54, DOI:10.1016/S0969-2126(00)00110-6.
- ^ Mikkola S, The mechanism of the metal ion promoted cleavage of RNA phosphodiester bonds involves a general acid catalysis by the metal aquo ion on the departure of the leaving group, in Perkin transactions 2, n. 8, 1999, pp. 1619–26, DOI:10.1039/a903691a.
- ^ Jankowski JAZ, Clinical gene analysis and manipulation: Tools, techniques and troubleshooting, Cambridge University Press, 1996, p. 14, ISBN 0-521-47896-0.
- ^ Yu Q, Identification of critical elements in the tRNA acceptor stem and TΨC loop necessary for human immunodeficiency virus type 1 infectivity, in J Virol, vol. 75, n. 10, 2001, pp. 4902–6, DOI:10.1128/JVI.75.10.4902-4906.2001.
- ^ Elliott MS, Inosine biosynthesis in transfer RNA by an enzymatic insertion of hypoxanthine, in J. Biol. Chem., vol. 259, n. 4, 1983, pp. 2407–10.
- ^ WA Cantara, The RNA Modification Database, RNAMDB: 2011 update, in Nucleic Acids Research, vol. 39, Database issue, January 2011, pp. D195–201, DOI:10.1093/nar/gkq1028.
- ^ Söll D, TRNA: Structure, biosynthesis, and function, ASM Press, 1995, p. 165, ISBN 1-55581-073-X.
- ^ Kiss T, Small nucleolar RNA-guided post-transcriptional modification of cellular RNAs, in The EMBO Journal, vol. 20, n. 14, 2001, pp. 3617–22, DOI:10.1093/emboj/20.14.3617.
- ^ King TH, Ribosome structure and activity are altered in cells lacking snoRNPs that form pseudouridines in the peptidyl transferase center, in Molecular Cell, vol. 11, n. 2, 2002, pp. 425–35, DOI:10.1016/S1097-2765(03)00040-6.
- ^ Mathews DH, Incorporating chemical modification constraints into a dynamic programming algorithm for prediction of RNA secondary structure, in Proc. Natl. Acad. Sci. USA, vol. 101, n. 19, 2004, pp. 7287–92, DOI:10.1073/pnas.0401799101.
- ^ Tan ZJ, Salt dependence of nucleic acid hairpin stability, in Biophys. J., vol. 95, n. 2, 2008, pp. 738–52, DOI:10.1529/biophysj.108.131524.
- ^ Nudler E, Transcription termination and anti-termination in E. coli, in Genes to Cells, vol. 7, n. 8, 2002, pp. 755–68, DOI:10.1046/j.1365-2443.2002.00563.x.
- ^ Jeffrey L Hansen, Structure of the RNA-dependent RNA polymerase of poliovirus, in Structure, vol. 5, n. 8, 1997, pp. 1109–22, DOI:10.1016/S0969-2126(97)00261-X.
- ^ Ahlquist P, RNA-Dependent RNA Polymerases, Viruses, and RNA Silencing, in Science, vol. 296, n. 5571, 2002, pp. 1270–73, DOI:10.1126/science.1069132.
- ^ a b c Cooper GC, The Cell: A Molecular Approach, 3rd, Sinauer, 2004, pp. 261–76, 297, 339–44, ISBN 0-87893-214-3.
- ^ Mattick JS, The evolution of controlled multitasked gene networks: the role of introns and other noncoding RNAs in the development of complex organisms, in Mol. Biol. Evol., vol. 18, n. 9, 1º September 2001, pp. 1611–30, DOI:10.1093/oxfordjournals.molbev.a003951.
- ^ Noncoding RNAs: the architects of eukaryotic complexity, in EMBO Reports, vol. 2, n. 11, 2001, pp. 986–91, DOI:10.1093/embo-reports/kve230.
- ^ Mattick JS, Challenging the dogma: the hidden layer of non-protein-coding RNAs in complex organisms (PDF), in BioEssays : News and Reviews in Molecular, Cellular and Developmental Biology, vol. 25, n. 10, October 2003, pp. 930–9, DOI:10.1002/bies.10332.
- ^ Mattick JS, The hidden genetic program of complex organisms, in Scientific American, vol. 291, n. 4, October 2004, pp. 60–7, DOI:10.1038/scientificamerican1004-60.
- ^ a b c Wirta W, Mining the transcriptome – methods and applications, Stockholm, School of Biotechnology, Royal Institute of Technology, 2006, ISBN 91-7178-436-5.
- ^ Rossi JJ, Ribozyme diagnostics comes of age, in Chemistry & Biology, vol. 11, n. 7, 2004, pp. 894–95, DOI:10.1016/j.chembiol.2004.07.002.
- ^ Gueneau de Novoa P, Williams KP, The tmRNA website: reductive evolution of tmRNA in plastids and other endosymbionts, in Nucleic Acids Res, vol. 32, Database issue, 2004, pp. D104–8, DOI:10.1093/nar/gkh102.
- ^ RW Carthew, Origins and Mechanisms of miRNAs and siRNAs., in Cell, DOI:10.1016/j.cell.2009.01.035. URL consultato il 20 febbraio 2009.
- ^ Kung-Hao Liang, A gene expression restriction network mediated by sense and antisense Alu sequences located on protein-coding messenger RNAs., in BMC Genomics, DOI:10.1186/1471-2164-14-325. URL consultato l'11 maggio 2013.
- ^ Wu L, Let me count the ways: mechanisms of gene regulation by miRNAs and siRNAs, in Mol. Cell, vol. 29, n. 1, January 2008, pp. 1–7, DOI:10.1016/j.molcel.2007.12.010.
- ^ Matzke MA, Planting the seeds of a new paradigm, in PLoS Biology, vol. 2, n. 5, 2004, pp. e133, DOI:10.1371/journal.pbio.0020133.
- ^ Vazquez F, Endogenous trans-acting siRNAs regulate the accumulation of Arabidopsis mRNAs, in Molecular Cell, vol. 16, n. 1, 2004, pp. 69–79, DOI:10.1016/j.molcel.2004.09.028.
- ^ Watanabe T, Endogenous siRNAs from naturally formed dsRNAs regulate transcripts in mouse oocytes, in Nature, vol. 453, n. 7194, May 2008, pp. 539–43, DOI:10.1038/nature06908.
- ^ Sontheimer EJ, Silence from within: endogenous siRNAs and miRNAs, in Cell, vol. 122, n. 1, July 2005, pp. 9–12, DOI:10.1016/j.cell.2005.06.030.
- ^ Doran G, RNAi – Is one suffix sufficient?, in Journal of RNAi and Gene Silencing, vol. 3, n. 1, 2007, pp. 217–19.
- ^ Pushparaj PN, RNAi and RNAa — The Yin and Yang of RNAome, in Bioinformation, vol. 2, n. 6, 2008, pp. 235–7, DOI:10.6026/97320630002235.
- ^ Horwich MD, The Drosophila RNA methyltransferase, DmHen1, modifies germline piRNAs and single-stranded siRNAs in RISC, in Current Biology, vol. 17, n. 14, 2007, pp. 1265–72, DOI:10.1016/j.cub.2007.06.030.
- ^ Girard A, A germline-specific class of small RNAs binds mammalian Piwi proteins, in Nature, vol. 442, n. 7099, 2006, pp. 199–202, DOI:10.1038/nature04917.
- ^ Horvath P, CRISPR/Cas, the Immune System of Bacteria and Archaea, in Science, vol. 327, n. 5962, 2010, pp. 167–70, DOI:10.1126/science.1179555.
- ^ Wagner EG, Antisense RNAs in bacteria and their genetic elements, in Adv Genet, vol. 46, 2002, pp. 361–98, DOI:10.1016/S0065-2660(02)46013-0.
- ^ Gilbert SF, Developmental Biology, 7th, Sinauer, 2003, pp. 101–3, ISBN 0-87893-258-5.
- ^ Amaral PP, Noncoding RNA in development, in Mammalian genome : official journal of the International Mammalian Genome Society, vol. 19, 7–8, October 2008, pp. 454–92, DOI:10.1007/s00335-008-9136-7.
- ^ Heard E, Human XIST yeast artificial chromosome transgenes show partial X inactivation center function in mouse embryonic stem cells, in Proc. Natl. Acad. Sci. USA, vol. 96, n. 12, 1999, pp. 6841–46, DOI:10.1073/pnas.96.12.6841.
- ^ Steitz TA, A general two-metal-ion mechanism for catalytic RNA, in Proc. Natl. Acad. Sci. U.S.A., vol. 90, n. 14, 1993, pp. 6498–502, DOI:10.1073/pnas.90.14.6498.
- ^ Xie J, Sno/scaRNAbase: a curated database for small nucleolar RNAs and cajal body-specific RNAs, in Nucleic Acids Res, vol. 35, Database issue, 2007, pp. D183–7, DOI:10.1093/nar/gkl873.
- ^ Omer AD, RNA-modifying machines in archaea, in Molecular Microbiology, vol. 48, n. 3, 2003, pp. 617–29, DOI:10.1046/j.1365-2958.2003.03483.x.
- ^ Cavaillé J, Targeted ribose methylation of RNA in vivo directed by tailored antisense RNA guides, in Nature, vol. 383, n. 6602, 1996, pp. 732–5, DOI:10.1038/383732a0.
- ^ Kiss-László Z, Site-specific ribose methylation of preribosomal RNA: a novel function for small nucleolar RNAs, in Cell, vol. 85, n. 7, 1996, pp. 1077–88, DOI:10.1016/S0092-8674(00)81308-2.
- ^ Daròs JA, Viroids: an Ariadne's thread into the RNA labyrinth, in EMBO Rep, vol. 7, n. 6, 2006, pp. 593–8, DOI:10.1038/sj.embor.7400706.
- ^ Kalendar R, Large retrotransposon derivatives: abundant, conserved but nonautonomous retroelements of barley and related genomes, in Genetics, vol. 166, n. 3, 2004, pp. 1437–50, DOI:10.1534/genetics.166.3.1437.
- ^ Podlevsky JD, The telomerase database, in Nucleic Acids Res, vol. 36, Database issue, 2008, pp. D339–43, DOI:10.1093/nar/gkm700.
- ^ Four plant Dicers mediate viral small RNA biogenesis and DNA virus induced silencing, in Nucleic Acids Res, vol. 34, n. 21, 2006, pp. 6233–46, DOI:10.1093/nar/gkl886.
- ^ Jana S, RNA interference: potential therapeutic targets, in Appl. Microbiol. Biotechnol., vol. 65, n. 6, 2004, pp. 649–57, DOI:10.1007/s00253-004-1732-1.
- ^ Schultz U, The interferon system of non-mammalian vertebrates, in Dev. Comp. Immunol., vol. 28, n. 5, 2004, pp. 499–508, DOI:10.1016/j.dci.2003.09.009.
- ^ Silencing or Stimulation? SiRNA Delivery and the Immune System, in Annual Review of Chemical and Biomolecular Engineering, vol. 2, 2011, pp. 77–96, DOI:10.1146/annurev-chembioeng-061010-114133.
- ^ Dahm R, Friedrich Miescher and the discovery of DNA, in Developmental Biology, vol. 278, n. 2, 2005, pp. 274–88, DOI:10.1016/j.ydbio.2004.11.028.
- ^ Caspersson T, Pentose nucleotides in the cytoplasm of growing tissues, in Nature, vol. 143, n. 3623, 1939, pp. 602–3, DOI:10.1038/143602c0.
- ^ Ochoa S, Enzymatic synthesis of ribonucleic acid (PDF), in Nobel Lecture, 1959.
- ^ Rich A, A New Two-Stranded Helical Structure: Polyadenylic Acid and Polyuridylic Acid, in Journal of the American Chemical Society, vol. 78, n. 14, 1956, pp. 3548–3549, DOI:10.1021/ja01595a086.
- ^ Holley RW, Structure of a ribonucleic acid, in Science, vol. 147, n. 3664, 1965, pp. 1462–65, DOI:10.1126/science.147.3664.1462.
- ^ Siebert S, Common sequence structure properties and stable regions in RNA secondary structures (PDF), in Dissertation, Albert-Ludwigs-Universität, Freiburg im Breisgau, 2006 (archiviato dall'url originale il March 9, 2012).
- ^ Szathmáry E, The origin of the genetic code: amino acids as cofactors in an RNA world, in Trends Genet, vol. 15, n. 6, 1999, pp. 223–9, DOI:10.1016/S0168-9525(99)01730-8.
- ^ Fiers W, Complete nucleotide-sequence of bacteriophage MS2-RNA: primary and secondary structure of replicase gene, in Nature, vol. 260, n. 5551, 1976, pp. 500–7, DOI:10.1038/260500a0.
- ^ Napoli C, Introduction of a chimeric chalcone synthase gene into petunia results in reversible co-suppression of homologous genes in trans, in Plant Cell, vol. 2, n. 4, 1990, pp. 279–89, DOI:10.1105/tpc.2.4.279.
- ^ Dafny-Yelin M, pSAT RNA interference vectors: a modular series for multiple gene down-regulation in plants, in Plant Physiol., vol. 145, n. 4, December 2007, pp. 1272–81, DOI:10.1104/pp.107.106062.
- ^ Ruvkun G, Glimpses of a tiny RNA world, in Science, vol. 294, n. 5543, 2001, pp. 797–99, DOI:10.1126/science.1066315.
- ^ Fichou Y, The potential of oligonucleotides for therapeutic applications, in Trends in Biotechnology, vol. 24, n. 12, 2006, pp. 563–70, DOI:10.1016/j.tibtech.2006.10.003.
Bibliografia
- David L. Nelson, Michael M. Cox, I Principi di Biochimica di Lehninger, 3ª ed., Bologna, Zanichelli, febbraio 2002, ISBN 88-08-09035-3.
Voci correlate
Altri progetti
 Wikimedia Commons contiene immagini o altri file su RNA
Wikimedia Commons contiene immagini o altri file su RNA
Collegamenti esterni
- (EN) IUPAC Gold Book, "ribonucleic acids (RNA)"
- L'RNA ribosomico, sul portale RAI Scuola
- Template:Thesaurus BNCF
| Controllo di autorità | Thesaurus BNCF 25310 · LCCN (EN) sh85113850 · GND (DE) 4076759-0 · BNE (ES) XX532187 (data) · BNF (FR) cb12175223g (data) · J9U (EN, HE) 987007536325805171 |
|---|

