SARS-CoV-2: differenze tra le versioni
ARD significa Distress respiratorio acuto |
→Struttura: frase riscritta meglio |
||
| Riga 71: | Riga 71: | ||
S1/S2 e S2': siti di taglio; PF: peptide di fusione, TM: dominio transmembranico, N- e C-: N-terminale e C-terminale]] |
S1/S2 e S2': siti di taglio; PF: peptide di fusione, TM: dominio transmembranico, N- e C-: N-terminale e C-terminale]] |
||
Una variante della proteina S originale di Wuhan, nota come D614G già individuata nei primi mesi del 2020 ha iniziato a prendere piede in Europa per poi diffondersi successivamente in [[Nord America]], [[Oceania]] e [[Asia]] per diventare progressivamente a partire da marzo la più diffusa al mondo<ref>{{Cita pubblicazione|nome=Bette|cognome=Korber|nome2=Will M.|cognome2=Fischer|nome3=Sandrasegaram|cognome3=Gnanakaran|data=20 agosto 2020|titolo=Tracking Changes in SARS-CoV-2 Spike: Evidence that D614G Increases Infectivity of the COVID-19 Virus|rivista=Cell|volume=182|numero=4|pp=812–827.e19|lingua=en|accesso=24 novembre 2020|doi=10.1016/j.cell.2020.06.043|url=https://www.cell.com/cell/abstract/S0092-8674(20)30820-5}}</ref>. Tale mutazione derivata da una [[Mutazione genetica|mutazione missenso]] nel gene codificante la proteina S, risultante nel cambio di un amminoacido [[aspartato ]] a una [[glicina]] nella posizine 614<ref>{{Cita pubblicazione|nome=Sandra|cognome=Isabel|nome2=Lucía|cognome2=Graña-Miraglia|nome3=Jahir M.|cognome3=Gutierrez|data=20 agosto 2020|titolo=Evolutionary and structural analyses of SARS-CoV-2 D614G spike protein mutation now documented worldwide|rivista=Scientific Reports|volume=10|numero=1|pp=14031|lingua=en|accesso=24 novembre 2020|doi=10.1038/s41598-020-70827-z|url=https://www.nature.com/articles/s41598-020-70827-z}}</ref>. |
Una variante della proteina S originale di Wuhan, nota come D614G già individuata nei primi mesi del 2020 ha iniziato a prendere piede in Europa per poi diffondersi successivamente in [[Nord America]], [[Oceania]] e [[Asia]] per diventare progressivamente a partire da marzo la più diffusa al mondo<ref>{{Cita pubblicazione|nome=Bette|cognome=Korber|nome2=Will M.|cognome2=Fischer|nome3=Sandrasegaram|cognome3=Gnanakaran|data=20 agosto 2020|titolo=Tracking Changes in SARS-CoV-2 Spike: Evidence that D614G Increases Infectivity of the COVID-19 Virus|rivista=Cell|volume=182|numero=4|pp=812–827.e19|lingua=en|accesso=24 novembre 2020|doi=10.1016/j.cell.2020.06.043|url=https://www.cell.com/cell/abstract/S0092-8674(20)30820-5}}</ref>. Tale mutazione derivata da una [[Mutazione genetica|mutazione missenso]] nel gene codificante la proteina S, risultante nel cambio di un amminoacido [[aspartato ]] a una [[glicina]] nella posizine 614<ref>{{Cita pubblicazione|nome=Sandra|cognome=Isabel|nome2=Lucía|cognome2=Graña-Miraglia|nome3=Jahir M.|cognome3=Gutierrez|data=20 agosto 2020|titolo=Evolutionary and structural analyses of SARS-CoV-2 D614G spike protein mutation now documented worldwide|rivista=Scientific Reports|volume=10|numero=1|pp=14031|lingua=en|accesso=24 novembre 2020|doi=10.1038/s41598-020-70827-z|url=https://www.nature.com/articles/s41598-020-70827-z}}</ref>. Nonostante la variante D614G presenti una morfologia e capacità neutralizzante simile alla variante di Wuhan, la prevalsa sul ceppo iniziale è stata spiegata da una aumentata velocità di trasmissione pur mantenendo una simile carica virale e grado di malattia causata, come mostrato in esperimenti animali <ref>{{Cita pubblicazione|nome=Yixuan J.|cognome=Hou|nome2=Shiho|cognome2=Chiba|nome3=Peter|cognome3=Halfmann|data=12 novembre 2020|titolo=SARS-CoV-2 D614G variant exhibits efficient replication ex vivo and transmission in vivo|rivista=Science|lingua=en|accesso=24 novembre 2020|doi=10.1126/science.abe8499|url=https://science.sciencemag.org/content/early/2020/11/11/science.abe8499}}</ref>.La variante D614G oltre al cambio nella posizione 614 è accompagnata nella maggiorparte dei casi da altre 3 mutazioni ed è stata considerata dall'agosto 2020 la forma mondiale dominante<ref>{{Cita pubblicazione|nome=Bette|cognome=Korber|nome2=Will M.|cognome2=Fischer|nome3=Sandrasegaram|cognome3=Gnanakaran|data=20 agosto 2020|titolo=Tracking Changes in SARS-CoV-2 Spike: Evidence that D614G Increases Infectivity of the COVID-19 Virus|rivista=Cell|volume=182|numero=4|pp=812–827.e19|lingua=en|accesso=24 novembre 2020|doi=10.1016/j.cell.2020.06.043|url=https://www.cell.com/cell/abstract/S0092-8674(20)30820-5}}</ref>. |
||
=== Persistenza del virus === |
=== Persistenza del virus === |
||
Versione delle 18:05, 8 gen 2021
| SARS-CoV-2 | |
|---|---|
 | |
| Classificazione scientifica | |
| Dominio | Riboviria |
| Regno | Orthornavirae |
| Phylum | Pisuviricota |
| Classe | Pisoniviricetes |
| Ordine | Nidovirales |
| Sottordine | Cornidovirineae |
| Famiglia | Coronaviridae |
| Sottofamiglia | Orthocoronavirinae |
| Genere | Betacoronavirus |
| Sottogenere | Sarbecovirus |
| Specie | SARS-related coronavirus |
| Sinonimi | |
|
coronavirus 2 da sindrome respiratoria acuta grave | |
| Nomi comuni | |
|
2019-nCoV | |
Il coronavirus 2 da sindrome respiratoria acuta grave, abbreviato in SARS-CoV-2 (acronimo dall'inglese severe acute respiratory syndrome coronavirus 2),[1][2] in precedenza nominato nuovo coronavirus del 2019 (2019-nCoV[3]), è un ceppo virale della specie SARS-related coronavirus, facente parte del genere Betacoronavirus (famiglia Coronaviridae), sottogenere Sarbecovirus, scoperto intorno alla fine del 2019; si tratta del settimo coronavirus riconosciuto in grado di infettare esseri umani.[2]
Il nome ufficiale dato dall'Organizzazione mondiale della sanità alla sindrome causata dal virus è COVID-19 (abbreviazione dell'inglese COronaVIrus Disease-2019).[4]
Il virus è stato sequenziato genomicamente dopo un test di acido nucleico effettuato su un campione prelevato da un paziente colpito da una polmonite, di cui non si conosceva la causa, all'inizio della pandemia del 2019-2020 a Wuhan.[5][6][7]
Al 12 marzo 2020 non sono ancora ben chiare molte sue caratteristiche e, sebbene sia stata accertata la sua capacità di trasmettersi da una persona a un'altra, permangono delle incertezze sulle esatte modalità di trasmissione e sulla patogenicità.[8]
Caratteristiche biologiche
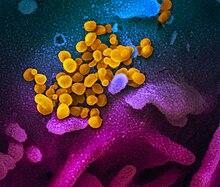
Il coronavirus SARS-CoV-2 è un ceppo virale appartenente sottogenere Sarbecovirus, della sottofamiglia dei coronavirus (Orthocoronavirinae), responsabili di patologie che vanno dal raffreddore comune a malattie più gravi come la sindrome respiratoria mediorientale (MERS) e la sindrome respiratoria acuta grave (SARS). I coronavirus sono una vasta famiglia di virus, ma solo sei (229E, NL63, OC43, HKU1, MERS-CoV, SARS-CoV) erano precedentemente noti per la capacità di infettare gli esseri umani; quindi il SARS-CoV-2 è il settimo.
La sostanziale differenza dai precedenti è il periodo di incubazione, che va da 2 a 14 giorni durante i quali non provoca alcun sintomo. Il 26 gennaio, Ma Xiaowei (ministro in carica per la Commissione Nazionale di Sanità cinese) ha dichiarato che "il nuovo coronavirus è contagioso, seppur limitatamente, anche nel suo periodo di incubazione, che dura fino a 14 giorni".[9] L'Organizzazione Mondiale della Sanità (OMS) ritiene che il tasso netto di riproduzione della trasmissione del virus da persona a persona sia tra 1,4 e 3,8. Questo valore indica il numero di altre persone a cui un paziente appena infetto può trasmettere la malattia, qualificando dunque il nuovo SARS-CoV-2 come infettivo quanto il SARS-CoV del 2002.[10] Durante una conferenza di aggiornamento del 29 gennaio 2020, l’Organizzazione Mondiale della Sanità fa sapere che il metodo di trasmissione non è solo diretto, ma anche indiretto per contatto.[11]
Il genoma del SARS-CoV-2 presenta l'89% di nucleotidi identici a quelli del SARS-like-CoVZXC21 (diffuso nei pipistrelli) e l'82% identici a quelli del SARS-CoV; tuttavia solo il 40% degli aminoacidi coincide con quelli dei coronavirus legati alla SARS.[12]
Non è ancora noto come il virus potrebbe essersi trasferito da ospiti a sangue freddo a ospiti a sangue caldo.[13] Un evento di ricombinazione omologa può aver mescolato un virus del sottogenere A (Embecovirus, virus simili a SARS Bat CoVZC45 e CoVZXC21) con la proteina legante del recettore di un Beta-CoV ancora sconosciuto.[14][15]
Genoma virale

Il 22 gennaio 2020, il Journal of Medical Virology ha pubblicato un rapporto con analisi genomica indicante che i serpenti nell'area di Wuhan sono "tra gli animali selvatici, il più probabile serbatoio" per il virus, ma sono necessarie ulteriori ricerche.[16]
Infatti, le sequenze del betacoronavirus di Wuhan mostrano somiglianze con i betacoronavirus trovati nei pipistrelli;[17] tuttavia, il virus è geneticamente distinto da altri coronavirus come quello correlato alla sindrome respiratoria acuta grave (SARS) e il coronavirus correlato alla sindrome respiratoria mediorientale da Coronavirus (MERS).[18]
Il SARS-CoV-2 è strettamente correlato al SARS-CoV-1 (identico dal 75% all'80%). Gli istituti di ricerca e per il controllo delle malattie cinesi hanno isolato cinque genomi del nuovo coronavirus, tra cui BetaCoV/Wuhan/IVDC-HB-01/2019, BetaCoV/Wuhan/IVDC-HB-04/2020, BetaCoV/Wuhan/IVDC-HB-05/2019, BetaCoV/Wuhan/WIV04/2019 e BetaCoV/Wuhan/IPBCAMS-WH-01/2019.[18][19][20] Il suo genoma è costituito da una singola elica di RNA di circa 30 kb (30 000 basi).[18]
Struttura

Ogni virione SARS-CoV-2 ha un diametro di circa 50-200 nanometri.[21]


Come altri coronavirus, SARS-CoV-2 presenta quattro proteine strutturali, note come: proteina S (spike), E (involucro), M (membrana) e N (nucleocapside); la proteina N contiene il genoma dell'RNA mentre le proteine S, E e M creano insieme il capside virale.[22] La proteina spike, che è stata analizzata a livello atomico mediante microscopia crioelettronica,[23][24] è quella che permette al virus di attaccarsi alla membrana di una cellula ospite.[22]
Gli esperimenti di modellizzazione delle proteine sulla proteina S del virus hanno suggerito che SARS-CoV-2 ha affinità con i recettori dell'enzima 2 di conversione dell'angiotensina (ACE2) delle cellule umane per usarle come "porta" di entrata nella cellula.[25]
Al 22 gennaio 2020, un gruppo cinese che lavorava con il genoma virale completo e un gruppo statunitense che utilizzava metodi di genetica inversa, hanno dimostrato indipendentemente che ACE2 era in grado di agire da recettore per SARS-CoV-2.[17][26][27][28][29][30] Gli studi hanno dimostrato che il SARS-CoV-2 ha un'affinità più elevata con ACE2 umano rispetto al SARS-CoV.[23]
La proteina S è suddivisa in due subunità S1 e S2, la subunità S1 contiene il dominio di unione al recettore (DUR) ed è responsabile dell'attacco iniziale del virus alla cellula, tramite unione S1-recettore cellulare, mentre la subunità S2 è responsabile della fusione nella membrana cellulare per l'inserimento del RNA virale all'interno. Nei virus maturi la proteina S è presente come un trimero a forma di fungo con un DUR localizzato su ognuna delle tre subunità S1 che fanno da testa poggiando su uno stelo molto flessibile[31] composto dalle subunità S2. Le proteine S appaiono distribuite aleatoriamente sulla superficie virale.[31] I DUR cambiano continuamente tra posizione alzata e sdraiata. A differenza della proteina S del SARS-CoV in cui i DUR sono preferibilmente in posizione alzata[32][33], la proteina S del SARS-CoV-2 mantiene prevalentemente i DUR in posizione sdraiata.[34][35] Questo spiega il fatto che nonostante il DUR del SARS-CoV-2 mostri maggiore affinità per l'ACE, la sua minore accessibilità fa risultare una affinità d'unione SARS-CoV-2-ACE comparabile o minore dell'affinità SARS-CoV-ACE. Ma questa caratteristica di avere un DUR più nascosto rende più facile l'evasione dall'immunosorveglianza dell'organismo ospite.[36]
Come le proteine di fusione di molti altri virus, la proteina S è attivata da proteasi cellulari presenti sulla superficie delle cellule umane. L'attivazione di tale proteina del SARS-CoV-2 è un processo che richiede il taglio proteolitico in due distinti punti, S1/S2 e S2', che genera separa le due subunità S1 e S2 che però rimangono unite non covalentemente[37][38]. Il taglio al sito S2' appena sopra il peptite idrofilo di fusione sembra sia responsabile dell'inizio del attività di fusione membranica[39][40].

Una variante della proteina S originale di Wuhan, nota come D614G già individuata nei primi mesi del 2020 ha iniziato a prendere piede in Europa per poi diffondersi successivamente in Nord America, Oceania e Asia per diventare progressivamente a partire da marzo la più diffusa al mondo[41]. Tale mutazione derivata da una mutazione missenso nel gene codificante la proteina S, risultante nel cambio di un amminoacido aspartato a una glicina nella posizine 614[42]. Nonostante la variante D614G presenti una morfologia e capacità neutralizzante simile alla variante di Wuhan, la prevalsa sul ceppo iniziale è stata spiegata da una aumentata velocità di trasmissione pur mantenendo una simile carica virale e grado di malattia causata, come mostrato in esperimenti animali [43].La variante D614G oltre al cambio nella posizione 614 è accompagnata nella maggiorparte dei casi da altre 3 mutazioni ed è stata considerata dall'agosto 2020 la forma mondiale dominante[44].
Persistenza del virus
Diverse ricerche cercano di stabilire la persistenza del SARS-CoV-2 su vari tipi di superfici e ambienti anche in relazione alle condizioni variabili di temperatura, umidità, ecc.
Aria
Ricerche indicano che il virus può rimanere infettivo negli aerosol per ore mentre sulle superfici fino a giorni.[45][46] Infatti, la COVID-19 è trasmessa dagli aerosol, in cui occorrono circa 66 minuti affinché si dimezzi il numero delle particelle di virus vitali. Il 25% mantiene ancora la virulenza dopo poco più di un'ora e il 12,5% della carica virale persiste dopo circa tre ore.[45]
Metalli e altri materiali
Sull'acciaio inossidabile, per dimezzare la carica virale sono necessarie 5 ore e 38 minuti. Sulla plastica, invece, l'emivita è di 6 ore e 49 minuti; sul cartone l'emivita è di circa tre ore e mezzo. La carica virale sul rame si dimezza più velocemente che altrove, dove la metà del virus viene inattivato entro 45 minuti.[45][47][48]
Acqua
I sistemi di sanificazione delle acque potabili dovrebbero garantire di rimuovere o inattivare il virus,[49] così come quelli delle piscine e delle vasche di idromassaggio.[49]
Al 23 marzo 2020 si ritiene che il rischio di trasmissione della COVID-19 attraverso i sistemi fognari sia basso; pur non potendolo escludere del tutto a oggi non ci sono prove che ciò si sia verificato. Nell'epidemia di SARS del 2003, è stata documentata la trasmissione associata agli aerosol delle acque reflue; per cui va monitorata l'efficienza dei sistemi di clorazione delle acque reflue.[49] Una ricerca indica come la ricerca con Wastewater-Based Epidemiology (WBE) individua efficacemente i casi positivi di SARS-CoV-2 stimati dai titoli virali delle acque reflue è di ordini di grandezza maggiore del numero di casi confermati clinicamente; questo facilità le autorità a comprendere meglio la progressione il tasso di mortalità e la progressione della malattia.[50]
Varianti genomiche e sierotipi
L'11 febbraio 2020, l'International Committee on Taxonomy of Viruses ha annunciato che, in base alle regole esistenti che calcolano le relazioni gerarchiche tra i coronavirus sulla base di cinque sequenze conservate di acidi nucleici, le differenze tra quello che era provvisoriamente chiamato 2019-nCoV e il ceppo virale dell'epidemia di SARS del 2003 erano insufficienti per ritenere le specie virali separate. Pertanto, hanno identificato 2019-nCoV come un ceppo di Coronavirus correlato alla SARS.[51]
Con un numero sufficiente di genomi sequenziati, è possibile ricostruire un albero filogenetico della storia delle mutazioni di una famiglia di virus. Al 12 gennaio 2020, cinque genomi di SARS-CoV-2 erano stati isolati a Wuhan e segnalati dal Centro cinese per il controllo e la prevenzione delle malattie;[52][53] il numero di genomi era già aumentato a 42 il 30 gennaio 2020.[54] Un'analisi filogenetica di questi campioni ha mostrato che erano altamente correlati, con al massimo sette mutazioni, ad un antenato comune, il che implicherebbe che la prima infezione umana si è verificata a novembre o dicembre 2019.[54] Al 7 maggio 2020, erano disponibili al pubblico 4690 genomi di SARS-CoV-2 campionati in sei continenti.[54]
A luglio 2020, gli scienziati hanno riferito che una variante SARS-CoV-2 più infettiva, con la variante della proteina spike G614, aveva sostituito D614 come forma dominante nella pandemia del 2019-2020.[55][56]
Nell'ottobre 2020 gli scienziati hanno descritto in un preprint che una variante, 20A.EU1, è stata osservata per la prima volta in Spagna all'inizio dell'estate ed è diventata la variante più frequente in diversi paesi europei.[57][58] Secondo il Medical Express la nuova variante che ha avuto origine in Spagna in estate è legata a un evento di super diffusione tra i lavoratori agricoli nel nord-est del paese.[59]
Nell'ottobre 2020, i ricercatori hanno scoperto un possibile gene di sovrapposizione (OLG), chiamato ORF3d, nel genoma del virus; non è noto se la proteina prodotta da ORF3d abbia qualche funzione, ma provoca una forte risposta immunitaria. ORF3d era stato identificato in un coronavirus che infetta i pangolini.[60][61]
La "variante danese" dei visoni

Una variante, trovata in Danimarca, chiamata cluster 5 (o ΔFVI-spike dallo Statens Serum Institut) ha provocato una rigorosa quarantena e una campagna di eutanasia negli allevamenti danesi di visoni.[62] Scoperta nello Jutland settentrionale, in Danimarca, si ritiene che sia stata diffusa dai visoni all'uomo negli allevamenti.
Il 4 novembre 2020 è stato annunciato che la popolazione di visoni in Danimarca sarebbe stata abbattuta per prevenire la possibile diffusione di questa mutazione e ridurre il rischio di nuove mutazioni. In sette comuni dello Jutland settentrionale sono stati introdotti un blocco e restrizioni di viaggio per impedire la diffusione della mutazione, che avrebbe potuto compromettere le risposte nazionali o internazionali alla pandemia.
L'Organizzazione Mondiale della Sanità ha affermato che il cluster 5 aveva una "sensibilità moderatamente ridotta agli anticorpi neutralizzanti",[63] e l'SSI ha avvertito che la mutazione avrebbe potuto ridurre l'effetto dei vaccini in fase di sviluppo, anche se era improbabile che li rendesse inefficaci.
In seguito al blocco e a test di massa, il 19 novembre 2020 l'SSI ha annunciato che il cluster 5 con ogni probabilità si era estinto.[64]
Ceppi
Ad agosto 2020 si ritiene che SARS-CoV-2 abbia almeno sei ceppi principali: L, S, V, G, GR e GH.[65] Il numero di sierotipi è attualmente sconosciuto.
La varietà L è stata la prima varietà scoperta a Wuhan nel dicembre 2019. Ad agosto 2020, il ceppo G e i ceppi correlati GR e GH erano i più diffusi. I ceppi L e V stanno gradualmente scomparendo.
Dall'iniziale esordio, nel novembre-dicembre 2019, dell'epidemia di SARS-CoV-2 a Wuhan in Cina, sono state individuate migliaia di varianti genomiche del virus, ciò anche per l'elevato tasso di sostituzione nucleotidica dei virus,[66] che in questo caso è pari a 8 × 10-4.[67] L'analisi genomica effettuata in Europa ed in particolare in Italia mostra, la presenza di diversi ceppi mutati del virus, con un tasso di mutazioni pari ad una ogni due settimane in media.[68]
Storia ed epidemiologia
Origine del virus
Diverse ipotesi formulate nei primi studi propendono per un'eziopatogenesi a probabile carattere zoonotico (come la SARS e la MERS). Più scienziati ritengono infatti che le malattie potrebbero aver avuto origine dal Bungarus multicinctus, un serpente altamente velenoso commerciato nel mercato umido di Wuhan, dove viene venduta carne di animali selvatici. Poiché la maggior parte del primo gruppo di umani infetti lavorava in quel mercato, si ipotizza che vi sia giunta una versione primordiale del virus, che da lì si sarebbe propagato nella provincia e nelle aree limitrofe.[16] Il 22 gennaio 2020 il Journal of Medical Virology ha pubblicato un rapporto con analisi genomica che afferma che i serpenti nell'area di Wuhan sono "il più probabile serbatoio di animali selvatici" per il virus, ma sono necessarie ulteriori ricerche. Stando ad alcune ipotesi, il virus avrebbe mutato prima dai pipistrelli e dopo i serpenti si sarebbe diffuso tramite un vettore sconosciuto.[16]
Secondo Daniel Lucey, della Georgetown University, le prime infezioni sarebbero occorse nel novembre 2019 o prima, essendoci uno studio pubblicato che mostra come tredici dei primi quarantuno pazienti riconosciuti non avevano alcunché a che fare con il mercato del pesce. Uno studio filogenetico basato su genomi pubblicati, suggerisce che la trasmissione possa essere avvenuta per il tramite di un singolo animale infetto.[69]
Le ipotesi sull'origine
Origine naturale
Il 17 marzo 2020, uno studio pubblicato su Nature afferma che la struttura genetica del virus è incompatibile con qualsiasi alterazione artificiale oggi conosciuta e che, a conferma dei precedenti studi, la sua origine è molto probabilmente animale.[70] Questa è la tesi condivisa dalla maggioranza degli scienziati.[71]
L'analisi filogenetica delle sequenze genomiche a lunghezza intera ottenute da pazienti infetti ha mostrato che SARS-CoV-2 è simile al coronavirus con sindrome respiratoria acuta grave (SARS-CoV)[72] e utilizza lo stesso recettore di ingresso cellulare, l'enzima di conversione dell'angiotensina 2 (ACE2).[73] Le speculazioni sull'origine artificiale di SARS-CoV-2 sono quindi probabilmente infondate.[74]
Origine artificiale
Una minoranza di scienziati ritiene invece che non si possa escludere la sua origine in laboratorio, con una possibile fuoruscita da esso avvenuta in modo accidentale durante la ricerca di un vaccino.[75][76] Il 14 settembre 2020 su Rai 3 in una inchiesta presentata nella trasmissione PresaDiretta, si fa riferimento all'ipotesi che il virus SARS-CoV-2 abbia avuto un'origine artificiale e sia accidentalmente uscito da un laboratorio.[77]
Giorgio Palù, virologo dell’Università di Padova, già presidente della Società europea di virologia, in un'intervista data il 9 ottobre 2020 al giornale online Ilsussidiario.net, non esclude in assoluto la possibilità che il virus SARS-CoV-2 sia stato prodotto nel laboratorio di Wuhan.[78]
Fattori epidemiologici
Verso la fine di gennaio 2020 il virus si è diffuso a Bangkok (Thailandia), Tokyo (Giappone), Seul (Corea del Sud), Cina, Taiwan, Hong Kong, Macao, in Malesia, Giappone, Stati Uniti, Vietnam, Singapore, Francia, Germania, Australia, Canada, Nepal, Cambogia, Sri Lanka, Italia, Emirati Arabi Uniti, Regno Unito, Brasile, Russia e Spagna.[79]
Alle 24:00 del 1º febbraio 2020, la Commissione Nazionale di Sanità (istituzione cinese) indicava: 14 411 casi confermati di cui 2 011 considerati gravi, 304 decessi, 328 pazienti guariti e dimessi, 19 544 casi sospetti. 118 478 persone sono sotto osservazione medica.[80][81]

Sebbene non fossero ancora del tutto chiare le modalità di trasmissione del virus, era stato confermato che è in grado di passare da persona a persona. Un funzionario della sanità pubblica nello stato di Washington negli Stati Uniti ha osservato che i coronavirus vengono trasmessi principalmente "attraverso uno stretto contatto con un altro individuo, in particolare tossendo e starnutendo su qualcun altro che si trova entro un raggio di circa 1-2 metri da quella persona".[82] Si ritenne, infatti, che nella maggior parte dei casi la diffusione tra persone avvenisse attraverso le goccioline respiratorie emesse da un individuo infetto mediante tosse o starnuti che, successivamente, vengono inalate da un soggetto sano che si trovasse nelle vicinanze. Non era chiaro se fosse possibile infettarsi dal virus anche dopo aver toccato superfici o oggetti ove sia presente portando poi le mani verso la propria bocca o verso il naso o gli occhi.[83]
Sebbene i virus respiratori siano trasmissibili solitamente quando il soggetto malato presenta anche i sintomi, sembrerebbe che il SARS-CoV-2 possa diffondersi anche in occasione di un contatto ravvicinato con un paziente asintomatico.[83] Si stima che il numero di riproduzione di base della trasmissione del virus da persona a persona sia tra il 1,4 e il 3,8. Tale valore indica il numero di altre persone a cui un paziente appena infetto possa trasmettere la malattia. Secondo quanto riferito, il nuovo coronavirus è stato finora in grado di trasmettersi in catena fino a un massimo di quattro persone.[84]
Il 22 gennaio 2020, alcuni scienziati avevano pubblicato un articolo che, dopo aver esaminato "umani, pipistrelli, galline, ricci, pangolini e due specie di serpenti", concludeva che il SARS-CoV-2 "sembra essere un virus ricombinante fra il coronavirus del pipistrello e un coronavirus di origine sconosciuta" e "tra gli animali selvatici il serpente è il serbatoio più probabile per il SARS-CoV-2 da cui poi viene trasmesso agli umani".[85][86] Ulteriori studi hanno inoltre suggerito che il SARS-CoV-2 sia originato a seguito della "combinazione di virus da pipistrelli e serpenti".[85][86][87] Tuttavia, parte della comunità scientifica ha contestato tali conclusioni sostenendo che il pipistrello doveva essere il serbatoio naturale, mentre l'ospite intermedio, un uccello o un mammifero e non gli stessi serpenti.[87][88]
Il 25 gennaio 2020 non era ancora stato confermato quale potesse essere il serbatoio naturale del SARS-CoV-2 nella fauna selvatica e l'ospite intermedio che lo ha trasmesso agli esseri umani. È stato invece confermato che il virus riesce a entrare nella cellula umana attraverso il recettore ACE2, come il virus SARS-CoV.[89]
Il 20 gennaio 2020 la trasmissione da persona a persona è stata confermata a Guangdong, in Cina, da Zhong Nanshan, capo del gruppo della commissione sanitaria che indagava sulla pandemia, rivelando che avviene attraverso le mucose di occhi, naso, bocca o contatto.
In Italia, incrociando i dati epidemiologici e molecolari, secondo uno studio italiano,[90][91] la diffusione potrebbe essere partita intorno al 25 gennaio: infatti, studiando la banca dati GISAID, si scopre che i tre genomi del virus prelevati dai malati di Codogno erano strettamente correlati a quello isolato primariamente da un paziente ammalatosi di COVID-19 in Baviera, tra il 24 e il 27 gennaio 2020.
Patologie

L'infezione da SARS-CoV-2 nell'uomo comporta una malattia chiamata COVID-19. I pazienti contagiati dal virus accusano solitamente sintomi simili all'influenza, come febbre (in oltre il 90% dei casi), tosse[92] secca (oltre l'80% dei casi), stanchezza, respiro corto (circa 20% dei casi) e difficoltà di respiro (circa 15% dei casi)[93] che sono stati descritti come "simil-influenzali".[94] Dall'analisi dei dati su 155 pazienti italiani deceduti al 6 marzo[95], condotta dall'Istituto Superiore di Sanità (ISS), sono meno comuni i sintomi gastrointestinali e no, come la diarrea, la congiuntivite e le eruzioni cutanee (da cui ha correlazione con la malattia di Kawasaki), o anche l'emottisi, cioè l'emissione di sangue dalle vie respiratorie, ad esempio con un colpo di tosse. Invece, uno studio cinese evidenzia principalmente in 99 casi su 204 pazienti, cioè circa il 48,5%, la presenza di diarrea o altri sintomi gastrointestinali (vomito, dolori addominali).[96] Sintomi che si hanno in una fase avanzata dell'infezione, sono la parziale (disosmia), o totale (anosmia) perdita olfattiva, o del gusto (disgeusia) che, al momento, non è chiaro se siano transitorie e possano terminare con la guarigione, o siano permanenti.[97][98]
Il 26 gennaio, Ma Xiaowei (Commissione Nazionale di Sanità) ha dichiarato che "il nuovo coronavirus è contagioso anche nel suo periodo di incubazione, che dura fino a 14 giorni".[9]
I casi di infezione grave possono causare polmonite, insufficienza renale acuta, fino ad arrivare al decesso.[99] I pazienti presentano anche leucopenia (carenza di globuli bianchi) e linfocitopenia (carenza di linfociti).
Similmente all'influenza e al SARS-CoV, il SARS-CoV-2 infetta e distrugge gli alveoli. Al collasso della barriera cellulare separante gli alveoli dai vasi sanguigni, liquido dai vasi penetra gli alveoli bloccando il trasporto dell'ossigeno al sangue. Una reazione eccessiva del sistema immunitario può peggiorare il danno ai tessuti, che se irreversibile può risultare letale. Ma analogamente al SARS-CoV e MERS-CoV il danno non si ferma ai polmoni. Un'infezione da SARS-CoV-2 può scatenare una risposta immunitaria eccessiva e può causare una tempesta di citochine che può condurre a un'insufficienza multipla d’organo e alla morte.
In una dichiarazione rilasciata il 23 gennaio 2020, il direttore generale dell'Organizzazione mondiale della sanità, Tedros Adhanom Ghebreyesus, ha dichiarato che un quarto degli infetti presentava ulteriori malattie gravi e che molti di quelli deceduti accusavano ulteriori patologie come ipertensione, diabete o malattie cardiovascolari che alteravano il sistema immunitario.[100] Uno studio dimostra che la circonferenza del collo è un fattore di rischio per la necessità di ventilazione invasiva meccanica ed esito peggiore della malattia.[101]
Uno studio condotto sui primi quarantuno pazienti ricoverati negli ospedali di Wuhan con casi confermati ha riferito che la maggioranza dei pazienti era in buona salute prima di contrarre l'infezione e che oltre un quarto dei soggetti precedentemente sani necessitava di terapia intensiva.[102][103] Tra la maggior parte di coloro che hanno necessitato di un ricovero in ospedale, i parametri vitali erano stabili al momento del ricovero e presentavano un basso numero di globuli bianchi e bassi linfociti.[93]
Sebbene si supponga che le prime infezioni siano avvenute già qualche mese prima,[69] i primi cinquantnove casi sospetti sono stati registrati tra la fine di dicembre 2019 e l'inizio di gennaio dell'anno successivo e tra questi l'infezione è stata confermata in quarantuno pazienti. Trenta (73%) di questi erano uomini e l'età media era di quarantanove anni; quasi un terzo (32%) presentava una patologia di base pregressa, tra cui otto con diabete, sei con ipertensione e sei con malattie cardiache. Due terzi di questo primo gruppo erano stati esposti al mercato all'ingrosso dei frutti di mare di Huanan. I sintomi riportati tra loro sono stati: quaranta (98%) con febbre, trentuno (76%) con tosse e diciotto (44%) con dolori muscolari e stanchezza. Sintomi meno frequenti includevano tosse con espettorato o emottisi, mal di testa e diarrea. Circa la metà del gruppo presentava carenza di respiro e per tredici è stato necessario il ricovero in terapia intensiva. L'esame tramite tomografia computerizzata effettuato su tutte le quarantuno persone contagiate ha rivelato la presenza di polmonite. Le complicanze includevano dodici pazienti con sindrome da distress respiratorio acuto, cinque con danno cardiaco acuto e quattro con infezione secondaria.[104]
Ricerca di un vaccino
Nel gennaio 2020, diverse organizzazioni e istituzioni hanno iniziato a lavorare sulla creazione di vaccini per il SARS-CoV-2 basato sul genoma pubblicato. Il 30 gennaio il programma quadro Orizzonte 2020 dell'UE ha pubblicato un invito a manifestare interesse.
Altrove, tre progetti sui vaccini sono supportati dalla Coalition for Epidemic Preparedness Innovations (CEPI), compresi i progetti delle società biotecnologiche Moderna e Inovio Pharmaceuticals e un altro dall'Università del Queensland. Il National Institutes of Health (NIH) degli Stati Uniti sta collaborando con Moderna per creare un vaccino RNA corrispondente a una protuberanza della superficie del coronavirus e spera di iniziare la produzione entro maggio 2020. In Australia, l'Università del Queensland sta studiando il potenziale di un vaccino a pinza molecolare che modificherebbe geneticamente le proteine virali per farle imitare il coronavirus e stimolare una reazione immunitaria. Inovio Pharmaceuticals, che sviluppa vaccinazioni del DNA che non sono ancora state approvate per uso umano, ha un vaccino candidato pronto per i test preclinici e sta collaborando con un'azienda cinese al fine di accelerarne l'accettazione da parte delle autorità di regolamentazione in Cina.
In un progetto indipendente, la Public Health Agency del Canada ha concesso l'autorizzazione al Centro internazionale per i vaccini (VIDO-InterVac) dell'Università del Saskatchewan di iniziare a lavorare su un vaccino. VIDO-InterVac mira ad avviare la produzione e la sperimentazione animale nel marzo 2020 e la sperimentazione umana nel 2021.
Note
- ^ Dopo la malattia anche il coronavirus ha un nome, Sars-CoV-2, in ANSA, 12 febbraio 2020.
- ^ a b Che cosa è il coronavirus, in Agenzia Giornalistica Italia, 7 marzo 2020. URL consultato il 18 luglio 2020.
- ^ (EN) Novel Coronavirus (2019-nCoV) Situation Report - 10 (PDF), su who.int, Organizzazione mondiale della sanità, 30 gennaio 2020. URL consultato il 18 luglio 2020.
- ^ L'OMS ha chiamato COVID-19 la sindrome causata dal nuovo coronavirus, in il Post, 11 febbraio 2020. URL consultato l'11 febbraio 2020 (archiviato l'11 febbraio 2020).
- ^ New-type coronavirus causes pneumonia in Wuhan: expert, su Xinhua, 9 gennaio 2020. URL consultato il 23 gennaio 2020.
- ^ http://www.chinacdc.cn/dfdt/201912/t20191226_209404.html
- ^ https://platform.gisaid.org/epi3/start/CoV2020
- ^ (EN) Novel coronavirus disease 2019 (COVID-19) pandemic: increased transmission in the EU/EEAand the UK – sixth update (PDF), su ecdc.europa.eu, 12 marzo 2020. URL consultato il 18 luglio 2020.
- ^ a b Coronavirus contagious even incubation stage, in https://amp.scmp.com/news/china/society/article/3047701/coronavirus-contagious-even-incubation-stage-chinas-health#.
- ^ ECDC sheet 2019-nCoV, in https://www.ecdc.europa.eu/sites/default/files/documents/Risk-assessment-pneumonia-Wuhan-China-26-Jan-2020_0.pdf.
- ^ WHO conference about 2019-CoV, in https://twitter.com/ungeneva/status/1222519769275883521?s=21.
- ^ (EN) Chan JF, Kok KH, Zhu Z, Chu H, To KK, Yuan S, Yuen KY, Genomic characterization of the 2019 novel human-pathogenic coronavirus isolated from a patient with atypical pneumonia after visiting Wuhan
- ^ Massimo Zito, Sintomi e probabile origine del coronavirus di Wuhan, su reccom.org, Reccom Magazine, 23 gennaio 2020. URL consultato il 23 gennaio 2020.
- ^ (EN) Wei Ji, Wei Wang e Xiaofang Zhao, Homologous recombination within the spike glycoprotein of the newly identified coronavirus may boost cross-species transmission from snake to human, in Journal of Medical Virology, DOI:10.1002/jmv.25682. URL consultato il 23 gennaio 2020.
- ^ (EN) Guangxiang “George” Luo, Haitao Guo e Shou-Jiang Gao, Snakes could be the original source of the new coronavirus outbreak in China, su The Conversation, 22 gennaio 2020. URL consultato il 23 gennaio 2020.
- ^ a b c Haitao Guo, Guangxiang "George" Luo e Shou-Jiang Gao, Snakes could be the source of the Wuhan coronavirus outbreak, su CNN, 23 gennaio 2020. URL consultato il 23 gennaio 2020.
- ^ a b Peng Zhou e al., A pneumonia outbreak associated with a new coronavirus of probable bat origin, in Nature, n. 579, 3 febbraio 2020, pp. 270-273, DOI:10.1038/s41586-020-2012-7.
- ^ a b c (EN) Coronavirus, su who.int. URL consultato il 16 gennaio 2020 (archiviato dall'url originale il 20 gennaio 2020).
- ^ Initial genome release of novel coronavirus, su Virological, 11 gennaio 2020. URL consultato il 12 gennaio 2020 (archiviato dall'url originale il 12 gennaio 2020).
- ^ Wuhan seafood market pneumonia virus isolate Wuhan-Hu-1, complete genome, 17 gennaio 2020.
- ^ Chen N, Zhou M, Dong X, Qu J, Gong F, Han Y, Qiu Y, Wang J, Liu Y, Wei Y, Sia J, You T, Zhang X, Zhang L, Epidemiological and clinical characteristics of 99 cases of 2019 novel coronavirus pneumonia in Wuhan, China: a descriptive study, in The Lancet, vol. 395, 15 febbraio 2020, pp. 507–513, DOI:10.1016/S0140-6736(20)30211-7, PMID 32007143, 10223. URL consultato il 9 marzo 2020 (archiviato dall'url originale il 31 gennaio 2020).
- ^ a b Wu C, Liu Y, Yang Y, Zhang P, Zhong W, Wang Y, Wang Q, Xu Y, Li M, Li X, Zheng M, Chen L, Li H, Analysis of therapeutic targets for SARS-CoV-2 and discovery of potential drugs by computational methods, in Acta Pharmaceutica Sinica B, febbraio 2020, DOI:10.1016/j.apsb.2020.02.008.
- ^ a b Wrapp D, Wang N, Corbett KS, Goldsmith JA, Hsieh CL, Abiona O, Graham BS, McLellan JS, Cryo-EM structure of the 2019-nCoV spike in the prefusion conformation, in Science, vol. 367, n. 6483, febbraio 2020, pp. 1260–1263, Bibcode:2020Sci...367.1260W, DOI:10.1126/science.abb2507, PMID 32075877.
- ^ Mandelbaum RF, Scientists Create Atomic-Level Image of the New Coronavirus's Potential Achilles Heel, su Gizmodo, 19 febbraio 2020. URL consultato il 13 marzo 2020 (archiviato dall'url originale l'8 marzo 2020).
- ^ Xu X, Chen P, Wang J, Feng J, Zhou H, Li X, Zhong W, Hao P, Evolution of the novel coronavirus from the ongoing Wuhan outbreak and modeling of its spike protein for risk of human transmission, in Science China Life Sciences, vol. 63, n. 3, marzo 2020, pp. 457–460, DOI:10.1007/s11427-020-1637-5, PMID 32009228.
- ^ Letko M, Munster V, Functional assessment of cell entry and receptor usage for lineage B β-coronaviruses, including 2019-nCoV (preprint), in bioRxiv, gennaio 2020, DOI:10.1101/2020.01.22.915660.
- ^ Letko M, Marzi A, Munster V, Functional assessment of cell entry and receptor usage for SARS-CoV-2 and other lineage B betacoronaviruses, in Nature Microbiology, febbraio 2020, DOI:10.1038/s41564-020-0688-y, PMID 32094589.
- ^ ((El Sahly HM)), Genomic Characterization of the 2019 Novel Coronavirus, in The New England Journal of Medicine. URL consultato il 9 febbraio 2020.
- ^ Gralinski LE, Menachery VD, Return of the Coronavirus: 2019-nCoV, in Viruses, vol. 12, n. 2, gennaio 2020, pp. 135, DOI:10.3390/v12020135, PMID 31991541.
- ^ Lu R, Zhao X, Li J, Niu P, Yang B, Wu H, Wang W, Song H, Huang B, Zhu N, Bi Y, Ma X, Zhan F, Wang L, Hu T, Zhou H, Hu Z, Zhou W, Zhao L, Chen J, Meng Y, Wang J, Lin Y, Yuan J, Xie Z, Ma J, Liu WJ, Wang D, Xu W, Holmes EC, Gao GF, Wu G, Chen W, Shi W, Tan W, Genomic characterisation and epidemiology of 2019 novel coronavirus: implications for virus origins and receptor binding, in The Lancet, vol. 395, n. 10224, febbraio 2020, pp. 565–574, DOI:10.1016/S0140-6736(20)30251-8, PMID 32007145.
- ^ a b (EN) Beata Turoňová, Mateusz Sikora e Christoph Schürmann, In situ structural analysis of SARS-CoV-2 spike reveals flexibility mediated by three hinges, in Science, 18 agosto 2020, DOI:10.1126/science.abd5223. URL consultato il 27 agosto 2020.
- ^ (EN) Yuan Yuan, Duanfang Cao e Yanfang Zhang, Cryo-EM structures of MERS-CoV and SARS-CoV spike glycoproteins reveal the dynamic receptor binding domains, in Nature Communications, vol. 8, n. 1, 10 aprile 2017, pp. 15092, DOI:10.1038/ncomms15092. URL consultato il 14 settembre 2020.
- ^ Miao Gui, Wenfei Song e Haixia Zhou, Cryo-electron microscopy structures of the SARS-CoV spike glycoprotein reveal a prerequisite conformational state for receptor binding, in Cell Research, vol. 27, n. 1, 23 dicembre 2016, pp. 119–129, DOI:10.1038/cr.2016.152. URL consultato il 27 agosto 2020.
- ^ Alexandra C. Walls, Young-Jun Park e M. Alejandra Tortorici, Structure, Function, and Antigenicity of the SARS-CoV-2 Spike Glycoprotein, in Cell, vol. 181, n. 2, 2020-04, pp. 281–292.e6, DOI:10.1016/j.cell.2020.02.058. URL consultato il 27 agosto 2020.
- ^ Daniel Wrapp, Nianshuang Wang e Kizzmekia S. Corbett, Cryo-EM structure of the 2019-nCoV spike in the prefusion conformation, in Science, vol. 367, n. 6483, 19 febbraio 2020, pp. 1260–1263, DOI:10.1126/science.abb2507. URL consultato il 27 agosto 2020.
- ^ Jian Shang, Yushun Wan e Chuming Luo, Cell entry mechanisms of SARS-CoV-2, in Proceedings of the National Academy of Sciences of the United States of America, vol. 117, n. 21, 26 maggio 2020, pp. 11727–11734, DOI:10.1073/pnas.2003138117. URL consultato il 27 agosto 2020.
- ^ Berend Jan Bosch, Willem Bartelink e Peter J. M. Rottier, Cathepsin L Functionally Cleaves the Severe Acute Respiratory Syndrome Coronavirus Class I Fusion Protein Upstream of Rather than Adjacent to the Fusion Peptide, in Journal of Virology, vol. 82, n. 17, 2008-9, pp. 8887–8890, DOI:10.1128/JVI.00415-08. URL consultato il 26 novembre 2020.
- ^ Sandrine Belouzard, Victor C. Chu e Gary R. Whittaker, Activation of the SARS coronavirus spike protein via sequential proteolytic cleavage at two distinct sites, in Proceedings of the National Academy of Sciences of the United States of America, vol. 106, n. 14, 7 aprile 2009, pp. 5871–5876, DOI:10.1073/pnas.0809524106. URL consultato il 26 novembre 2020.
- ^ Ikenna G. Madu, Shoshannah L. Roth e Sandrine Belouzard, Characterization of a Highly Conserved Domain within the Severe Acute Respiratory Syndrome Coronavirus Spike Protein S2 Domain with Characteristics of a Viral Fusion Peptide, in Journal of Virology, vol. 83, n. 15, 2009-8, pp. 7411–7421, DOI:10.1128/JVI.00079-09. URL consultato il 26 novembre 2020.
- ^ Alexandra C. Walls, M. Alejandra Tortorici e Berend-Jan Bosch, Cryo-electron microscopy structure of a coronavirus spike glycoprotein trimer, in Nature, vol. 531, n. 7592, 2016, pp. 114–117, DOI:10.1038/nature16988. URL consultato il 26 novembre 2020.
- ^ (EN) Bette Korber, Will M. Fischer e Sandrasegaram Gnanakaran, Tracking Changes in SARS-CoV-2 Spike: Evidence that D614G Increases Infectivity of the COVID-19 Virus, in Cell, vol. 182, n. 4, 20 agosto 2020, pp. 812–827.e19, DOI:10.1016/j.cell.2020.06.043. URL consultato il 24 novembre 2020.
- ^ (EN) Sandra Isabel, Lucía Graña-Miraglia e Jahir M. Gutierrez, Evolutionary and structural analyses of SARS-CoV-2 D614G spike protein mutation now documented worldwide, in Scientific Reports, vol. 10, n. 1, 20 agosto 2020, pp. 14031, DOI:10.1038/s41598-020-70827-z. URL consultato il 24 novembre 2020.
- ^ (EN) Yixuan J. Hou, Shiho Chiba e Peter Halfmann, SARS-CoV-2 D614G variant exhibits efficient replication ex vivo and transmission in vivo, in Science, 12 novembre 2020, DOI:10.1126/science.abe8499. URL consultato il 24 novembre 2020.
- ^ (EN) Bette Korber, Will M. Fischer e Sandrasegaram Gnanakaran, Tracking Changes in SARS-CoV-2 Spike: Evidence that D614G Increases Infectivity of the COVID-19 Virus, in Cell, vol. 182, n. 4, 20 agosto 2020, pp. 812–827.e19, DOI:10.1016/j.cell.2020.06.043. URL consultato il 24 novembre 2020.
- ^ a b c van Doremalen N, Bushmaker T, Morris DH, Holbrook MG, Gamble A, Williamson BN, Tamin A, Harcourt JL, Thornburg NJ, Gerber SI, Lloyd-Smith JO, de Wit E, Munster VJ, Aerosol and Surface Stability of SARS-CoV-2 as Compared with SARS-CoV-1, in The New England Journal of Medicine, marzo 2020, DOI:10.1056/NEJMc2004973, PMID 32182409. URL consultato il 20 marzo 2020.
- ^ (EN) John A. Lednicky, Michael Lauzardo e Z. Hugh Fan, Viable SARS-CoV-2 in the air of a hospital room with COVID-19 patients, in medRxiv, 4 agosto 2020, pp. 2020.08.03.20167395, DOI:10.1101/2020.08.03.20167395. URL consultato il 19 agosto 2020.
- ^ (EN) Luca Fiorillo, Gabriele Cervino e Marco Matarese, COVID-19 Surface Persistence: A Recent Data Summary and Its Importance for Medical and Dental Settings, in International Journal of Environmental Research and Public Health, vol. 17, n. 9, 30 aprile 2020, pp. 3132, DOI:10.3390/ijerph17093132. URL consultato il 17 maggio 2020.
- ^ a b c Municipal Water and COVID-19, su cdc.gov. URL consultato il 23 marzo 2020.
- ^ Wu F, Zhang J, Xiao A, Gu X, Lee WL, Armas F, Kauffman K, Hanage W, Matus M, Ghaeli N, Endo N, Duvallet C, Poyet M, Moniz K, Washburne AD, Erickson TB, Chai PR, Thompson J, Alm EJ, SARS-CoV-2 Titers in Wastewater Are Higher than Expected from Clinically Confirmed Cases, in mSystems, vol. 5, n. 4, luglio 2020, DOI:10.1128/mSystems.00614-20, PMID 32694130. URL consultato il 20 settembre 2020.
- ^ (EN) Coronaviridae Study Group of the International Committee on Taxonomy of Viruses, The species Severe acute respiratory syndrome-related coronavirus: classifying 2019-nCoV and naming it SARS-CoV-2, in Nature Microbiology, vol. 5, n. 4, 2020-04, pp. 536–544, DOI:10.1038/s41564-020-0695-z. URL consultato il 22 dicembre 2020.
- ^ www.epicov.org, https://www.epicov.org/epi3/start/CoV2020. URL consultato il 22 dicembre 2020.
- ^ (EN) Novel 2019 coronavirus genome, su Virological, 11 gennaio 2020. URL consultato il 22 dicembre 2020.
- ^ a b c auspice, su nextstrain.org. URL consultato il 22 dicembre 2020.
- ^ (EN) New, more infectious strain of COVID-19 now dominates global cases of virus: study, su medicalxpress.com. URL consultato il 22 dicembre 2020.
- ^ (EN) Bette Korber, Will M. Fischer e Sandrasegaram Gnanakaran, Tracking Changes in SARS-CoV-2 Spike: Evidence that D614G Increases Infectivity of the COVID-19 Virus, in Cell, vol. 182, n. 4, 2020-08, pp. 812–827.e19, DOI:10.1016/j.cell.2020.06.043. URL consultato il 22 dicembre 2020.
- ^ (EN) Sam Meredith, A new coronavirus variant is seen spreading across Europe, research says, su CNBC, 29 ottobre 2020. URL consultato il 22 dicembre 2020.
- ^ (EN) Emma B. Hodcroft, Moira Zuber e Sarah Nadeau, Emergence and spread of a SARS-CoV-2 variant through Europe in the summer of 2020, Epidemiology, 28 ottobre 2020, DOI:10.1101/2020.10.25.20219063.. URL consultato il 22 dicembre 2020.
- ^ Neil M. Expert: New covid-19 variant may have spread here via north. Irish Examiner. Oct 30 2020. Available from: https://search-proquest-com.wikipedialibrary.idm.oclc.org/newspapers/expert-new-covid-19-variant-may-have-spread-here/docview/2456035970/se-2?accountid=196403.
- ^ (EN) Peter Dockrill, Scientists Just Found a Mysteriously Hidden 'Gene Within a Gene' in SARS-CoV-2, su ScienceAlert. URL consultato il 22 dicembre 2020.
- ^ (EN) Chase W Nelson, Zachary Ardern e Tony L Goldberg, Dynamically evolving novel overlapping gene as a factor in the SARS-CoV-2 pandemic, in eLife, vol. 9, 1º ottobre 2020, pp. e59633, DOI:10.7554/eLife.59633. URL consultato il 22 dicembre 2020.
- ^ - The Washington Post, su Washington Post, 1º gennaio 1970. URL consultato il 23 dicembre 2020.
- ^ 6 countries find coronavirus at mink farms; fears mutation could hinder vaccine, in The Times of Israel, 8 novembre 2020. URL consultato il 9 novembre 2020.«Italy, the Netherlands, Spain and Sweden are the other nations to have discovered SARS-CoV-2 in minks, WHO said in a statement.»
- ^ De fleste restriktioner læmpes i Nordjylland, su sum.dk, Sundheds- og Ældreministeriet, 19 novembre 2020.
- ^ (EN) The six strains of SARS-CoV-2, su ScienceDaily. URL consultato il 22 dicembre 2020.
- ^ Drake JW, Charlesworth B, Charlesworth D, Crow JF, Rates of spontaneous mutation, in Genetics, vol. 148, n. 4, April 1998, pp. 1667–86, PMC 1460098, PMID 9560386.
- ^ (LT) auspice - Global, su Nextstrain. URL consultato il 22 dicembre 2020.
- ^ (LT) auspice - Italy, su Nextstrain. URL consultato il 22 dicembre 2020.
- ^ a b (EN) Jon Cohen, Wuhan seafood market may not be source of novel virus spreading globally, su ScienceMag American Association for the Advancement of Science. (AAAS), 26 gennaio 2020. URL consultato il 29 gennaio 2020 (archiviato dall'url originale il 27 gennaio 2020).
- ^ (EN) Kristian G. Andersen, Andrew Rambaut, W. Ian Lipkin, Edward C. Holmes & Robert F. Garry, The proximal origin of SARS-CoV-2, Nature, 17 marzo 2020.
- ^ Andersen KG, Rambaut A, Lipkin WI, Holmes EC, Garry RF, The proximal origin of SARS-CoV-2, in Nat. Med., vol. 26, n. 4, aprile 2020, pp. 450–452, DOI:10.1038/s41591-020-0820-9, PMC 7095063, PMID 32284615. URL consultato il 18 settembre 2020.
- ^ Wang H, Li X, Li T, Zhang S, Wang L, Wu X, Liu J, The genetic sequence, origin, and diagnosis of SARS-CoV-2, in Eur. J. Clin. Microbiol. Infect. Dis., vol. 39, n. 9, settembre 2020, pp. 1629–1635, DOI:10.1007/s10096-020-03899-4, PMC 7180649, PMID 32333222. URL consultato il 17 settembre 2020.
- ^ Dallavilla T, Bertelli M, Morresi A, Bushati V, Stuppia L, Beccari T, Chiurazzi P, Marceddu G, Bioinformatic analysis indicates that SARS-CoV-2 is unrelated to known artificial coronaviruses, in Eur Rev Med Pharmacol Sci, vol. 24, n. 8, aprile 2020, pp. 4558–4564, DOI:10.26355/eurrev_202004_21041, PMID 32373995. URL consultato il 18 settembre 2020.
- ^ (EN) E.Sallard, J.Halloy, D.Casane, E.Decroly, J.van Helden, Tracing the origins of SARS-COV-2 in coronavirus phylogenies, su hal.archives-ouvertes.fr, 13 agosto 2020 (in press). URL consultato il 13 agosto 2020. Contiene una presentazione delle ipotesi contrapposte.
- ^ (FR) Le coronavirus, fabriqué à partir du virus du sida ? La thèse très contestée du professeur Montagnier, su Le Monde, 17 aprile 2020. URL consultato il 13 agosto 2020.
- ^ SARS-Cov-2 Identikit di un killer, su rai.it, RAI Ufficio Stampa. URL consultato il 18 settembre 2020.
- ^ Marco Biscella, IL COVID È ARTIFICIALE?/ “È stato costruito in laboratorio a Wuhan: ecco le 3 prove”, su ilsussidiario.net, www.ilsussidiario.net. URL consultato il 9 ottobre 2020.
- ^ Realtime 2019-nCoV Map, su bnonews.com.
- ^ 2019nCoV situation - China, su nhc.gov.cn. URL consultato il 29 gennaio 2020 (archiviato dall'url originale il 29 gennaio 2020).
- ^ China National Health Center [collegamento interrotto], su en.nhc.gov.cn.
- ^ Erika Edwards, How does coronavirus spread?.
- ^ a b CDC, How 2019-nCoV Spreads, su cdc.gov. URL consultato il 1º febbraio 2020.
- ^ Tina Hesman Saey, How the new coronavirus stacks up against SARS and MERS, su sciencenews.org, 24 gennaio 2020. URL consultato il 25 gennaio 2020 (archiviato dall'url originale il 25 gennaio 2020).
- ^ a b Haitao Guo, Guangxiang "George" Luo, Shou-Jiang Gao, Snakes Could Be the Original Source of the New Coronavirus Outbreak in China, su Scientific American, 22 gennaio 2020. URL consultato il 24 gennaio 2020 (archiviato dall'url originale il 25 gennaio 2020).
- ^ a b Wei Ji, Wei Wang, Xiaofang Zhao, Junjie Zai e Xingguang Li, Homologous recombination within the spike glycoprotein of the newly identified coronavirus may boost cross‐species transmission from snake to human, in Journal of Medical Virology, 22 gennaio 2020, DOI:10.1002/jmv.25682. URL consultato il 22 gennaio 2020.
- ^ a b Ewen Callaway e David Cyranoski, Why snakes probably aren't spreading the new China virus, in Nature, 23 gennaio 2020, DOI:10.1038/d41586-020-00180-8. URL consultato il 23 gennaio 2020 (archiviato dall'url originale il 25 gennaio 2020).
- ^ Megan Multeni, No, the Wuhan Virus Is Not a 'Snake Flu', su Wired, 23 gennaio 2020. URL consultato il 24 gennaio 2020 (archiviato dall'url originale il 24 gennaio 2020).
- ^ Zheng-Li Shi, Peng Zhou, Xing-Lou Yang, Xian-Guang Wang, Ben Hu, Lei Zhang, Wei Zhang, Hao-Rui Si, Yan Zhu, Bei Li e Chao-Lin Huang, Discovery of a novel coronavirus associated with the recent pneumonia outbreak in humans and its potential bat origin, in bioRxiv, 23 gennaio 2020, pp. 2020.01.22.914952, DOI:10.1101/2020.01.22.914952.
- ^ (EN) Gianguglielmo Zehender, Alessia Lai, Annalisa Bergna, Luca Meroni, Agostino Riva, Claudia Balotta, Maciej Tarkowski, Arianna Gabrieli, Dario Bernacchia, Stefano Rusconi, Giuliano Rizzardini, Antinori e Massimo Galli, Genomic Characterisation And Phylogenetic Analysis Of Sars-Cov-2 In Italy, in Journal of medical virology, medRxIV, 20 marzo 2020, DOI:10.1101/2020.03.15.20032870.
- ^ Il virus Sars-Cov-2 era in Italia già da gennaio, su ANSA, 26 marzo 2020. URL consultato il 26 marzo 2020.
- ^ Wuhan pneumonia virus outbreak: What we know so far, su CNA. URL consultato il 21 gennaio 2020 (archiviato dall'url originale il 22 gennaio 2020).
- ^ a b Hui DS, I Azhar E, Madani TA, Ntoumi F, Kock R, Dar O, Ippolito G, Mchugh TD, Memish ZA, Drosten C, Zumla A, Petersen E, The continuing 2019-nCoV epidemic threat of novel coronaviruses to global health - The latest 2019 novel coronavirus outbreak in Wuhan, China, in International Journal of Infectious Diseases : IJID : Official Publication of the International Society for Infectious Diseases, vol. 91, gennaio 2020, pp. 264–266, DOI:10.1016/j.ijid.2020.01.009, PMID 31953166.
- ^ (EN) DOH monitors child from Wuhan, China who manifested flu-like symptoms, su Yahoo. URL consultato il 21 gennaio 2020 (archiviato dall'url originale il 22 gennaio 2020).
- ^ Comunicato Stampa n°17/2020 - Coronavirus, febbre e affanno sintomi iniziali più comuni per i deceduti positivi a Covid-19, su iss.it, 7 marzo 2020. URL consultato il 20 marzo 2020.
- ^ (EN) Pan Lei, Mu Mi, Ren Hong Gang, Yang Pengcheng, Sun Yu, Wang Rungsheng, Yan Junhong, Li Pibao, Hu Baoguang, Hu Chao, Jin Yuan, Niu Xun, Ping Rongyu, Du Yingzhen, Li Tianzhi, Liu Chengxia, Xu Guogang, Hu Qinyong e Tu Lei, Clinical characteristics of COVID-19 patients with digestive symptoms in Hubei, China: a descriptive, cross-sectional, multicenter study (PDF), su The American Journal of Gastroenterology, 18 marzo 2020. URL consultato il 20 marzo 2020.
- ^ Sintomi Coronavirus, anche perdita gusto e olfatto "La notiamo in fase avanzata", su ilsussidiario.net.
- ^ Loss of sense of smell as marker of COVID-19 infection (PDF), su entuk.org.
- ^ (EN) Q&A on coronaviruses, su www.who.int. URL consultato il 27 gennaio 2020.
- ^ WHO Director-General's statement on the advice of the IHR Emergency Committee on Novel Coronavirus, su www.who.int.
- ^ (EN) Stefano Di Bella, Roberto Cesareo e Paolo De Cristofaro, Neck circumference as reliable predictor of mechanical ventilation support in adult inpatients with COVID‐19: A multicentric prospective evaluation, in Diabetes/Metabolism Research and Reviews, 14 giugno 2020, DOI:10.1002/dmrr.3354. URL consultato il 21 luglio 2020.
- ^ Andrew Joseph, New coronavirus can cause infections with no symptoms and sicken otherwise healthy people, studies show [collegamento interrotto], in STAT, 24 gennaio 2020. URL consultato il 27 gennaio 2020.
- ^ (EN) Chaolin Huang, Yeming Wang, Xingwang Li, Lili Ren, Jianping Zhao, Yi Hu, Li Zhang, Guohui Fan, Jiuyang Xu, Xiaoying Gu e Zhenshun Cheng, Clinical features of patients infected with 2019 novel coronavirus in Wuhan, China, in The Lancet, 24 gennaio 2020, pp. S0140673620301835, DOI:10.1016/S0140-6736(20)30183-5.
- ^ Chaolin Huang, Yeming Wang, Xingwang Li, Lili Ren, Jianping Zhao, Yi Hu, Li Zhang, Guohui Fan, Jiuyang Xu, Xiaoying Gu e Zhenshun Cheng, Clinical features of patients infected with 2019 novel coronavirus in Wuhan, China, in The Lancet, vol. 0, 24 gennaio 2020, DOI:10.1016/S0140-6736(20)30183-5, ISSN 0140-6736, PMID 31986264.
Altri progetti
 Wikiquote contiene citazioni di o su SARS-CoV-2
Wikiquote contiene citazioni di o su SARS-CoV-2 Wikimedia Commons contiene immagini o altri file su SARS-CoV-2
Wikimedia Commons contiene immagini o altri file su SARS-CoV-2 Wikispecies contiene informazioni su SARS-CoV-2
Wikispecies contiene informazioni su SARS-CoV-2
Voci correlate
- Coronavirus
- SARS
- MERS
- COVID-19
- Pandemia di COVID-19 del 2019-2021
- Pandemia
- Epidemia di SARS del 2002-2004
- Epidemia
- SARS-CoV
- SARS-related coronavirus
- MERS-CoV
- Virus trasmessi da pipistrelli
Collegamenti esterni
- Corona MAP - Servizio gratuito con la mappa di diffusione del virus per provincia Italiana e del resto del mondo. Aggiornato con dati da fonti ufficiali.
- FAQ - Infezione da coronavirus 2019-nCoV, su salute.gov.it, Ministero della Salute.
- (EN) 2019-nCoV Global Cases by Johns Hopkins CSSE, su gisanddata.maps.arcgis.com.ci
- Taxonomy browser (Severe acute respiratory syndrome coronavirus 2), su NCBI.
| Controllo di autorità | Thesaurus BNCF 68706 · GND (DE) 1206288906 · BNE (ES) XX6068537 (data) · BNF (FR) cb17874459p (data) |
|---|
