Orthocoronavirinae
| Orthocoronavirinae | |
|---|---|
 | |
| Intervallo geologico | |
| Classificazione scientifica | |
| Dominio | Riboviria |
| Regno | Orthornavirae |
| Phylum | Pisuviricota |
| Classe | Pisoniviricetes |
| Ordine | Nidovirales |
| Sottordine | Cornidovirineae |
| Famiglia | Coronaviridae |
| Sottofamiglia | Orthocoronavirinae |
Orthocoronavirinae è una sottofamiglia di virus, noti anche come coronavirus, della famiglia Coronaviridae, del sottordine Cornidovirineae, dell'ordine Nidovirales.[1] In passato era classificata come genere.
Suddivisa nei generi (in passato sottogeneri) Alphacoronavirus, Betacoronavirus, Gammacoronavirus e Deltacoronavirus, questi includono genogruppi filogeneticamente compatti di RNA avvolto, a senso positivo, e con un nucleocapside di simmetria elicoidale. Alphacoronavirus e Betacoronavirus derivano dal pool genico dei pipistrelli.
La "dimensione genomica" dei coronavirus varia da circa 26 a 32 kilobasi, straordinariamente grande per un virus a RNA.[2][3] Il loro numero sta crescendo rapidamente con diversi nuovi coronavirus scoperti di recente, tra cui SARS-CoV-1 scoperto nel 2002, MERS-CoV nel 2012 e SARS-CoV-2 scoperto nel 2019 a Wuhan, in Cina.
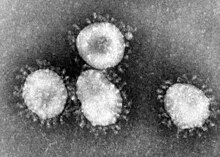
Il nome "coronavirus" deriva dal termine latino corona, a sua volta derivato dal greco κορώνη (korṓnē, "ghirlanda"), che significa "corona" o "aureola". Ciò si riferisce ai peplomeri o spikes[4] (proteine S) formate dai virioni (la forma infettiva del virus) visibile al microscopio elettronico, che presenta una serie di glicoproteine superficiali che danno un'immagine che ricorda una corona reale o una corona solare. I peplomeri sono proteine virali che popolano la superficie del virus e determinano il tropismo nell'ospite.
Si pensa che il primo caso riportato di coronavirus sia dovuto a veterinari tedeschi che nel 1912 descrissero il caso di un gatto febbricitante con un enorme ingrossamento del ventre. Ma solo negli anni 1960 un virus con struttura a forma di corona che causava comuni raffreddori venne isolato e venne associato al fenomeno, inclusi altri riscontrati in svariati animali. I ricercatori pensarono che i coronavirus fossero capaci di causare negli umani solo sintomi lievi, fino a che non ci fu l'epidemia di SARS nel 2003[5].
I coronavirus sono responsabili di diverse patologie nei mammiferi e negli uccelli, dal verificarsi di diarrea nei bovini e nei suini e a malattie respiratorie delle vie superiori nei polli. Nell'uomo, provocano infezioni delle vie respiratorie, spesso di lieve entità come il raffreddore comune, ma in rari casi potenzialmente letali come polmoniti e bronchiti.
I coronavirus sono stati responsabili delle gravi epidemie di SARS del 2002-2004, di quella della MERS del 2012-2014 e della pandemia di COVID-19 del 2019-2023.
Struttura
[modifica | modifica wikitesto]
I coronavirus sono virus a RNA positivo dal diametro di circa 80-160 nm, il che li rende tra i più grandi virus capaci di attaccare l'essere umano. Con 30 000 basi genetiche, i coronavirus hanno il più ampio genoma tra i virus a RNA, inoltre è uno dei pochi virus a RNA ad avere un meccanismo di correzione di lettura genetica che previene l'accumulo di mutazioni. Il nome del virus deriva dalla classica forma apprezzabile al microscopio elettronico a trasmissione a "corona". Questo aspetto è dato dalla presenza di spinule rappresentate dalla glicoproteina che attraversa il pericapside, raggiungendo il rivestimento proteico, detta proteina spinula o proteina S, con proprietà emoagglutinanti e di fusione. La struttura del virus è quella più o meno tipica dei virus rivestiti: presenta quindi un nucleocapside a simmetria elicoidale e un pericapside costituito da un doppio strato fosfolipidico di origine cellulare; tra questi due strati si interpone un coat proteico costituito dalla proteina M (matrice). Nel nucleocapside si ritrova il genoma costituito da un ssRNA+ (un filamento di RNA singolo a polarità positiva) da 27-30 kilo basi che codifica per 7 proteine virali ed è associato alla proteina N.
I coronavirus si attaccano alla membrana cellulare delle cellule bersaglio grazie alle loro proteine S che interagiscono con l'aminopeptidasi N della membrana. Alcuni coronavirus possono legare l'acido N-acetil neuraminico grazie all'espressione della glicoproteina E3. Non è chiaro se la penetrazione della cellula sia effettuata mediante fusione del pericapside con la membrana plasmatica o per endocitosi. All'interno del citoplasma della cellula il coronavirus rilascia il suo RNA a singolo filamento positivo che si attacca ai ribosomi, dove viene tradotto. La traduzione comporta la produzione di una RNA-polimerasi RNA-dipendente (proteina L) che trascrive un RNA a filamento negativo da cui poi è possibile ottenere nuovi RNA a filamento positivo del coronavirus, nonché le sette proteine che esso codifica. A ciascun nuovo filamento di RNA positivo si associa la proteina N, mentre le proteine del pericapside si integrano nella membrana del reticolo endoplasmatico. Un traslocatore trasferisce i nuovi nucleocapsidi nel lume del reticolo endoplasmatico; successivamente da questo gemmano vescicole che costituiscono i nuovi virioni che possono essere rilasciati per esocitosi.
I generi
[modifica | modifica wikitesto]La famiglia dei coronavirus è stata divisa in quattro generi distinti:[6][7]
- Alphacoronavirus (α-CoV)
- Betacoronavirus (β-CoV)
- Gammacoronavirus (γ-CoV)
- Deltacoronavirus (δ-CoV)
Il genere alphacoronavirus (precedentemente noto come gruppo 1 dei Coronavirus, CoV-1) comprende i sottogruppi 1a e 1b, che sono prototipati dal coronavirus umano 229E (HCoV-229E) e HCoV-NL63, nonché dalla nuova specie alphacoronavirus 1 (incluso virus della gastroenterite trasmissibile suina, TGEV), rispettivamente.
Il genere betacoronavirus (precedentemente gruppo 2 dei coronavirus, Cov-2) comprende diversi sottogruppi, con i più importanti (sottogruppi 2a e 2b) prototipati dalla specie murine coronavirus (incluso il virus dell'epatite murina, MHV) e SARS-related coronavirus. Si divide nei sottogeneri Embecovirus, Hibecovirus, Merbecovirus, Nobecovirus e Sarbecovirus.
Il genere gammacoronavirus comprende tutti i coronavirus aviari identificati fino al 2009.
Coronavirus di interesse umano
[modifica | modifica wikitesto]
I coronavirus sono stati scoperti negli anni sessanta dalle cavità nasali dei pazienti con raffreddore comune.[8] Questi virus furono successivamente chiamati Coronavirus umano 229E (HCoV-229E) e Coronavirus umano OC43 (HCoV-OC43).[9] Sono stati identificati altri due membri di questa famiglia (Coronavirus umano NL63, HCoV-NL63, nel 2004; Coronavirus umano HKU1, HCoV-HKU1, nel 2005) e sono stati coinvolti in infezioni del tratto respiratorio più gravi.
Non esistono vaccini o farmaci antivirali considerati validi dalla comunità scientifica per la prevenzione o per il trattamento delle patologie indotte.
Si ritiene che i coronavirus causino una percentuale significativa di tutti i raffreddori comuni negli adulti e nei bambini. I sintomi che si riscontrano più frequentemente sono febbre e adenoidite acuta con maggior incidenza durante l'inverno e l'inizio della primavera.[10] In molti casi i coronavirus possono causare polmonite, polmonite virale diretta o polmonite batterica secondaria; inoltre possono portare anche allo sviluppo di bronchite, bronchite virale diretta o bronchite batterica secondaria.[11]
I ceppi causa delle principali patologie che interessano l'uomo appartengono al genere Betacoronavirus.[12]
Il coronavirus umano scoperto nel 2003, SARS-CoV-1, causa una grave sindrome respiratoria acuta (SARS) e ha una patogenesi unica, perché causa infezioni del tratto respiratorio superiore e inferiore.[11]
La variante SARS dei coronavirus, apparsa inizialmente in Cina nella provincia del Guangdong nel novembre 2002 e isolata per la prima volta l'anno successivo, ha le stesse identiche caratteristiche morfologiche degli altri coronavirus, ma sembra sia una specie del tutto nuova derivata probabilmente da un serbatoio animale (non ancora noto) che ben si è adattato all'uomo. Tra i fattori che il virus della SARS utilizza per incrementare notevolmente la sua virulenza rispetto agli altri coronavirus, c'è un potente sistema di inibizione dell'interferone.
Un altro focolaio pericoloso provocato da un diverso ceppo di coronavirus ha avuto inizio nel giugno 2012 in Arabia Saudita. La malattia è stata perciò indicata col nome di sindrome respiratoria mediorientale da Coronavirus o MERS (dall'acronimo in inglese). Sono stati accertati con test di laboratorio almeno 2000 casi nel mondo, di cui oltre i 3/4 in Arabia Saudita;[13] fino al giugno 2015 c'erano già stati oltre 500 morti (su circa 1500 casi registrati fino a quella data).[14]
Il 31 dicembre 2019 è stato segnalato un nuovo ceppo di questo virus a Wuhan, in Cina,[15] identificato come un nuovo ceppo di β-CoV dal Gruppo 2B con una somiglianza genetica del 70% circa rispetto al SARS-CoV-1. Il nuovo ceppo, di conseguenza, è stato nominato SARS-CoV-2.
A gennaio 2020 sono conosciuti 7 ceppi di coronavirus in grado di infettare gli umani:
- Coronavirus umano 229E (HCoV-229E)
- Coronavirus umano OC43 (HCoV-OC43)
- Coronavirus umano NL63 (HCoV-NL63)
- Coronavirus umano HKU1 (HCoV-HFU1)[3]
- Coronavirus da sindrome respiratoria acuta grave (SARS-CoV-1)
- Coronavirus della sindrome respiratoria mediorientale (MERS-CoV), conosciuto anche come Novel Coronavirus 2012 (2012-nCoV)
- Coronavirus 2 da sindrome respiratoria acuta grave (SARS-CoV-2),[16][17] conosciuto anche come Novel Coronavirus 2019 (2019-nCoV) o Coronavirus di Wuhan.[18]
La trasmissione dei coronavirus tra umani avviene principalmente attraverso le goccioline respiratorie emesse da un individuo infetto mediante tosse o starnuti, che successivamente vengono inalate da un soggetto sano che si trovi nelle vicinanze. Sembrerebbe che sia possibile infettarsi anche dopo aver toccato superfici o oggetti ove sia presente il virus e portando successivamente le mani verso la propria bocca o verso il naso o gli occhi.[ fonte errata, non presente[19]]
Sebbene i virus respiratori siano trasmissibili solitamente quando il soggetto malato presenta anche i sintomi, il coronavirus SARS-CoV-2 può diffondersi anche in occasione di un contatto ravvicinato con un paziente infetto asintomatico.[ fonte errata, non presente[19]]
Tassonomia
[modifica | modifica wikitesto]- Orthocoronavirinae
- Alphacoronavirus
- Betacoronavirus
- Deltacoronavirus
- Gammacoronavirus
- Cegacovirus
- Igacovirus
- Avian coronavirus – specie tipo
I nuovi coronavirus
[modifica | modifica wikitesto]Con la sigla nCoV vengono genericamente indicate nuove specie o ceppi di Coronavirus che non sono mai stati precedentemente identificati nell'uomo.[20]
Note
[modifica | modifica wikitesto]- ^ Taxonomy browser (Orthocoronavirinae), su ncbi.nlm.nih.gov. URL consultato il 14 marzo 2020.
- ^ de Groot RJ, Baker SC, Baric R, Enjuanes L, Gorbalenya AE, Holmes KV, Perlman S, Poon L, Rottier PJ, Talbot PJ, Woo PC, Ziebuhr J, Family Coronaviridae, in AMQ King, E Lefkowitz, MJ Adams e EB Carstens (a cura di), Ninth Report of the International Committee on Taxonomy of Viruses, Elsevier, Oxford, 2011, pp. 806-828, ISBN 978-0-12-384684-6.
- ^ a b International Committee on Taxonomy of Viruses, ICTV Master Species List 2009 – v10 (XLS), su talk.ictvonline.org, 24 agosto 2010.
- ^ Kenneth J. Ryan, Sherris. Microbiologia medica, 7ª ed., 1º gennaio 2021.
- ^ nature.com, https://www.nature.com/articles/d41586-020-01315-7.
- ^ (EN) Taxonomy, su ICTV. URL consultato il 19 gennaio 2020.
- ^ Taxonomy browser (Orthocoronavirinae), su National Center for Biotechnology Information. URL consultato il 13 marzo 2020.
- ^ Jeffrey S. Kahn e Kenneth McIntosh, History and recent advances in coronavirus discovery, in The Pediatric Infectious Disease Journal, vol. 24, 11 Suppl, 2005-11, pp. S223–227, discussion S226, DOI:10.1097/01.inf.0000188166.17324.60. URL consultato il 14 marzo 2020.
- ^ Geller C, Varbanov M, Duval RE, Coronavirus umani: approfondimenti sulla resistenza ambientale e la loro influenza sullo sviluppo di nuove strategie antisettiche, in Viruses.
- ^ (EN) Liu P, Shi L, Zhang W, He J, Liu C, Zhao C, Kong SK, Loo JF, Gu D, Hu L, Prevalence and genetic diversity analysis of human coronaviruses among cross-border children, in Virology Journal, vol. 14, n. 1, novembre 2017, p. 230, DOI:10.1186/s12985-017-0896-0, PMC 5700739, PMID 29166910.
- ^ a b (EN) Forgie S, Marrie TJ, Healthcare-associated atypical pneumonia, in Seminars in Respiratory and Critical Care Medicine, vol. 30, n. 1, febbraio 2009, pp. 67-85, DOI:10.1055/s-0028-1119811, PMID 19199189.
- ^ (EN) Cluster of pneumonia cases caused by a novel coronavirus, Wuhan, China (PDF), su ECDC (European Centre for Disease Prevention and Control), 17 gennaio 2020. URL consultato il 19 gennaio 2020.
- ^ (EN) WHO MERS-CoV Global Summary and Assessment of Risk (PDF), su WHO [OMS], 2017. URL consultato il 19 gennaio 2020.
- ^ (EN) Epidemiological update: Middle East respiratory syndrome coronavirus (MERS-CoV), su ECDC (European Centre for Disease Prevention and Control), 2015. URL consultato il 19 gennaio 2020.
- ^ Il nuovo coronavirus, spiegato bene, su Il Post, 27 gennaio 2020. URL consultato il 27 gennaio 2020.
- ^ (EN) Laboratory testing of human suspected cases of novel coronavirus (nCoV) infection. Interim guidance, 10 January 2020 (PDF), su apps.who.int. URL consultato il 14 gennaio 2020 (archiviato il 20 gennaio 2020).
- ^ (EN) Novel Coronavirus 2019, Wuhan, China | CDC, su cdc.gov, 23 gennaio 2020. URL consultato il 23 gennaio 2020 (archiviato il 20 gennaio 2020).
- ^ Pneumonia of unknown cause – China, su who.int, World Health Organization, 5 gennaio 2020. URL consultato il 23 gennaio 2020 (archiviato il 7 gennaio 2020).
- ^ a b (EN) Transmission of Novel Coronavirus (2019-nCoV) | CDC, su cdc.gov, 31 gennaio 2020. URL consultato il 1º febbraio 2020.
- ^ Ministero della Salute, FAQ - Covid-19, domande e risposte, su salute.gov.it. URL consultato il 1º giugno 2020.
Bibliografia
[modifica | modifica wikitesto]- Murray, Patrick R., Pfaller, Michael A. e Fadda, Giovanni., Microbiologia medica, EMSI, 2008, ISBN 978-88-86669-56-6, OCLC 875233240.
- International Committee on Taxonomy of Viruses, Virus taxonomy: ninth report of the International Committee on Taxonomy of Viruses, Elsevier, 2011, ISBN 9780123846846, OCLC 767516716.
Voci correlate
[modifica | modifica wikitesto]Altri progetti
[modifica | modifica wikitesto] Wikimedia Commons contiene immagini o altri file su Orthocoronavirinae
Wikimedia Commons contiene immagini o altri file su Orthocoronavirinae Wikispecies contiene informazioni su Orthocoronavirinae
Wikispecies contiene informazioni su Orthocoronavirinae
Collegamenti esterni
[modifica | modifica wikitesto]- Taxonomy browser (Orthocoronavirinae), su NCBI.
- NEJM: Resources on the Coronavirus (Covid-19) outbreak, su nejm.org.
| Controllo di autorità | GND (DE) 4276392-7 |
|---|
