Proteasoma
I proteasomi (o proteosomi) sono dei complessi di proteasi ATP-dipendenti che degradano all'interno della cellula le proteine aberranti, attraverso reazioni di proteolisi[1]. Le proteine da degradare sono contraddistinte dal loro legame con l'ubiquitina.
I proteasomi sono presenti nelle cellule di tutti gli eucarioti ed archea[2], nonché in alcuni batteri[3]. La struttura e la funzione di questi complessi è altamente conservata, soprattutto tra le specie eucariotiche[4].
Per "la scoperta della degradazione delle proteine mediata da ubiquitina" è stato assegnato il Premio Nobel per la chimica del 2004 ad Aaron Ciechanover, Avram Hershko ed Irwin Rose.
A causa del ruolo dei proteasomi nella regolazione del ciclo cellulare e dell'apoptosi, sono oggi un bersaglio rilevante nelle terapie antitumorali[5][6].
Struttura[modifica | modifica wikitesto]

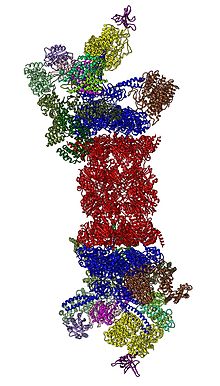
Ogni proteasoma è costituito da una subunità centrale chiamata 20S[7] che svolge l'attività proteolitica: è formata da 4 anelli costituiti da 7 subunità ciascuno. I due anelli centrali catalitici sono chiamati β, mentre i due laterali α.
Ogni subunità 20S, poi, può legare ad entrambe le estremità una subunità detta 19S, andando a formare il proteasoma 26S, oppure PA28 (formato da PA28α e PA28β) che, sostituendo 19S aumenta l'attività del complesso. Sia PA28 che 19S sono subunità regolatorie che mirano a controllare l'attività del proteasoma. La loro localizzazione è importante per degradare le proteine in modo corretto.
Ruolo nel sistema immunitario adattativo[modifica | modifica wikitesto]
Nella serie di eventi che prendono parte alla processazione dell'antigene, in ambito immunologico, il proteasoma costituisce una tappa importantissima. In particolare la stimolazione di interferone causa la trascrizione e la successiva traduzione di diverse subunità catalitiche per il complesso: LMP2 e LMP7 (entrambe codificate nel complesso maggiore di istocompatibilità) e MECL-1. Queste strutture vanno a sostituire le subunità β acquisendo, quindi una diversa specificità catalitica: aumentano i tagli a seguito di amminoacidi idrofobici e si abbassano quelli dopo residui acidi. Questo perché si vengono a generare peptidi che legano preferibilimente le molecole MHC e prediligono il trasporto attraverso TAP1, due tappe importanti nella formazione di antigeni che dovranno poi essere esposti su molecole MHC di classe I. Nell'immunoproteasoma diventa di fondamentale importanza la presenza delle subunità PA28 la cui produzione viene stimolata dall'interferone-γ.
Ubiquitina[modifica | modifica wikitesto]
L'ubiquitina è un peptide di 76 amminoacidi (3 ripetuti), altamente conservato negli eucarioti (nei procarioti non è presente). Nelle cellule funge da marcatore di proteine da degradare nella via proteasomiale, secondo i meccanismi descritti più avanti. La sua attivazione avviene grazie al suo legame con l'enzima E1 (enzima attivatore dell'ubiquitina). Successivamente viene trasferita all'enzima E2 (enzima coniugatore dell'ubiquitina), dal quale può passare alla proteina bersaglio o, nella maggior parte dei casi, ad un ulteriore enzima, detto E3 (ubiquitina ligasi), che catalizza l'attacco al polipeptide da degradare: è quindi una proteina regolatoria.
Proteolisi[modifica | modifica wikitesto]
La via proteolitica proteasomiale inizia nel citosol, dove le proteine da degradare vengono riconosciute dall'enzima ubiquitinante. L'attacco del peptide avviene a livello del gruppo amminico di un residuo laterale di lisina. A questo ne seguono molti altri, in modo da avere una proteina bersaglio che presenti numerosissime ramificazioni, rappresentate da residui di ubiquitina.
Il polipeptide viene così riconosciuto dalle subunità α del proteasoma, che tramite l'idrolisi di ATP permette l'ingresso della proteina ed un suo corretto dispiegamento all'interno della struttura. Contemporaneamente le molecole di ubiquitina vengono rimosse e liberate nel citosol, per essere riutilizzate.
Il prodotto finale della degradazione è una serie di peptidi di 6-10 amminoacidi di lunghezza, corrispondenti alla distanza dei siti proteolitici all'interno del proteasoma. Questa dunque è la seconda differenza fondamentale rispetto al lisosoma, che lide i polipeptidi in amminoacidi singoli.
Note[modifica | modifica wikitesto]
- ^ ALBERTS, B., JOHNSON, A., LEWIS, J., RAFF, M., ROBERTS, K., & WALTER, P., Molecular biology of the cell, Garland Science.
- ^ J. A. Maupin-Furlow, H. L. Wilson e S. J. Kaczowka, Proteasomes in the archaea: from structure to function, in Frontiers in Bioscience: A Journal and Virtual Library, vol. 5, 1º settembre 2000, pp. D837–865, DOI:10.2741/furlow. URL consultato l'11 settembre 2021.
- ^ Samuel H. Becker e K. Heran Darwin, Bacterial Proteasomes: Mechanistic and Functional Insights, in Microbiology and Molecular Biology Reviews : MMBR, vol. 81, n. 1, 14 dicembre 2016, DOI:10.1128/MMBR.00036-16. URL consultato l'11 settembre 2021.
- ^ Keiji Tanaka, The proteasome: Overview of structure and functions, in Proceedings of the Japan Academy. Series B, Physical and Biological Sciences, vol. 85, n. 1, 2009-1, pp. 12–36, DOI:10.2183/pjab.85.12. URL consultato l'11 settembre 2021.
- ^ Julian Adams, The proteasome: structure, function, and role in the cell, in Cancer Treatment Reviews, 29 Suppl 1, 2003-05, pp. 3–9, DOI:10.1016/s0305-7372(03)00081-1. URL consultato l'11 settembre 2021.
- ^ (EN) J. B. Almond e G. M. Cohen, The proteasome: a novel target for cancer chemotherapy, in Leukemia, vol. 16, n. 4, 2002-04, pp. 433–443, DOI:10.1038/sj.leu.2402417. URL consultato l'11 settembre 2021.
- ^ Molto spesso in biologia molecolare si usa dare il nome a molecole o a parti di esse con il coefficiente di sedimentazione espresso in Svedberg. L'esempio più famoso è senz'altro per i ribosomi. Il coefficiente di una molecola non è la somma dei coefficienti delle subunità che la costituiscono.
Bibliografia[modifica | modifica wikitesto]
- Kriegenburg F, Poulsen EG, Koch A, Krüger E, Hartmann-Petersen R. (2011)
- Redox Control of the Ubiquitin-Proteasome System: From Molecular Mechanisms to Functional Significance PMID 21314436
- Abbas, Lichtman, Pillai, Immunologia cellulare e molecolare, ELSEVIER, 2012
- Murphy, Janeway's Immunobiology, Garland Scienze, 2012
Altri progetti[modifica | modifica wikitesto]
 Wikimedia Commons contiene immagini o altri file su proteasoma
Wikimedia Commons contiene immagini o altri file su proteasoma
Collegamenti esterni[modifica | modifica wikitesto]
http://www.treccani.it/enciclopedia/proteosoma/ https://www.ncbi.nlm.nih.gov/pubmed/?term=21314436
| Controllo di autorità | GND (DE) 4315827-4 |
|---|