Clostridioides difficile: differenze tra le versioni
m →Epidemie storiche: |date ----> |data |
Nessun oggetto della modifica Etichetta: Editor wikitesto 2017 |
||
| Riga 2: | Riga 2: | ||
|nome=Clostridioides difficile |
|nome=Clostridioides difficile |
||
|statocons= |
|statocons= |
||
|immagine=Clostridium difficile |
|immagine=Clostridium difficile 01.jpg |
||
|didascalia= ''Clostridioides difficile'' |
|didascalia= ''Clostridioides difficile'' |
||
<!-- CLASSIFICAZIONE --> |
<!-- CLASSIFICAZIONE --> |
||
| Riga 46: | Riga 46: | ||
}} |
}} |
||
'''''Clostridioides difficile''''', una volta conosciuto come ''Clostridium difficile'', è un [[Bacteria|batterio]] appartenente alla [[Famiglia (tassonomia)|famiglia]] [[Clostridiaceae]]. Come altre [[specie]] affini, |
'''''Clostridioides difficile''''', una volta conosciuto come ''Clostridium difficile''<ref>{{Cita pubblicazione|nome=Paul A.|cognome=Lawson|nome2=Diane M.|cognome2=Citron|nome3=Kerin L.|cognome3=Tyrrell|data=2016-08|titolo=Reclassification of Clostridium difficile as Clostridioides difficile (Hall and O'Toole 1935) Prévot 1938|rivista=Anaerobe|volume=40|pp=95–99|accesso=2021-01-22|doi=10.1016/j.anaerobe.2016.06.008|url=https://pubmed.ncbi.nlm.nih.gov/27370902/}}</ref> o ''Peptoclostridium difficile''<ref>{{Cita web|url=https://www.ncbi.nlm.nih.gov/assembly/GCF_900014645.1/|titolo=Peptoclostridium difficile - Genome - Assembly - NCBI|sito=www.ncbi.nlm.nih.gov|accesso=2021-01-23}}</ref>, è un [[Bacteria|batterio]] sporigeno appartenente alla [[Famiglia (tassonomia)|famiglia]] [[Clostridiaceae]]. È un bastoncello, [[Gram +]], anaerobio. Come altre [[specie]] affini, quando produce specifiche tossine, può essere patogeno per l'[[homo sapiens sapiens|uomo]]. |
||
È un bastoncello [[Gram +]] anaerobio. |
|||
Ampiamente diffuso nel suolo è presente anche nell’apparato gastrointestinale di vari animali: cani, gatti, cavalli, maiali, roditori. Il ''C.difficile'' fa parte della normale [[Microbiota umano|flora saprofita dell’intestino umano]] e colonizza tra il 3% e il 15% degli adulti sani. Può essere isolato nell’ 80% delle feci dei bambini fino a 1 anno di età, colonizzazione favorita dall’immaturità della flora batterica intestinale. |
|||
I fattori di virulenza del ''C. difficile'' sono: [[enterotossina]], spore e [[ialuronidasi]]. Questo bacillo si trova normalmente nel [[microbiota umano]] per cui, se si utilizzano per lungo tempo antibiotici, questi possono distruggere anche quei batteri che tengono confinato il ''C. difficile'' il quale può prendere il sopravvento e provocare crampi addominali e malattie varie (colite pseudomembranosa). Queste malattie hanno un decorso benigno e autolimitanti tranne nel caso in cui ci sono complicanze che possono compromettere la parete intestinale con un possibile passaggio in circolo del bacillo, con conseguente [[sepsi]] e quindi morte dell'individuo. L'isolamento può avvenire tramite la [[coprocoltura]] in [[Terreno selettivo per Clostridium difficile|terreni selettivi per C. difficile]]. L'enterotossina può essere saggiata con il metodo [[ELISA]], mentre la citotossina con rivelazione di tossicità in colture cellulari. |
|||
La specie C.difficile è definita da un grande e diversificato pan-genoma con livelli estremi di plasticità evolutiva che è stata modellata per lunghi periodi di tempo dal flusso genico e dalla [[Ricombinazione genetica|ricombinazione]], spesso tra lignaggi divergenti. Sono stati isolati centinaia di diversi ceppi del ''C.difficile,'' non tutti tossigeni. La continua evoluzione del ''C.difficile'' in risposta alle attività ambientali e antropiche ha portato alla rapida comparsa e alla diffusione in tutto il mondo di lignaggi clonali virulenti. |
|||
L'[[Infezione da Clostridioides difficile|infezione da ''Closteridioides difficile'']] è, in alcuni paesi, la causa più comune di infezioni nosocomiali e causerebbe circa 15000 morti l'anno negli USA e 3700 nella UE/EEAA.<ref>{{Cita pubblicazione|nome=Evelyn|cognome=Balsells|nome2=Ting|cognome2=Shi|nome3=Callum|cognome3=Leese|titolo=Global burden of Clostridium difficile infections: a systematic review and meta-analysis|rivista=Journal of Global Health|volume=9|numero=1|accesso=2021-01-23|doi=10.7189/jogh.09.010407|url=https://www.ncbi.nlm.nih.gov/pmc/articles/PMC6304170/}}</ref><ref>{{Cita web|url=https://www.cdc.gov/hai/dpks/deadly-diarrhea/dpk-deadly-diarrhea.html|titolo=Nearly half a million Americans suffer from C. difficile infections in|autore=CDC|sito=Centers for Disease Control and Prevention|data=2018-12-31|lingua=en-us|accesso=2021-01-23}}</ref><ref>{{Cita web|url=https://www.ecdc.europa.eu/en/clostridium-difficile-infections/facts|titolo=Clostridium difficile infections - Facts and surveillance|sito=European Centre for Disease Prevention and Control|lingua=en|accesso=2021-01-23}}</ref> |
|||
== Cenni storici == |
== Cenni storici == |
||
Hall e O'Toole, in materiale fecale di neonati, nel 1935 individuarono il batterio: ''Bacillus difficilis'', chiamato così per la difficoltà incontrata nell'isolamento e per l'estrema lentezza di crescita dello stesso sul terreno colturale. Nel 1970 fu rinominato come ''Clostridium difficile''.<ref name=Hall_1935>{{Cita pubblicazione | autore = Hall, Ivan C. |autore2= O'Toole. Elizabeth| titolo = Intestinal flora in newborn infants with a description of a new pathogenic anaerobe, ''Bacillus difficilis'' |rivista = [[Archives of Pediatrics & Adolescent Medicine|American Journal of Diseases of Children]] | anno = 1935 | volume = 49 |numero = 2|pp = 390–402| url = http://archpedi.ama-assn.org/cgi/content/summary/49/2/390}}</ref><ref name="Chen-2008">{{Cita pubblicazione | cognome = Chen | nome = X. | coautori = K. Katchar; JD. Goldsmith; N. Nanthakumar; A. Cheknis; DN. Gerding; CP. Kelly | titolo = A mouse model of Clostridium difficile-associated disease. | rivista = Gastroenterology | volume = 135 | numero = 6 | pp = 1984-92 | mese=dicembre| anno = 2008 | doi = 10.1053/j.gastro.2008.09.002 | pmid = 18848941 }}</ref><ref name="Kelly-2008">{{Cita pubblicazione | cognome = Kelly | nome = CP. | coautori = JT. LaMont | titolo = Clostridium difficile--more difficult than ever. | rivista = N Engl J Med | volume = 359 | numero = 18 | pp = 1932-40 | mese=ottobre| anno = 2008 | doi = 10.1056/NEJMra0707500 | pmid = 18971494 }}</ref><ref name="Hu-2009">{{Cita pubblicazione | cognome = Hu | nome = MY. | coautori = K. Katchar; L. Kyne; S. Maroo; S. Tummala; V. Dreisbach; H. Xu; DA. Leffler; CP. Kelly | titolo = Prospective derivation and validation of a clinical prediction rule for recurrent Clostridium difficile infection. | rivista = Gastroenterology | volume = 136 | numero = 4 | pp = 1206-14 | mese=aprile| anno = 2009 | doi = 10.1053/j.gastro.2008.12.038 | pmid = 19162027 }}</ref> Mentre il primo caso di [[Colite pseudomembranosa]] era stato documentato da Finney nel 1893 in una autopsia di una donna, morta per una [[diarrea]] emorragica post-intervento chirurgico, che presentava pseudomembrane simil-[[difterite|difteriche]] nel [[colon]].<ref name="Lamont-2002">{{Cita pubblicazione | cognome = Lamont | nome = JT. | titolo = Theodore E. Woodward Award. How bacterial enterotoxins work: insights from in vivo studies. | rivista = Trans Am Clin Climatol Assoc | volume = 113 | pp = 167-80; discussion 180-1 | anno = 2002 | pmid = 12053708 }}</ref><ref name="GillespieHawkey2006">{{Cita libro|autore=Stephen H. Gillespie|autore2=Peter M. Hawkey|titolo=Principles and Practice of Clinical Bacteriology|url=http://books.google.com/books?id=Mfaf4twXsP4C&pg=PA557|data=1º luglio 2006|editore=John Wiley and Sons|pagine=557–|isbn=978-0-470-03532-0}}</ref> |
Hall e O'Toole, in materiale fecale di neonati, nel 1935 individuarono il batterio: ''Bacillus difficilis'', chiamato così per la difficoltà incontrata nell'isolamento e per l'estrema lentezza di crescita dello stesso sul terreno colturale. Nel 1970 fu rinominato come ''Clostridium difficile''.<ref name=Hall_1935>{{Cita pubblicazione | autore = Hall, Ivan C. |autore2= O'Toole. Elizabeth| titolo = Intestinal flora in newborn infants with a description of a new pathogenic anaerobe, ''Bacillus difficilis'' |rivista = [[Archives of Pediatrics & Adolescent Medicine|American Journal of Diseases of Children]] | anno = 1935 | volume = 49 |numero = 2|pp = 390–402| url = http://archpedi.ama-assn.org/cgi/content/summary/49/2/390}}</ref><ref name="Chen-2008">{{Cita pubblicazione | cognome = Chen | nome = X. | coautori = K. Katchar; JD. Goldsmith; N. Nanthakumar; A. Cheknis; DN. Gerding; CP. Kelly | titolo = A mouse model of Clostridium difficile-associated disease. | rivista = Gastroenterology | volume = 135 | numero = 6 | pp = 1984-92 | mese=dicembre| anno = 2008 | doi = 10.1053/j.gastro.2008.09.002 | pmid = 18848941 }}</ref><ref name="Kelly-2008">{{Cita pubblicazione | cognome = Kelly | nome = CP. | coautori = JT. LaMont | titolo = Clostridium difficile--more difficult than ever. | rivista = N Engl J Med | volume = 359 | numero = 18 | pp = 1932-40 | mese=ottobre| anno = 2008 | doi = 10.1056/NEJMra0707500 | pmid = 18971494 }}</ref><ref name="Hu-2009">{{Cita pubblicazione | cognome = Hu | nome = MY. | coautori = K. Katchar; L. Kyne; S. Maroo; S. Tummala; V. Dreisbach; H. Xu; DA. Leffler; CP. Kelly | titolo = Prospective derivation and validation of a clinical prediction rule for recurrent Clostridium difficile infection. | rivista = Gastroenterology | volume = 136 | numero = 4 | pp = 1206-14 | mese=aprile| anno = 2009 | doi = 10.1053/j.gastro.2008.12.038 | pmid = 19162027 }}</ref> Mentre il primo caso di [[Colite pseudomembranosa]] era stato documentato da Finney nel 1893 in una autopsia di una donna, morta per una [[diarrea]] emorragica post-intervento chirurgico, che presentava pseudomembrane simil-[[difterite|difteriche]] nel [[colon]].<ref name="Lamont-2002">{{Cita pubblicazione | cognome = Lamont | nome = JT. | titolo = Theodore E. Woodward Award. How bacterial enterotoxins work: insights from in vivo studies. | rivista = Trans Am Clin Climatol Assoc | volume = 113 | pp = 167-80; discussion 180-1 | anno = 2002 | pmid = 12053708 }}</ref><ref name="GillespieHawkey2006">{{Cita libro|autore=Stephen H. Gillespie|autore2=Peter M. Hawkey|titolo=Principles and Practice of Clinical Bacteriology|url=http://books.google.com/books?id=Mfaf4twXsP4C&pg=PA557|data=1º luglio 2006|editore=John Wiley and Sons|pagine=557–|isbn=978-0-470-03532-0}}</ref> |
||
Nel 1968 su criceti, normalmente usati per lo studio del ''C. |
Nel 1968 su criceti, normalmente usati per lo studio del ''C.difficile'', trattati con l'[[antibiotico]] [[lincomicina]] si osservò la comparsa di una grave [[colite pseudomembranosa]] sperimentale mortale.<ref name="GillespieHawkey2006"/> |
||
Nel 1970 animali trattati con antibiotici presentavano un'alta quantità nelle feci di [[tossina]] patogena prodotta dal ''C.difficile'', individuando con la tossina la causa primaria delle manifestazioni cliniche della patologia della colite pseudomembranosa.<ref name="GillespieHawkey2006"/> |
|||
Nel 1980 furono individuate due distinte tossine (A e B) prodotte dal ''C.difficile''.<ref name="Brook-1980">{{Cita pubblicazione | cognome = Brook | nome = I. | titolo = Isolation of toxin producing Clostridium difficile from two children with oxacillin- and dicloxacillin-associated diarrhea. | rivista = Pediatrics | volume = 65 | numero = 6 | pp = 1154-6 | mese=giugno| anno = 1980 | pmid = 7375240 }}</ref> |
|||
Le tossine A e B sono trascritte su un [[locus genico|locus]] di patogenicità che comprende cinque [[gene|geni]]: due geni della tossina, tcdA (tossina A) e tcdB (tossina B), e tre geni regolatori, uno dei quali (tcdC) codifica un regolatore negativo della trascrizione della tossina.<ref name="Chopra-2010">{{Cita pubblicazione | cognome = Chopra | nome = T. | coautori = GJ. Alangaden; P. Chandrasekar | titolo = Clostridium difficile infection in cancer patients and hematopoietic stem cell transplant recipients. | rivista = Expert Rev Anti Infect Ther | volume = 8 | numero = 10 | pp = 1113-9 | mese=ottobre| anno = 2010 | doi = 10.1586/eri.10.95 | pmid = 20954878 }}</ref><ref>{{Cita pubblicazione|nome=Patrizia|cognome=Spigaglia|nome2=Paola|cognome2=Mastrantonio|data=2002-9|titolo=Molecular Analysis of the Pathogenicity Locus and Polymorphism in the Putative Negative Regulator of Toxin Production (TcdC) among Clostridium difficile Clinical Isolates|rivista=Journal of Clinical Microbiology|volume=40|numero=9|pp=3470–3475|accesso=2021-01-22|doi=10.1128/JCM.40.9.3470-3475.2002|url=https://www.ncbi.nlm.nih.gov/pmc/articles/PMC130716/}}</ref> |
|||
La prima sequenza genomica completa di un ceppo di ''C.difficile'' è stata pubblicata per la prima volta nel 2005 dal Sanger Institute nel Regno Unito. Si trattava del ceppo 630, un ceppo virulento e resistente ai farmaci isolato in Svizzera nel 1982. |
|||
Nel 1970 animali trattati con antibiotici presentavano un'alta quantità nelle feci di [[tossina]] patogena prodotta dal ''C. difficile'', individuando con la tossina la causa primaria delle manifestazioni cliniche della patologia della PMC.<ref name="GillespieHawkey2006"/> |
|||
Nel 1980 furono individuate due distinte tossine (A e B) prodotte dal ''C. difficile''.<ref name="Brook-1980">{{Cita pubblicazione | cognome = Brook | nome = I. | titolo = Isolation of toxin producing Clostridium difficile from two children with oxacillin- and dicloxacillin-associated diarrhea. | rivista = Pediatrics | volume = 65 | numero = 6 | pp = 1154-6 | mese=giugno| anno = 1980 | pmid = 7375240 }}</ref> |
|||
Le tossine A e B sono trascritte su un [[locus genico|locus]] di patogenicità che comprende cinque [[gene|geni]]: due geni della tossina, tcdA (tossina A) e tcdB (tossina B), e tre geni regolatori, uno dei quali (tcdC) codifica per un regolatore negativo della trascrizione della tossina.<ref name="Chopra-2010">{{Cita pubblicazione | cognome = Chopra | nome = T. | coautori = GJ. Alangaden; P. Chandrasekar | titolo = Clostridium difficile infection in cancer patients and hematopoietic stem cell transplant recipients. | rivista = Expert Rev Anti Infect Ther | volume = 8 | numero = 10 | pp = 1113-9 | mese=ottobre| anno = 2010 | doi = 10.1586/eri.10.95 | pmid = 20954878 }}</ref> |
|||
== Batteriologia == |
== Batteriologia == |
||
[[File:Clostridium difficile colonies.png|miniatura|Colonie di ''Clostrgaridioides difficile'' su [[Agar agar|agar]]]] |
|||
{{...|medicina}} |
|||
Troviamo spore di ''Clostridioides difficili'' nel terreno, negli ospedali e nelle case per anziani. La forma attiva dei batteri si trova solo nel tratto intestinale . |
|||
Al microscopio, dopo la colorazione di Gram , sono bacilli allungati con un'estremità leggermente gonfia. Il germe stesso è gram-positivo . La sua cultura è ottimale in ambiente basico di agar a 37 ° C . Quando le condizioni si fanno difficili, i batteri producono spore resistenti all'essiccazione, a temperature estreme e a molti prodotti chimici e disinfettanti.<ref>{{Cita pubblicazione|nome=Dale N.|cognome=Gerding|nome2=Carlene A.|cognome2=Muto|nome3=Robert C.|cognome3=Owens|data=2008-01-15|titolo=Measures to control and prevent Clostridium difficile infection|rivista=Clinical Infectious Diseases: An Official Publication of the Infectious Diseases Society of America|volume=46 Suppl 1|pp=S43–49|accesso=2021-01-22|doi=10.1086/521861|url=https://pubmed.ncbi.nlm.nih.gov/18177221/}}</ref><ref>{{Cita pubblicazione|nome=William A.|cognome=Rutala|nome2=Maria F.|cognome2=Gergen|nome3=David J.|cognome3=Weber|data=2012-12|titolo=Efficacy of different cleaning and disinfection methods against Clostridium difficile spores: importance of physical removal versus sporicidal inactivation|rivista=Infection Control and Hospital Epidemiology|volume=33|numero=12|pp=1255–1258|accesso=2021-01-22|doi=10.1086/668434|url=https://pubmed.ncbi.nlm.nih.gov/23143366/}}</ref>. |
|||
Le spore sono metabolicamente dormienti e altamente resistenti alle procedure di disinfezione standard, consentendo loro di persistere per lunghi periodi nell'ambiente. Le spore ingerite da ospiti sensibili possono riattivarsi (o germogliare) in risposta a specifici acidi biliari nell'intestino tenue e tornare a uno stile di vita attivo per produrre tossine e causare malattie. Le spore sono altamente trasmissibili e responsabili della contaminazione degli ambienti |
|||
== Patogenesi == |
|||
Il ''Clostridioides difficile'' è un germe della flora digestiva commensale. Rilevato anche nelle feci degli infanti è spesso asintomatico per l'incapacità della tossina, nei ceppi tossigenici, di legarsi ai recettori degli [[Enterocita|enterociti]] ancora immaturi. Alcune sue mutazioni emergenti sono multi-resistenti (si sono adattate alla maggior parte degli antibiotici) e in caso di disturbo della flora digestiva provocato dalla somministrazione di antibiotici, possono svilupparsi in modo significativo. La grande variabilità dei diversi ceppi rende difficile la loro caratterizzazione molecolare. Quando nel 2003 l'analisi molecolare ha portato all'identificazione di un ceppo di ''C.difficile'' caratterizzato come gruppo BI mediante analisi di endonucleasi di restrizione, NAP1 (nordamericano a campo pulsato) mediante elettroforesi su gel a campo pulsato e ribotipo 027, il "nuovo" ceppo è è stato chiamato " ''C.difficile'' BI / NAP1 / 027 ". La diversa terminologia riflette le tecniche predominanti utilizzate per la tipizzazione epidemiologica.<ref>Spigaglia, P., Barbanti, F., Mastrantonio, P., European Study Group on Clostridium difficile (ESGCD), Ackermann, G., Balmelli, C., … & Drudy, D. (2011). ''[https://academic.oup.com/jac/article/66/10/2227/732177 Multidrug resistance in European Clostridium difficile clinical isolates].'' Journal of Antimicrobial Chemotherapy, 66(10), 2227-2234.</ref><ref>Kuijper, E. J., van den Berg, R. J., Debast, S., Visser, C. E., Veenendaal, D., Troelstra, A., … & Notermans, D. W. (2006). ''[https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3374440/ Clostridium difficile ribotype 027, toxinotype III, the Netherlands]''. Emerging infectious diseases, 12(5), 827.</ref><ref>{{Cita pubblicazione|nome=John G.|cognome=Bartlett|data=2006-11-21|titolo=Narrative Review: The New Epidemic of Clostridium difficile–Associated Enteric Disease|rivista=Annals of Internal Medicine|volume=145|numero=10|pp=758–764|accesso=2021-01-21|doi=10.7326/0003-4819-145-10-200611210-00008|url=https://www.acpjournals.org/doi/10.7326/0003-4819-145-10-200611210-00008}}</ref> |
|||
La diarrea da ''C. difficile'' è causa del 15-20% di tutte le diarree associate all'uso degli antibiotici ed è associata ad alta [[morbilità]] e [[mortalità]] in ambito sanitario. |
|||
Circa il 3-5% dei casi può sviluppare colite pseudomembranosa da ''C. difficile'', con complicazioni gravi come la perforazione, [[Ileo (patologia)|ileo]] grave con [[megacolon]] tossico, [[shock]] settico per [[setticemia]] refrattaria, o morte. |
|||
Del C.difficile esistono sia ceppi tossigenici (cioè produttori di tossine) che non tossigenici, ma solo le forme tossigeniche sono causa di malattia negli esseri umani. |
|||
Un numero significativo di pazienti necessita di [[colectomia]] di emergenza.<ref name="Sailhamer-2009">{{Cita pubblicazione | cognome = Sailhamer | nome = EA. | coautori = K. Carson; Y. Chang; N. Zacharias; K. Spaniolas; M. Tabbara; HB. Alam; MA. DeMoya; GC. Velmahos | titolo = Fulminant Clostridium difficile colitis: patterns of care and predictors of mortality. | rivista = Arch Surg | volume = 144 | numero = 5 | pp = 433-9; discussion 439-40 | mese=maggio| anno = 2009 | doi = 10.1001/archsurg.2009.51 | pmid = 19451485 }}</ref> |
|||
I batteri producono citotossine polipeptidiche ad alta massa molecolare, A e B. Alcuni ceppi producono solo una delle tossine, altri producono entrambe. La tossina A provoca una reazione infiammatoria che coinvolge l'ipersecrezione di liquido e la necrosi emorragica attraverso l'attivazione del rilascio di citochine da parte dei neutrofili. La citotossina B depolimerizza l'actina, la principale proteina del citoscheletro, e quindi aiuta nella distruzione dei tessuti. L'azione combinata delle tossine provoca necrosi dell'epitelio superficiale ed edema (gonfiore fluidico) nelle zone dell'intestino colpite. La proliferazione del ''C.difficile'' è normalmente contrastata dalla normale microflora intestinale, che si ritiene inibisca l'attaccamento del batterio e delle sue tossine alle pareti intestinali. L'alterazione dell'equilibrio microbico intestinale con la terapia antibiotica e la maggiore esposizione al batterio in ambiente ospedaliero consente al ''C.difficile'' di colonizzare individui suscettibili. Inoltre, è stato dimostrato che le concentrazioni subinibitorie di antibiotici promuovono una maggiore produzione di tossine da parte di ''C.difficile'' .<ref>{{Cita pubblicazione|nome=Daniel R.|cognome=Knight|nome2=Briony|cognome2=Elliott|nome3=Barbara J.|cognome3=Chang|data=2015-7|titolo=Diversity and Evolution in the Genome of Clostridium difficile|rivista=Clinical Microbiology Reviews|volume=28|numero=3|pp=721–741|accesso=2021-01-21|doi=10.1128/CMR.00127-14|url=https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4475645/}}</ref><ref name=":0">{{Cita pubblicazione|nome=Mohammed|cognome=Sebaihia|nome2=Brendan W.|cognome2=Wren|nome3=Peter|cognome3=Mullany|data=2006-07|titolo=The multidrug-resistant human pathogen Clostridium difficile has a highly mobile, mosaic genome|rivista=Nature Genetics|volume=38|numero=7|pp=779–786|accesso=2021-01-21|doi=10.1038/ng1830|url=https://pubmed.ncbi.nlm.nih.gov/16804543/}}</ref><ref>{{Cita pubblicazione|nome=Donald C.|cognome=Vinh|data=2005-02-01|titolo=Strains and toxins of Clostridium|rivista=CMAJ : Canadian Medical Association Journal|volume=172|numero=3|pp=312–313|accesso=2021-01-21|doi=10.1503/cmaj.1041642|url=https://www.ncbi.nlm.nih.gov/pmc/articles/PMC545744/}}</ref><ref>{{Cita pubblicazione|nome=L. Clifford|cognome=McDonald|nome2=George E.|cognome2=Killgore|nome3=Angela|cognome3=Thompson|data=2005-12-08|titolo=An Epidemic, Toxin Gene–Variant Strain of Clostridium difficile|rivista=New England Journal of Medicine|volume=353|numero=23|pp=2433–2441|accesso=2021-01-21|doi=10.1056/NEJMoa051590|url=https://doi.org/10.1056/NEJMoa051590}}</ref><ref>{{Cita pubblicazione|nome=Rawish|cognome=Fatima|nome2=Muhammad|cognome2=Aziz|data=2019-01-29|titolo=The Hypervirulent Strain of Clostridium Difficile: NAP1/B1/027 - A Brief Overview|rivista=Cureus|volume=11|numero=1|lingua=en|accesso=2021-01-21|doi=10.7759/cureus.3977|url=https://www.cureus.com/articles/17022-the-hypervirulent-strain-of-clostridium-difficile-nap1b1027---a-brief-overview}}</ref><ref>{{Cita pubblicazione|nome=Gayatri|cognome=Vedantam|nome2=Andrew|cognome2=Clark|nome3=Michele|cognome3=Chu|data=2012-03-01|titolo=Clostridium difficile infection|rivista=Gut Microbes|volume=3|numero=2|pp=121–134|accesso=2021-01-21|doi=10.4161/gmic.19399|url=https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3370945/}}</ref> |
|||
Le manifestazioni cliniche dell'infezione/colonizzazione del ''C. difficile'' sono molto varie per gravità; infatti si possono avere: |
|||
== Ceppi == |
|||
* Colonizzazione asintomatica, |
|||
Il ''C. difficile'' è tassonomicamente distinto da molti altri ben noti clostridi, con una struttura di popolazione diversificata che comprende centinaia di ceppi diversi sparsi in almeno 6 [[Clade|cladi]] filogenetici. Del ''C.difficile'' esistono sia ceppi tossigenici (cioè produttori di tossine) che non tossigenici, ma solo le forme tossigeniche sono causa di malattia negli esseri umani. La patogenicità dipende infatti dalla presenza delle tossine A (TcdA, enterotossina) e B (TcdB, citotossina). Sono stati identificati oltre 400 diversi ceppi tossigenici.<ref>{{Cita pubblicazione|nome=Kate E.|cognome=Dingle|nome2=David|cognome2=Griffiths|nome3=Xavier|cognome3=Didelot|data=19-mag-2011|titolo=Clinical Clostridium difficile: Clonality and Pathogenicity Locus Diversity|rivista=PLOS ONE|volume=6|numero=5|pp=e19993|lingua=en|accesso=2021-01-23|doi=10.1371/journal.pone.0019993|url=https://journals.plos.org/plosone/article?id=10.1371/journal.pone.0019993}}</ref> |
|||
* Diarrea senza colite associata agli antibiotici |
|||
* Colite senza formazione di pseudomenbrane associata all'uso di antibiotici |
|||
* Colite pseudomembranosa, |
|||
* e malattia fulminante con: [[megacolon]] tossico, [[Ileo (patologia)|ileo]], perforazione, [[sepsi]]. |
|||
Tutti i ceppi tossigenici esprimono la tossina TcdB, con o senza la tossina TcdA. La tossina B ha infatti attività citotossica più potente della tossina A. |
|||
Tutte queste manifestazioni sono correlate alla [[CDAD]] (''Clostridioides Difficile Associated Disease'') o malattia associata al ''Clostridioides difficile''. |
|||
Entrambe le tossine sopravvivono nell’ambiente acido dello stomaco e sono in grado di legarsi alla superficie delle cellule epiteliali delle mucose intestinali dove vengono internalizzate e dove catalizzano la glicosilazione di alcune proteine citoplasmatiche, con successiva morte cellulare. Una terza tossina di cui non è chiara la patogenicità, la tossina binaria (CDT), è prodotta da alcuni ceppi di ''C.difficile''. Questa tossina ha dimostrato di incrementare la virulenza di ''C.difficile'' attraverso l’adenosina difosfato-ribosilazione di actina, inducendo la formazione di protrusioni di microtubuli nelle cellule facilitando l’adesione del batterio sulla superficie cellulare. La tossina CDT può essere presente anche nei ceppi dove le tossine A e B sono assenti, riscontrati nei pazienti asintomatici.<ref>{{Cita pubblicazione|nome=C.|cognome=Eckert|nome2=A.|cognome2=Emirian|nome3=A.|cognome3=Le Monnier|data=2015-01|titolo=Prevalence and pathogenicity of binary toxin–positive Clostridium difficile strains that do not produce toxins A and B|rivista=New Microbes and New Infections|volume=3|pp=12–17|lingua=en|accesso=2021-01-23|doi=10.1016/j.nmni.2014.10.003|url=https://linkinghub.elsevier.com/retrieve/pii/S2052297514000055}}</ref> |
|||
Esistono ceppi che producono quantità massive di tossine comparsi sin dal 2000. In particolare, ''C.difficile'' PCR-ribotipo 027 (BI/NAP1/027) è stato associato ad un aumento della gravità dei casi, delle ricorrenze e della mortalità. Il ceppo produce entrambe le tossine in gran quantità a causa di una mutazione (delezione) del gene regolatore tdcC e possiede, analogamente ad altre specie di [[Clostridium|Clostridi]], la tossina binaria CDT. Oltre allo 027, anche altri ribotipi epidemici considerati ipervirulenti sono emersi, alcuni a diffusione internazionale, come lo 078 e lo 018. |
|||
La fisiopatologia della diarrea da ''Clostridioides difficile'' o CDAD richiede prima l'alterazione del microbiota umano dovuta a terapie antibiotiche, quindi la contestuale colonizzazione del ''Clostridioides difficile'', e, infine, se avviene il rilascio successivo di due potenti enterotossine designate come tossina A e tossina B, si manifesta clinicamente la [[Colite pseudomembranosa|PMC]]. |
|||
Una varietà di approcci di tipizzazione molecolare sono stati sviluppati per studiare il ''C.difficile'' e CDI, tra cui: ''multilocus sequence typing'' (MLST), ''multilocus variable-number tandem-repeat analysis (''MLVA), ''surface layer protein A gene sequence typing,'' ''amplificated fragment length polymorphism,'' elettroforesi su gel a campo pulsato (PFGE), analisi dell'endonucleasi di restrizione (REA), tossinotipizzazione (basata sui dati di sequenza delle tossine A e B) e ribotipizzazione PCR.<ref name=":1">{{Cita libro|cognome=European Centre for Disease Prevention and Control.|titolo=Laboratory procedures for diagnosis and typing of human Clostridium difficile infection.|url=https://data.europa.eu/doi/10.2900/04291|accesso=2021-01-23|data=2018|editore=Publications Office|DOI=10.2900/04291}}</ref> Quando nel 2005, l'analisi molecolare ha portato all'identificazione di un tipo di ceppo di ''C.difficile'' caratterizzato come gruppo BI mediante analisi di endonucleasi di restrizione , NAP1 di tipo a campo pulsato nordamericano mediante elettroforesi su gel a campo pulsato e ribotipo 027 mediante ribotipizzazione PCR multiplex è stato chiamato ''C.difficile'' BI/NAP1/027. La diversa terminologia riflette le tecniche predominanti utilizzate per la tipizzazione epidemiologica, anche se la ribotipizzazione PCR è quella più ampiamente accettata in Europa dove l'''Anaerobe Reference Laboratory'' di [[Cardiff]] (UK) tiene la collezione dei ceppi e assegna la numerazione dei ribotipi PCR. In Nord America, PFGE è il metodo di digitazione preferito.<ref>{{Cita web|url=http://www.medscape.com/viewarticle/770003|titolo=The Continually Evolving Clostridium difficile Species|sito=Medscape|accesso=2021-01-23}}</ref> |
|||
Recentemente si è compresa l'importanza della risposta immunitaria, in particolare delle [[IgG]] dirette contro la tossina A,, infatti, ciò è riconosciuto come motivo fondamentale dell'espressione della PMC nell'uomo. Inoltre, le tossine del ''Clostridioides difficile'' sono fattori di virulenza necessari sia negli animali che nell'uomo in quanto i ceppi non produttori di tossine non provocano malattie. |
|||
I primi eventi critici a seguito dell'esposizione alla tossina sono dovuti al rilascio della [[sostanza P]] e di neuropeptidi correlato al [[gene]] della [[calcitonina]] ([[CGRP]]) da neuroni sensoriali afferenti e l'attivazione dei [[macrofago|macrofagi]] della [[lamina propria]] intestinale e [[mastocita|mastociti]]. Questi peptidi, a loro volta fanno rilasciare una complessa cascata di altri mediatori infiammatori dalle cellule della lamina propria.<ref name="Lamont-2002"/> |
|||
Nel Nord America, a partire dal 2016, il ceppo NAP1 è stato sostituito da nuovi ceppi in alcune aree della British Columbia. Questi nuovi ceppi includono NAP2, NAP4, NAP9 e NAP11 e alcuni ceppi che non hanno una designazione NAP. La frequenza di questi nuovi ceppi è aumentata dal 2008 al 2013 in una regione studiata, sostituendo i batteri NAP1 originariamente più comuni e riconoscibili. |
|||
== Epidemiologia e fattori di rischio == |
|||
L'organismo è onnipresente. Colonizza l'intestino di una piccola percentuale di individui sani (<5%). L'esposizione agli antibiotici è associata al suo sviluppo eccessivo e alle manifestazioni patologiche della malattia successive (infezione endogena). |
|||
I principali fattori di rischio per sviluppare l’infezione da ''Clostridioides difficile'' sono: età avanzata, terapia antibiotica, obesità, ipoalbuminemia, ipogammaglobulinemia. |
|||
Poiché il ''C. difficile'' è resistente alla maggior parte degli antibiotici, esso è in grado di svilupparsi durante una terapia antibiotica (dismicrobismo antibiotico indotto). Esso si trasmette da persona a persona per via fecale-orale. Il batterio forma spore resistenti al calore, che si ritrovano in un ospedale o in una casa di cura in soggetti ricoverati per lunghi periodi di tempo. |
|||
Può essere rintracciato in quasi tutte le aree dell'ospedale, infatti, per esempio, le spore possono essere rilevati nelle stanze d'ospedale di pazienti infetti (soprattutto intorno ai letti e bagni) e per questo può essere una fonte di infezione esogena ([[malattia nosocomiale]]). Una volta che le spore vengono ingerite, superano pressoché indenni la barriera gastrica essendo acido-resistenti, e il batterio si evolve nella propria forma vegetativa nel colon, dove si moltiplica. |
|||
In generale, la maggior parte dei metodi di identificazione sono congruenti anche se non si ha una corrispondenza biunivoca tra le diverse tipizzazioni e solo le tecniche MLVA e REA consentono una sufficiente discriminazione tra diversi focolai.<ref>{{Cita pubblicazione|nome=George|cognome=Killgore|nome2=Angela|cognome2=Thompson|nome3=Stuart|cognome3=Johnson|data=2008-02|titolo=Comparison of seven techniques for typing international epidemic strains of Clostridium difficile: restriction endonuclease analysis, pulsed-field gel electrophoresis, PCR-ribotyping, multilocus sequence typing, multilocus variable-number tandem-repeat analysis, amplified fragment length polymorphism, and surface layer protein A gene sequence typing|rivista=Journal of Clinical Microbiology|volume=46|numero=2|pp=431–437|accesso=2021-01-24|doi=10.1128/JCM.01484-07|url=https://pubmed.ncbi.nlm.nih.gov/18039796/}}</ref><ref>{{Cita pubblicazione|nome=Fred C.|cognome=Tenover|nome2=Thomas|cognome2=Åkerlund|nome3=Dale N.|cognome3=Gerding|data=2011-5|titolo=Comparison of Strain Typing Results for Clostridium difficile Isolates from North America▿|rivista=Journal of Clinical Microbiology|volume=49|numero=5|pp=1831–1837|accesso=2021-01-24|doi=10.1128/JCM.02446-10|url=https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3122689/}}</ref> |
|||
I principali fattori di rischio per sviluppare l'infezione da ''C. difficile'' sono: |
|||
* età avanzata,<ref>{{Cita pubblicazione|nome=K. W.|cognome=Garey|data=2008-10|titolo=A clinical risk index for Clostridium difficile infection in hospitalised patients receiving broad-spectrum antibiotics|rivista=The Journal of Hospital Infection|volume=70|numero=2|pp=142–147|accesso=2019-04-15|doi=10.1016/j.jhin.2008.06.026|url=https://www.ncbi.nlm.nih.gov/pubmed/18723249|nome2=T. K.|cognome2=Dao-Tran|nome3=Z. D.|cognome3=Jiang}}</ref> |
|||
* antibioticoterapia,<ref>{{Cita pubblicazione|nome=Robert C.|cognome=Owens, Jr.|data=2008-01-15|titolo=Antimicrobial‐Associated Risk Factors for Clostridium difficile Infection|rivista=Clinical Infectious Diseases|volume=46|numero=s1|pp=S19–S31|lingua=en|accesso=2019-04-15|doi=10.1086/521859|url=https://academic.oup.com/cid/article-lookup/doi/10.1086/521859|nome2=Curtis J.|cognome2=Donskey|nome3=Robert P.|cognome3=Gaynes}}</ref> |
|||
* obesità,<ref>{{Cita pubblicazione|nome=Jihad|cognome=Bishara|data=2013-8|titolo=Obesity as a risk factor for Clostridium difficile infection|rivista=Clinical Infectious Diseases: An Official Publication of the Infectious Diseases Society of America|volume=57|numero=4|pp=489–493|accesso=2019-04-15|doi=10.1093/cid/cit280|url=https://www.ncbi.nlm.nih.gov/pubmed/23645850|nome2=Raymond|cognome2=Farah|nome3=Julnar|cognome3=Mograbi}}</ref> |
|||
* ipoalbuminemia<ref>{{Cita pubblicazione|nome=Alessandra|cognome=di Masi|data=2018-09-22|titolo=Human Serum Albumin Is an Essential Component of the Host Defense Mechanism Against Clostridium difficile Intoxication|rivista=The Journal of Infectious Diseases|volume=218|numero=9|pp=1424–1435|lingua=en|accesso=2019-04-15|doi=10.1093/infdis/jiy338|url=https://academic.oup.com/jid/article/218/9/1424/5032506|nome2=Loris|cognome2=Leboffe|nome3=Fabio|cognome3=Polticelli}}</ref> e |
|||
* ipogammaglobulinemia.<ref>{{Cita pubblicazione|nome=Patricia|cognome=Muñoz|data=2007-9|titolo=Clostridium difficile-associated diarrhea in heart transplant recipients: is hypogammaglobulinemia the answer?|rivista=The Journal of Heart and Lung Transplantation: The Official Publication of the International Society for Heart Transplantation|volume=26|numero=9|pp=907–914|accesso=2019-04-15|doi=10.1016/j.healun.2007.07.010|url=https://www.ncbi.nlm.nih.gov/pubmed/17845929|nome2=Maddalena|cognome2=Giannella|nome3=Luís|cognome3=Alcalá}}</ref> |
|||
I principali database dove sono registrate le caratterizzazioni genetiche dei diversi ceppi di ''C.difficile sono:'' Enterobase<ref>{{Cita web|url=https://enterobase.warwick.ac.uk/species/index/clostridium|titolo=EnteroBase|sito=enterobase.warwick.ac.uk|accesso=2021-01-24}}</ref> ''Public databases for molecular typing and microbial genome diversity'' (PubMLST).<ref>{{Cita web|url=https://pubmlst.org/organisms/clostridioides-difficile|titolo=Clostridioides difficile|sito=PubMLST|lingua=en|accesso=2021-01-24}}</ref> |
|||
Attualmente si stima che questo organismo infetti oltre il 30% dei soggetti ammessi negli ospedali degli Stati Uniti, rendendo la colite da ''C. difficile'' una delle più comuni infezioni nosocomiali. Si stima, inoltre, che nei soli Stati Uniti ogni anno circa 10-12 milioni di adulti siano infettati da questo organismo; di questi, circa un terzo diventa sintomatico. |
|||
Lo sviluppo della malattia negli anziani è particolarmente grave e frequente in quanto questi vengono ricoverati più spesso e per periodi più lunghi.<ref name="Lamont-2002"/> |
|||
Alcuni ceppi, ad esempio RT017 RT078 RT027, possono vivere con basse concentrazioni di zucchero [[trealosio]] ; questi ceppi sono diventati più comuni dopo che il trealosio è stato introdotto come additivo alimentare nei primi anni 2000, aumentando così l'assunzione cibi addizionati di trealosio.<ref>{{Cita pubblicazione|nome=J.|cognome=Collins|nome2=C.|cognome2=Robinson|nome3=H.|cognome3=Danhof|data=2018-01-18|titolo=Dietary trehalose enhances virulence of epidemic Clostridium |
|||
Inoltre, si stima che circa l'1,5% dei pazienti ambulatoriali che ricevono antibiotici svilupperanno la CDAD. |
|||
difficile|rivista=Nature|volume=553|numero=7688|pp=291–294|accesso=2021-01-24|doi=10.1038/nature25178|url=https://www.ncbi.nlm.nih.gov/pmc/articles/PMC5984069/}}</ref><ref>{{Cita pubblicazione|autore=|nome=David W.|cognome=Eyre|autore2=|autore3=|nome2=Xavier|cognome2=Didelot|nome3=Anthony M.|cognome3=Buckley|data=2019-05-01|titolo=Clostridium difficile trehalose metabolism variants are common and not associated with adverse patient outcomes when variably present in the same lineage|rivista=EBioMedicine|volume=43|numero=|pp=347–355|lingua=En|accesso=2021-01-24|doi=10.1016/j.ebiom.2019.04.038|url=https://www.thelancet.com/journals/ebiom/article/PIIS2352-3964(19)30277-4/abstract}}</ref> |
|||
[https://web.archive.org/web/20100623082358/http://sfghdean.ucsf.edu/barnett/FCM/MISC/0610FarahiClostriium.ppt] |
|||
Dal 2005, la morbilità e la mortalità derivanti da ''C. difficile'' sono aumentate costantemente in tutto il mondo in funzione dell'insorgenza di ceppi definiti ipervirulenti (in particolare il ribotipo 027). I meccanismi attualmente proposti che distinguono i ceppi ipervirulenti dai ceppi precedenti (meno virulenti) sono oggetto di controversia. Sono stati proposti: l'aumentato tasso di sporulazione, l'aumentata produzione di tossine , la capacità di sopravanzare i ceppi enedemici nell'intestino dell'ospite ma come questo patogeno sia diventato la principale causa di infezioni nosocomiali rimane sconosciuto e la classificazione di "ceppo ipervirulento" resta arbitraria.<ref>{{Cita pubblicazione|nome=Michelle|cognome=Merrigan|nome2=Anilrudh|cognome2=Venugopal|nome3=Michael|cognome3=Mallozzi|data=2010-10-01|titolo=Human Hypervirulent Clostridium difficile Strains Exhibit Increased Sporulation as Well as Robust Toxin Production|rivista=Journal of Bacteriology|volume=192|numero=19|pp=4904–4911|lingua=en|accesso=2021-01-24|doi=10.1128/JB.00445-10|url=https://jb.asm.org/content/192/19/4904}}</ref><ref>{{Cita pubblicazione|nome=Laith|cognome=Yakob|nome2=Thomas V.|cognome2=Riley|nome3=David L.|cognome3=Paterson|data=2015-07-28|titolo=Mechanisms of hypervirulent Clostridium difficile ribotype 027 displacement of endemic strains: an epidemiological model|rivista=Scientific Reports|volume=5|accesso=2021-01-24|doi=10.1038/srep12666|url=https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4517512/}}</ref>Molti ceppi di C.difficile sono stati rilevati negli animali ed in alimenti ricavati da animali facendo emergere anche l'ipotesi che molte trasmissioni del ''C.difficile'' nell'uomo siano correlate con l'alimentazione di carne o pesce o interspecie.<ref>{{Cita pubblicazione|nome=Alexander|cognome=Rodriguez-Palacios|nome2=Kevin Q|cognome2=Mo|nome3=Bhavan U.|cognome3=Shah|data=2019-11-22|titolo=Global and Historical Distribution of Clostridioides difficile in the Human Diet (1981-2019): Systematic Review and Meta-Analysis of 21886 Samples Reveal Sources of Heterogeneity, High-Risk Foods, and Unexpected Higher Prevalence Towards the Tropic|editore=Infectious Diseases (except HIV/AIDS)|lingua=en|accesso=2021-01-24|doi=10.1101/19012450|url=http://medrxiv.org/lookup/doi/10.1101/19012450}}</ref><ref>{{Cita pubblicazione|nome=Dallas G.|cognome=Hoover|nome2=Alexander|cognome2=Rodriguez-Palacios|data=2013-09|titolo=Transmission of Clostridium difficile in foods|rivista=Infectious Disease Clinics of North America|volume=27|numero=3|pp=675–685|accesso=2021-01-23|doi=10.1016/j.idc.2013.05.004|url=https://pubmed.ncbi.nlm.nih.gov/24011836}}</ref><ref>{{Cita pubblicazione|nome=K.|cognome=Keel|nome2=J. S.|cognome2=Brazier|nome3=K. W.|cognome3=Post|data=2007-06-01|titolo=Prevalence of PCR Ribotypes among Clostridium difficile Isolates from Pigs, Calves, and Other Species|rivista=Journal of Clinical Microbiology|volume=45|numero=6|pp=1963–1964|lingua=en|accesso=2021-01-24|doi=10.1128/JCM.00224-07|url=https://jcm.asm.org/content/45/6/1963}}</ref><ref>{{Cita pubblicazione|nome=Alexander W. W.|cognome=Brown|nome2=Robert B.|cognome2=Wilson|data=2018-08|titolo=Clostridium difficile colitis and zoonotic origins-a narrative review|rivista=Gastroenterology Report|volume=6|numero=3|pp=157–166|accesso=2021-01-23|doi=10.1093/gastro/goy016|url=https://pubmed.ncbi.nlm.nih.gov/30151199/}}</ref><ref>{{Cita pubblicazione|nome=Alan M.|cognome=Mc Govern|nome2=Niki F.|cognome2=Foster|nome3=Lynette A.|cognome3=Pereira|data=2016-08|titolo=Human Clostridium difficile infection caused by a livestock-associated PCR ribotype 237 strain in Western Australia|rivista=JMM case reports|volume=3|numero=4|pp=e005062|accesso=2021-01-23|doi=10.1099/jmmcr.0.005062|url=https://pubmed.ncbi.nlm.nih.gov/28348781/}}</ref><ref>{{Cita pubblicazione|cognome=Luis G Arroyo|cognome2=Stephen A Kruth|cognome3=Barbara M Willey|data=2005-02-01|titolo=PCR ribotyping of Clostridium difficile isolates originating from human and animal sources|rivista=Journal of Medical Microbiology|volume=54|numero=2|lingua=en|accesso=2021-01-23|doi=10.1099/jmm.0.45805-0#tab2|url=https://www.microbiologyresearch.org/content/journal/jmm/10.1099/jmm.0.45805-0#tab2}}</ref> |
|||
Secondo il dott. Erik Dubberke della Washington University di St. Louis, ogni anno negli USA 500.000 americani sono infettati dal ''C. difficile'' in ambiente ospedaliero.<ref name="urlMedscape Network Log In">{{Cita web | url = http://www.medscape.com/viewarticle/750205?src=mp&spon=20 | titolo = Medscape Network Log In | autore = | data = | lingua = en | accesso=28 settembre 2011}}</ref> |
|||
== Genoma == |
|||
Il pan-genoma del C.difficile è composto da un genoma centrale (quei geni presenti in tutti gli isolati) e da un genoma accessorio o adattativo (geni assenti da uno o più ceppi o unici per un particolare ceppo). |
|||
Una grande percentuale (11%) del genoma è costituita da elementi genetici mobili, principalmente sotto forma di [[Trasposone|trasposoni]] coniugativi. |
|||
==Segni e sintomi== |
|||
{{vedi anche|Colite pseudomembranosa|CDAD}} |
|||
La consistenza, la quantità, l'odore e il colore delle feci sono importanti per avere un primo campanello d'allarme, di solito quando siamo in presenza di questo batterio le feci risultano molto abbondanti, scariche diarroiche con feci non formate, odore acido, molto forte e color ocra con ristagno di sedimenti filamentosi somiglianti a gelatina. |
|||
La [[colite pseudomembranosa]] è la maggiore e più grave patologia provocata dal ''C. difficile''. Prevede l'instaurarsi del microrganismo con conseguente produzione di [[tossina|tossine]] che andranno ad interagire con gli enterociti glicosilando numerose proteine e avviando meccanismi [[apoptosi|apoptotici]]. La liberazione di [[citochina|citochine]] in concomitanza con la morte cellulare richiama in loco un cospicuo numero di cellule della difesa immunitaria le quali, mantenendo lo stato di infiammazione, danneggeranno ulteriormente le cellule. |
|||
L'accumulo di detriti cellulari muco ed altro sulla mucosa intestinale produce la ''pseudomembrana'' che andrà progressivamente espandendosi alla mucosa intestinale. |
|||
L'analisi ha identificato quattro distinti cluster statisticamente supportati comprendenti un clade ipervirulento, una tossina A - B +clade e due cladi con isolati umani e animali. Le differenze genetiche tra i cladi hanno rivelato diverse isole genetiche relative alla virulenza e all'adattamento di nicchia, tra cui resistenza agli antibiotici, motilità, adesione e metabolismo enterico. Solo il 19,7% dei geni era condiviso da tutti i ceppi, |
|||
== Diagnosi == |
|||
La diagnosi si basa su test immunoenzimatici (CLIA) sulle feci in cui viene innanzitutto ricercato l'enzima glutammato deidrogenasi (GDH), prodotto da tutti i ceppi di Clostridium. Se il campione risulta GDH positivo, si procede alla ricerca delle tossine A e B, prima con test immunoenzimatico e poi con un test molecolare di conferma. |
|||
Il genoma di C.difficile è ampio e geneticamente diverso, mostrando livelli notevoli di plasticità e livelli bassissimi di conservazione tra i ceppi. |
|||
=== Test sulle feci === |
|||
Tramite test sulle feci si può ottenere tempestivamente una risposta sulla presenza del batterio. |
|||
''C. difficile'' possiede un genoma "aperto" con livelli estremi di plasticità, con accesso e scambio frequente con più ambienti ospiti e pool genetici batterici. Di conseguenza, poiché vengono sequenziati più ceppi di C.difficile da linee divergenti e diverse fonti animali e ambientali, questa stima aumenterà probabilmente. |
|||
=== ELISA === |
|||
{{vedi anche|ELISA}} |
|||
ELISA è un acronimo derivato dall'espressione inglese enzyme-linked immunosorbent assay. Si tratta di un versatile metodo d'analisi immunologica usato in biochimica per rilevare la presenza di una sostanza usando uno o più anticorpi ad uno dei quali è legato un enzima: tale metodica d'indagine rientra nella categoria dei test immunoenzimatici |
|||
La prima sequenza genomica completa di un ceppo di C.difficile è stata pubblicata per la prima volta nel 2005 dal Sanger Institute nel Regno Unito. Si trattava del ceppo 630, un ceppo virulento e resistente ai farmaci isolato in Svizzera nel 1982. Gli scienziati del Sanger Institute hanno sequenziato genomi di circa 30 [[Coltura pura|isolati]] di ''C.difficile'' utilizzando tecnologie di sequenziamento di nuova generazione da 454 Life Sciences e Illumina . |
|||
=== PCR Real time === |
|||
{{vedi anche|Reazione a catena della polimerasi}} |
|||
{{...|medicina}} |
|||
I ricercatori della McGill University di Montreal hanno sequenziato il genoma del ceppo di C.difficile ''del'' Quebec altamente virulento nel 2005 utilizzando una tecnologia di sequenziamento ad altissima velocità. I test hanno comportato l'esecuzione di 400.000 reazioni di sequenziamento parallelo del DNA del genoma del batterio, che era stato frammentato per il sequenziamento. Queste sequenze sono state assemblate computazionalmente per formare una sequenza genomica completa. |
|||
=== CT === |
|||
{{vedi anche|tomografia computerizzata}} |
|||
{{...|medicina}} |
|||
Nel 2012, gli scienziati dell'Università di Oxford hanno sequenziato i genomi di C.difficile da 486 casi verificatisi in quattro anni nell'Oxfordshire utilizzando le tecnologie di sequenziamento di nuova generazione. |
|||
== Prevenzione == |
|||
{{...|medicina}} |
|||
Nel 2006, Sebaihia e colleghi hanno descritto il primo genoma chiuso completamente sequenziato e annotato di C.difficile (ceppo 630; RT012).<ref name=":0" /> Questo ceppo virulento, altamente trasmissibile e multiresistente di C.difficile ''è'' stato originariamente isolato nel 1982 da un paziente con PMC a Zurigo, Svizzera. Il sequenziamento e l'annotazione del ceppo 630 hanno rivelato un grande cromosoma circolare di 4.290.252 bp (4,3 Mb), 3.776 sequenze putative di codifica delle proteine (CDS) e un contenuto di GC del 29,06% . È stato anche identificato un plasmide circolare (pCD630) di 7.881 bp contenente 11 CDS .<ref name=":0" /> Cinque anni dopo, Monot e colleghi<ref>{{Cita pubblicazione|nome=Marc|cognome=Monot|nome2=Caroline|cognome2=Boursaux-Eude|nome3=Marie|cognome3=Thibonnier|data=2011-08|titolo=Reannotation of the genome sequence of Clostridium difficile strain 630|rivista=Journal of Medical Microbiology|volume=60|numero=Pt 8|pp=1193–1199|accesso=2021-01-24|doi=10.1099/jmm.0.030452-0|url=https://pubmed.ncbi.nlm.nih.gov/21349987/}}</ref> hanno riannotato il genoma del ceppo 630 utilizzando un approccio trascrittomico e proteomico combinato per aggiornare le funzioni putative di> 500 geni precedentemente putativi o sconosciuti. Da allora, molti altri genomi di dimensioni comprese tra 4,1 e 4,3 Mbp sono stati completamente sequenziati e annotati: CD37 (RT009; isolato negli Stati Uniti nel 1980), M68 (RT017; isolato in Irlanda nel 2006), CF5 (RT017; isolato in Belgio nel 1995), M120 (RT078; isolato nel Regno Unito nel 2007), G46 (RT027; isolato nel Regno Unito nel 2006), R20291(RT027; isolato nel Regno Unito nel 2006), 196 (RT027; isolato in Francia nel 1985), 2007855 (RT027; isolato negli Stati Uniti nel 2007) e BI1 (RT027; isolato negli Stati Uniti nel 1988).<ref>{{Cita pubblicazione|nome=Tom|cognome=Gaulton|nome2=Raju|cognome2=Misra|nome3=Graham|cognome3=Rose|data=2015-03-26|titolo=Complete Genome Sequence of the Hypervirulent Bacterium Clostridium difficile Strain G46, Ribotype 027|rivista=Genome Announcements|volume=3|numero=2|accesso=2021-01-24|doi=10.1128/genomeA.00073-15|url=https://pubmed.ncbi.nlm.nih.gov/25814591/}}</ref><ref>{{Cita pubblicazione|nome=Michael S. M.|cognome=Brouwer|nome2=Elaine|cognome2=Allan|nome3=Peter|cognome3=Mullany|data=2012-04|titolo=Draft genome sequence of the nontoxigenic Clostridium difficile strain CD37|rivista=Journal of Bacteriology|volume=194|numero=8|pp=2125–2126|accesso=2021-01-24|doi=10.1128/JB.00122-12|url=https://pubmed.ncbi.nlm.nih.gov/22461557/}}</ref><ref>{{Cita pubblicazione|nome=Miao|cognome=He|nome2=Mohammed|cognome2=Sebaihia|nome3=Trevor D.|cognome3=Lawley|data=2010-04-20|titolo=Evolutionary dynamics of Clostridium difficile over short and long time scales|rivista=Proceedings of the National Academy of Sciences of the United States of America|volume=107|numero=16|pp=7527–7532|accesso=2021-01-24|doi=10.1073/pnas.0914322107|url=https://pubmed.ncbi.nlm.nih.gov/20368420/}}</ref> I genomi di riferimento come quelli dei ceppi 630 e M120 svolgono un ruolo importante nella pipeline di analisi dei dati NGS. Comprendono una sequenza inequivocabile e contigua di nucleotidi noti che attraversano l'intero cromosoma e plasmidi (se presenti), fornendo quindi un riferimento di altissima qualità per la mappatura dei genomi in bozza. |
|||
== Trattamento == |
|||
=== Farmacologico === |
|||
Si basa su una terapia [[antibiotico|antibiotica]], eventualmente associata alla somministrazione di Saccharomyces boulardii oppure Lattobacilli ([[probiotico|probiotici]])<ref name="Badger-2012">{{Cita pubblicazione | cognome = Badger | nome = VO. | coautori = NA. Ledeboer; MB. Graham; CE. Edmiston | titolo = Clostridium difficile: Epidemiology, Pathogenesis, Management, and Prevention of a Recalcitrant Healthcare-Associated Pathogen. | rivista = JPEN J Parenter Enteral Nutr | mese=maggio| anno = 2012 | doi = 10.1177/0148607112446703 | pmid = 22577120 }}</ref><ref name="Malaguarnera-2012">{{Cita pubblicazione | cognome = Malaguarnera | nome = G. | coautori = F. Leggio; M. Vacante; M. Motta; M. Giordano; A. Bondi; F. Basile; S. Mastrojeni; A. Mistretta; M. Malaguarnera; MA. Toscano | titolo = Probiotics in the gastrointestinal diseases of the elderly. | rivista = J Nutr Health Aging | volume = 16 | numero = 4 | pp = 402-10 | mese=aprile| anno = 2012 | pmid = 22499466 }}</ref><ref name="Morrow-2012">{{Cita pubblicazione | cognome = Morrow | nome = LE. | coautori = V. Gogineni; MA. Malesker | titolo = Probiotics in the intensive care unit. | rivista = Nutr Clin Pract | volume = 27 | numero = 2 | pp = 235-41 | mese=aprile| anno = 2012 | doi = 10.1177/0884533612440290 | pmid = 22473797 }}</ref><ref name="Moudgal-2012">{{Cita pubblicazione | cognome = Moudgal | nome = V. | coautori = JD. Sobel | titolo = Clostridium difficile colitis: a review. | rivista = Hosp Pract (Minneap) | volume = 40 | numero = 1 | pp = 139-48 | mese=febbraio| anno = 2012 | doi = 10.3810/hp.2012.02.954 | pmid = 22406889 }}</ref> |
|||
In sintesi, gli studi MLST su diverse raccolte di ceppi di ''C. difficile'' suggeriscono che ci siano cinque [[Stipite virale|lignaggi]] o [[Clade|cladi]] che possono generalmente essere suddivisi per i più importanti ribotipi PCR; 027, 017, 023, 078 e un ampio gruppo comprendente il resto dei ribotipi PCR che rappresenta un lignaggio interessante altamente divergente. |
|||
*[[Metronidazolo]] a dosi di 250 mg per os 4 volte al dì per 7-10 gg, |
|||
== Epigenoma == |
|||
*[[Vancomicina]] a dosi di 125 mg per os 4 volte al dì per 7-10 gg, o in alternativa |
|||
''C. difficile'' ha un [[Epigenetica|epigenoma]] altamente diversificato, con 17 motivi di metilazione di alta qualità segnalati finora, la maggior parte relativi al tipo 6mA. È stato dimostrato che la metilazione in uno di questi motivi, CAAAA A, altamente conservato, influisce sulla sporulazione, un passaggio chiave nella trasmissione della malattia da C.difficile , nonché sulla lunghezza delle cellule, sulla formazione di biofilm e sulla colonizzazione dell'ospite.<ref>{{Cita pubblicazione|nome=Pedro H.|cognome=Oliveira|nome2=John W.|cognome2=Ribis|nome3=Elizabeth M.|cognome3=Garrett|data=2020-1|titolo=Epigenomic characterization of Clostridioides difficile finds a conserved DNA methyltransferase that mediates sporulation and pathogenesis|rivista=Nature microbiology|volume=5|numero=1|pp=166–180|accesso=2021-01-22|doi=10.1038/s41564-019-0613-4|url=https://www.ncbi.nlm.nih.gov/pmc/articles/PMC6925328/}}</ref> |
|||
*[[Colestiramina]] a dosi di 4 g per os 3 volte al dì per 10 gg (terapia ormai superata). |
|||
* [[Fidaxomicina]] a dosi di 200 mg per os 2 volte al dì per 10 gg |
|||
== Batteriofagi == |
|||
=== Batterioterapia fecale === |
|||
Un altro componente importante del [[mobiloma]] di ''C.difficile'' sono i batteriofagi (fagi). I fagi si sono evoluti con il ''C.difficile'' per periodi di tempo molto lunghi e l'infezione dei fagi è una parte intrinseca della storia naturale e della biologia del C.difficile . l'acquisizione di fagi e la loro perdita dal genoma di ''C.difficile'' sono eventi genetici significativi che hanno avuto un impatto sull'evoluzione dell'ospite.<ref>{{Cita pubblicazione|nome=Katherine R.|cognome=Hargreaves|nome2=Martha R. J.|cognome2=Clokie|data=2014-04-28|titolo=Clostridium difficile phages: still difficult?|rivista=Frontiers in Microbiology|volume=5|accesso=2021-01-22|doi=10.3389/fmicb.2014.00184|url=https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4009436/}}</ref> |
|||
{{vedi anche|Batterioterapia fecale}} |
|||
Di recente la [[batterioterapia fecale]] è stata sviluppata dal Team del Dr. [[Thomas Borody|Thomas J. Borody]] a [[Sydney]], Australia, come trattamento alternativo nella Colite pseudomembranosa, specie nelle forme recidivanti e resistenti alla terapia con [[vancomicina]].<ref name="Borody-2003">{{Cita pubblicazione | cognome = Borody | nome = TJ. | coautori = EF. Warren; S. Leis; R. Surace; O. Ashman | titolo = Treatment of ulcerative colitis using fecal bacteriotherapy. | rivista = J Clin Gastroenterol | volume = 37 | numero = 1 | pp = 42-7 | mese=luglio| anno = 2003 | pmid = 12811208 }}</ref><ref name="Borody-2004">{{Cita pubblicazione | cognome = Borody | nome = TJ. | coautori = EF. Warren; SM. Leis; R. Surace; O. Ashman; S. Siarakas | titolo = Bacteriotherapy using fecal flora: toying with human motions. |rivista = J Clin Gastroenterol | volume = 38 | numero = 6 | pp = 475-83 | mese=luglio| anno = 2004 | pmid = 15220681 }}</ref> |
|||
Questa strategia terapeutica è stato a lungo usata nell'allevamento degli animali, per esempio, per evitare la [[salmonella|salmonellosi]] nei polli.<ref name="Nurmi-1973">{{Cita pubblicazione | cognome = Nurmi | nome = E. | coautori = M. Rantala | titolo = New aspects of Salmonella infection in broiler production. | rivista = Nature | volume = 241 | numero = 5386 | pp = 210-1 | mese=gennaio| anno = 1973 | pmid = 4700893 }}</ref> |
|||
Inoltre, è utilmente utilizzata su [[organismo modello|topi di laboratorio]] [[germ free]] per rendere ''umanizzate'' le feci degli stessi animali, in modo da poter studiare gli effetti di farmaci o altro sull'ecosistema umano riprodotto nel topo.<ref name="Turnbaugh-2009">{{Cita pubblicazione | cognome = Turnbaugh | nome = PJ. | coautori = VK. Ridaura; JJ. Faith; FE. Rey; R. Knight; JI. Gordon | titolo = The effect of diet on the human gut microbiome: a metagenomic analysis in humanized gnotobiotic mice. |rivista = Sci Transl Med | volume = 1 | numero = 6 | pp = 6ra14 | mese=novembre| anno = 2009 | doi = 10.1126/scitranslmed.3000322 | pmid = 20368178 }}</ref> |
|||
Almeno otto [[Batteriofago|batteriofagi]], principalmente temperati cioè lisogeni, sono stati isolati dal C.difficile , con dimensioni del genoma comprese tra circa 30 e circa 60 kb. Sia i ceppi di ''C.difficile'' ''di origine'' ambientale che quelli di origine clinica sono portatori di un insieme diversificato e prevalente di [[Profago|profagi]] . |
|||
=== Chirurgico === |
|||
== Rilevanza clinica == |
|||
{{...|medicina}} |
|||
{{Vedi anche|Infezione da Clostridioides difficile}} |
|||
Solo i ceppi di ''Clostridioides difficile'' che producono tossine sono di rilevanza clinica. |
|||
=== Terapie sperimentali === |
|||
* CDA-1 e CDB-1 (noto anche come: MDX-066/MDX-1388 e MBL-CDA1/MBL-CDB1) è un [[anticorpo monoclonale]] sperimentale che ha completato uno studio di [[Fase II]] su 200 pazienti con colite pseudomembranosa.<ref name=Campus>{{Cita pubblicazione | titolo = op-line data from randomized, double-blind, placebo controlled Phase 2 clinical trial indicate statistically significant reduction in recurrences of CDAD | url = http://www.umassmed.edu/Content.aspx?id=62670 | autore = Campus, University of Massachusetts Worcester | accesso = 16 agosto 2011 | urlmorto = sì | urlarchivio = https://web.archive.org/web/20101227152634/http://umassmed.edu/Content.aspx?id=62670 | dataarchivio = 27 dicembre 2010 }}</ref><ref name=CenterWatch>{{Cita pubblicazione | titolo = Clostridium Difficile-Associated Diarrhea | url = http://www.centerwatch.com/clinical-trials/results/new-therapies/nmt-details.aspx?CatID=554 | autore = CenterWatch | accesso = 16 agosto 2011 | urlarchivio = https://web.archive.org/web/20110929154200/http://www.centerwatch.com/clinical-trials/results/new-therapies/nmt-details.aspx?CatID=554 | dataarchivio = 29 settembre 2011 | urlmorto = sì }}</ref><ref name=Business>{{Cita pubblicazione | titolo = MDX 066, MDX 1388 Medarex, University of Massachusetts Medical School clinical data (phase II)(diarrhea) | url = http://business.highbeam.com/436989/article-1G1-193894218/mdx-066-mdx-1388-medarex-university-massachusetts-medical | autore = Business, Highbeam | accesso = 16 agosto 2011 | urlarchivio = https://web.archive.org/web/20121014202844/http://business.highbeam.com/436989/article-1G1-193894218/mdx-066-mdx-1388-medarex-university-massachusetts-medical | dataarchivio = 14 ottobre 2012 | urlmorto = sì }}</ref> |
|||
*[[Nitazoxanide]],<ref name="Shah-2010">{{Cita pubblicazione | cognome = Shah | nome = D. | coautori = MD. Dang; R. Hasbun; HL. Koo; ZD. Jiang; HL. DuPont; KW. Garey | titolo = Clostridium difficile infection: update on emerging antibiotic treatment options and antibiotic resistance. | rivista = Expert Rev Anti Infect Ther | volume = 8 | numero = 5 | pp = 555-64 | mese=maggio| anno = 2010 | doi = 10.1586/eri.10.28 | pmid = 20455684 }}</ref> |
|||
* [[Rifaximina]],<ref name="Shah-2010"/> |
|||
* [[Rifalazil]],<ref name="Shah-2010"/> |
|||
* [[Fidaxomicin]],<ref name="Shah-2010"/> |
|||
* [[Nitazoxanide]],<ref name="Shah-2010"/> |
|||
* [[Tigeciclina]]<ref name="Shah-2010"/> e |
|||
* [[Ramoplanina]].<ref name="Shah-2010"/> |
|||
La rapida evoluzione della resistenza agli antibiotici nel ''C.difficile''<ref>{{Cita pubblicazione|nome=Saeed S.|cognome=Banawas|data=2018-02-21|titolo=Clostridium difficile Infections: A Global Overview of Drug Sensitivity and Resistance Mechanisms|rivista=BioMed Research International|volume=2018|accesso=2021-01-24|doi=10.1155/2018/8414257|url=https://www.ncbi.nlm.nih.gov/pmc/articles/PMC5841113/}}</ref> e i conseguenti effetti sulla prevenzione e il trattamento delle infezioni da ''C. difficile'' sono motivo di preoccupazione per la salute pubblica. <ref>{{Cita pubblicazione|nome=Patrizia|cognome=Spigaglia|nome2=Paola|cognome2=Mastrantonio|nome3=Fabrizio|cognome3=Barbanti|data=2018|titolo=Antibiotic Resistances of Clostridium difficile|rivista=Advances in Experimental Medicine and Biology|volume=1050|pp=137–159|accesso=2021-01-24|doi=10.1007/978-3-319-72799-8_9|url=https://pubmed.ncbi.nlm.nih.gov/29383668/}}</ref> |
|||
== Prognosi == |
|||
{{...|medicina}} |
|||
I fattori di [[virulenza]] del ''C.difficile'' sono: [[enterotossina]], spore e [[ialuronidasi]]. Questo bacillo si trova normalmente nel [[microbiota umano]] per cui, se si utilizzano per lungo tempo antibiotici, questi possono distruggere anche quei batteri che tengono confinato il C.difficile il quale può prendere il sopravvento e provocare crampi addominali e malattie varie (colite pseudomembranosa). Queste malattie hanno un decorso benigno e autolimitanti tranne nel caso in cui ci sono complicanze che possono compromettere la parete intestinale con un possibile passaggio in circolo del bacillo, con conseguente [[sepsi]] e quindi morte dell'individuo. |
|||
== Recidive == |
|||
Solitamente dopo un primo trattamento con metronidazolo o vancomicina, ''Clostridioides difficile'' sviluppa nuove ricorrenze in circa il 20% delle persone. Dopo le successive recidive i tassi di ricorrenza aumentano al 40% e 60%.<ref name="Kelly-2008"/><ref name="Shah-2010"/><ref name="Freeman-2010">{{Cita pubblicazione | cognome = Freeman | nome = J. | coautori = MP. Bauer; SD. Baines; J. Corver; WN. Fawley; B. Goorhuis; EJ. Kuijper; MH. Wilcox | titolo = The changing epidemiology of Clostridium difficile infections. | rivista = Clin Microbiol Rev | volume = 23 | numero = 3 | pp = 529-49 | mese=luglio| anno = 2010 | doi = 10.1128/CMR.00082-09 | pmid = 20610822 }}</ref><ref name="Bouza-2009">{{Cita pubblicazione | cognome = Bouza | nome = E. | titolo = Clostridium difficile infection: same incidence and worse prognosis? | rivista = Clin Infect Dis | volume = 48 | numero = 5 | pp = 577-9 | mese=marzo| anno = 2009 | doi = 10.1086/596704 | pmid = 19191642 }}</ref><ref name="Davey-2006">{{Cita pubblicazione | cognome = Davey | nome = P. | coautori = E. Brown; L. Fenelon; R. Finch; I. Gould; A. Holmes; C. Ramsay; E. Taylor; P. Wiffen; M. Wilcox | titolo = Systematic review of antimicrobial drug prescribing in hospitals. | rivista = Emerg Infect Dis | volume = 12 | numero = 2 | pp = 211-6 | mese=febbraio| anno = 2006 | pmid = 16494744 }}</ref><ref name="Blossom-2007">{{Cita pubblicazione | cognome = Blossom | nome = DB. | coautori = LC. McDonald | titolo = The challenges posed by reemerging Clostridium difficile infection. | rivista = Clin Infect Dis | volume = 45 | numero = 2 | pp = 222-7 | mese=luglio| anno = 2007 | doi = 10.1086/518874 | pmid = 17578783 }}</ref> |
|||
== Epidemie storiche == |
|||
<!-- 4 giugno 2003, due focolai di un ceppo molto virulento di questo batterio sono stati riportati in Montreal e Calgary, Alberta , in Canada . Fonti inserire il conteggio morte al livello più basso 36 e alto come 89, con circa 1.400 casi nel 2003 e nei primi mesi del 2004. ''C. difficile'' infezioni continuato ad essere un problema nel sistema sanitario Quebec alla fine del 2004. A partire dal marzo 2005, si era diffuso nella Toronto Area, ospedalizzare 10 persone. Uno è morto mentre gli altri venivano dimessi. |
|||
Una epidemia simile ha avuto luogo presso Stoke Mandeville Hospital nel Regno Unito tra il 2003 e il 2005. Il locale epidemiologia di ''C. difficile'' può offrire indizi su come la sua diffusione può riguardare la quantità di tempo che un paziente trascorre in ospedale e / o un centro di riabilitazione. E 'anche campioni capacità delle istituzioni' di rilevare i tassi di aumento, e la loro capacità di rispondere con più aggressivo il lavaggio delle mani campagne, i metodi di quarantena e la disponibilità di yogurt contenente colture vive di pazienti a rischio di infezione. |
|||
È stato suggerito che entrambi i focolai canadesi e inglesi sono stati correlati agli apparentemente più virulento ceppo NAP1/027 del batterio. Questo ceppo romanzo, noto anche come Quebec ceppo , è stata anche coinvolta in una epidemia in due ospedali olandesi ( Harderwijk e Amersfoort , entrambi del 2005). Una teoria per spiegare la maggiore virulenza dei 027 è che si tratta di una hyperproducer sia di tossine A e B, e che alcuni antibiotici possano effettivamente stimolare il batterio a hyperproduce. |
|||
1 OTTOBRE 2006, ''C. difficile'' è stato detto di avere ucciso almeno 49 persone negli ospedali di Leicester , in Inghilterra più di otto mesi, secondo un Servizio Sanitario Nazionaleindagine. Altri 29 casi simili sono stati studiati da medici legali .<ref>{{Cita news |titolo=Trust confirms 49 superbug deaths |newspaper=[[BBC News]]|url=http://news.bbc.co.uk/1/hi/england/leicestershire/5396800.stm}}</ref> Il dipartimento britannico della Salute memo trapelato poco dopo rivelato notevole preoccupazione nel governo circa il batterio, descritto come "endemica in tutto il servizio sanitario".<ref>{{Cita news| url= http://www.timesonline.co.uk/article/0,,2-2541472,00.html| titolo=Leaked memo reveals that targets to beat MRSA will not be met| autore=Nigel Hawkes| data=2007-01-11| pubblicazione=The Times| accesso = 2007-01-11 | location=London}}</ref> |
|||
27 ottobre 2006, 9 decessi sono stati attribuiti al batterio in Quebec, Canada.<ref>{{Cita web| url=http://cnews.canoe.ca/CNEWS/Canada/2006/10/27/2145519.html| titolo=C. difficile blamed for 9 death in hospital near Montreal|data=11 January 200| opera=cNews| accesso = 2007-01-11}}</ref> |
|||
18 novembre 2006, il batterio è stato segnalato per essere stato responsabile di 12 morti in Quebec, Canada. Questa morte 12 riportata è stata solo due giorni dopo la Hyacinthe St. Honoré Mercier ha annunciato che l'epidemia era sotto controllo. Trentuno pazienti sono stati diagnosticati con ''Clostridioides difficile'' e quattro (al sab 18 nov) erano ancora sotto osservazione. Addetti alle pulizie ha adottato misure nel tentativo di eliminare il focolaio. |
|||
''C. difficile'' è stato citato su 6.480 certificati di morte nel 2006 in Gran Bretagna.<ref>{{Cita news |titolo=12th person dies of C. difficile at Quebec hospital |newspaper=[[CBC News]] |data=2006-November-18 |url=http://www.cbc.ca/canada/story/2006/11/18/difficile-outbreak.html}}</ref> |
|||
27 febbraio 2007, un nuovo focolaio è stato identificato a Trillium Health Centre a Mississauga, Ontario, dove sono stati diagnosticati 14 persone con i batteri. I batteri erano dello stesso ceppo di quello in Quebec. I funzionari non sono stati in grado di determinare se ''C. difficile'' è stato responsabile morte di quattro pazienti oltre i precedenti due mesi.<ref>[http://toronto.ctv.ca/servlet/an/local/CTVNews/20070228/cdifficile_mississauga_outbreak_070228/20070228/?hub=TorontoHome CTV Toronto - C. difficile outbreak linked to fatal strain - CTV News, Shows and Sports - Canadian Television]</ref> |
|||
Tra febbraio e giugno 2007, tre pazienti a Loughlinstown Hospital di Dublino, in Irlanda sono stati trovati dal medico legale sia morto a causa di ''C. difficile'' l'infezione. In un'inchiesta, la Corte del medico legale ha scoperto che l'ospedale non aveva designato team di controllo delle infezioni o microbiologo consulente personale.<ref>{{Cita news |titolo=Superbug in hospitals linked to four deaths|newspaper=Irish Independent |data=2007-October-10|url=http://www.independent.ie/national-news/superbug-in-hospitals-linked-to-four-deaths-1139062.html}}</ref> |
|||
Ottobre 2007, Maidstone e Tunbridge Wells NHS Trust è stato fortemente criticato dalla Commissione Sanità in merito alla sua gestione di una grande epidemia di ''C. difficile'' nei suoi ospedali in Kent da aprile 2004 a settembre 2006. Nella sua relazione, la Commissione stima che circa 90 pazienti ", sicuramente o probabilmente" morti in seguito dell'infezione.<ref>Healthcare Commission press release:[http://www.healthcarecommission.org.uk/newsandevents/pressreleases.cfm/cit_id/5875/FAArea1/customWidgets.content_view_1/usecache/falseHealthcare watchdog finds significant failings in infection control at Maidstone and Tunbridge Wells NHS Trust], 11 October 2007</ref><ref>{{Cita news |titolo=Health Secretary intervenes in superbug row |newspaper=Daily Telegraph |data=2007-October-11|url=http://www.telegraph.co.uk/news/main.jhtml?xml=/news/2007/10/11/ncdiff611.xml}}</ref> |
|||
Novembre 2007, il ceppo 027 è diffuso in diversi ospedali nel sud della Finlandia, con dieci morti su 115 pazienti infetti riportato 2007/12/14.<ref>[http://www.hs.fi/kotimaa/artikkeli/%C3%84rh%C3%A4kk%C3%A4+suolistobakteeri+on+tappanut+jo+kymmenen+potilasta/1135232592793Ärhäkkä suolistobakteeri on tappanut jo kymmenen potilasta - HS.fi - Kotimaa]</ref> |
|||
Novembre 2009, quattro morti a Nostra Signora di Lourdes Hospital in Irlanda, sono possibili collegamenti a ''Clostridioides difficile'' l'infezione. Un ulteriore 12 pazienti sono risultati positivi per l'infezione, e altri 20 mostrano segni di infezione, 10 novembre 2009.<ref>{{Cita news|titolo=Possible C Diff link to Drogheda deaths |newspaper=RTE News |data=2009-November-10|url=http://www.rte.ie/news/2009/1110/health.html}}</ref> |
|||
Marzo 2010, Da febbraio 2009 a febbraio 2010 199 pazienti di Herlev ospedale in Danimarca era sospettato di essere infettati con il ceppo 027. Nella prima metà del 2009, 29 morti negli ospedali di Copenaghen dopo essere stati infettati dal batterio.<ref>[http://www.bt.dk/sygdomme/199-ramt-af-draeber-diarre-paa-herlev-sygehus 199 hit by the killer diarrhea at Herlev Hospital ], BT 3'th march 2010</ref> |
|||
Maggio 2010, un totale di 138 pazienti in quattro diversi ospedali in Danimarca infettati con il ceppo 027 (Herlev, Amager, Gentofte e Hvidovre) oltre ad alcuni fatti isolati in altri ospedali.<ref>[http://nyhederne.tv2.dk/article/30384149/ Four hospitals affected by the dangerous bacterium ] {{Webarchive|url=https://web.archive.org/web/20130705140243/http://nyhederne.tv2.dk/article/30384149/ |data=5 luglio 2013 }}, TV2 News May 7'th 2010</ref> |
|||
28 maggio 2011 un focolaio in Ontario, in Canada è stata riportata, con 26 decessi a partire dal 24 luglio, 2011.<ref>{{Cita news|titolo=C. difficile linked to 26th death in Ontario|url=http://www.cbc.ca/news/canada/ottawa/story/2011/07/24/tor-c-difficile-death-stcatharines623.html|accesso=2011-July-24|newspaper=CBC News|data=2011-July-25}}</ref> --> |
|||
L'isolamento può avvenire tramite la [[coprocoltura]] in [[Terreno selettivo per Clostridium difficile|terreni selettivi per C.difficile]]. L'enterotossina può essere saggiata con il metodo [[ELISA]], mentre la citotossina con rivelazione di tossicità in colture cellulari. |
|||
== Note == |
== Note == |
||
<references/> |
<references/> |
||
| Riga 226: | Riga 162: | ||
==Voci correlate== |
==Voci correlate== |
||
*[[Infezione da Clostridioides difficile]] |
|||
*[[Terreno selettivo per Clostridioides difficile]] |
*[[Terreno selettivo per Clostridioides difficile]] |
||
*[[Infezioni correlate all'assistenza]] |
|||
==Altri progetti== |
==Altri progetti== |
||
Versione delle 14:34, 25 gen 2021
| Clostridioides difficile | |
|---|---|
 | |
| Classificazione scientifica | |
| Dominio | Prokaryota |
| Regno | Bacteria |
| Phylum | Firmicutes |
| Classe | Clostridia |
| Ordine | Clostridiales |
| Famiglia | Clostridiaceae |
| Genere | Clostridioides |
| Specie | C. difficile |
| Nomenclatura binomiale | |
| Clostridioides difficile Lawson & Rainey, 2016 | |
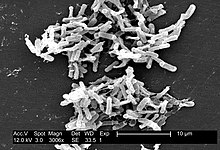
Clostridioides difficile, una volta conosciuto come Clostridium difficile[1] o Peptoclostridium difficile[2], è un batterio sporigeno appartenente alla famiglia Clostridiaceae. È un bastoncello, Gram +, anaerobio. Come altre specie affini, quando produce specifiche tossine, può essere patogeno per l'uomo.
Ampiamente diffuso nel suolo è presente anche nell’apparato gastrointestinale di vari animali: cani, gatti, cavalli, maiali, roditori. Il C.difficile fa parte della normale flora saprofita dell’intestino umano e colonizza tra il 3% e il 15% degli adulti sani. Può essere isolato nell’ 80% delle feci dei bambini fino a 1 anno di età, colonizzazione favorita dall’immaturità della flora batterica intestinale.
La specie C.difficile è definita da un grande e diversificato pan-genoma con livelli estremi di plasticità evolutiva che è stata modellata per lunghi periodi di tempo dal flusso genico e dalla ricombinazione, spesso tra lignaggi divergenti. Sono stati isolati centinaia di diversi ceppi del C.difficile, non tutti tossigeni. La continua evoluzione del C.difficile in risposta alle attività ambientali e antropiche ha portato alla rapida comparsa e alla diffusione in tutto il mondo di lignaggi clonali virulenti.
L'infezione da Closteridioides difficile è, in alcuni paesi, la causa più comune di infezioni nosocomiali e causerebbe circa 15000 morti l'anno negli USA e 3700 nella UE/EEAA.[3][4][5]
Cenni storici
Hall e O'Toole, in materiale fecale di neonati, nel 1935 individuarono il batterio: Bacillus difficilis, chiamato così per la difficoltà incontrata nell'isolamento e per l'estrema lentezza di crescita dello stesso sul terreno colturale. Nel 1970 fu rinominato come Clostridium difficile.[6][7][8][9] Mentre il primo caso di Colite pseudomembranosa era stato documentato da Finney nel 1893 in una autopsia di una donna, morta per una diarrea emorragica post-intervento chirurgico, che presentava pseudomembrane simil-difteriche nel colon.[10][11]
Nel 1968 su criceti, normalmente usati per lo studio del C.difficile, trattati con l'antibiotico lincomicina si osservò la comparsa di una grave colite pseudomembranosa sperimentale mortale.[11]
Nel 1970 animali trattati con antibiotici presentavano un'alta quantità nelle feci di tossina patogena prodotta dal C.difficile, individuando con la tossina la causa primaria delle manifestazioni cliniche della patologia della colite pseudomembranosa.[11] Nel 1980 furono individuate due distinte tossine (A e B) prodotte dal C.difficile.[12] Le tossine A e B sono trascritte su un locus di patogenicità che comprende cinque geni: due geni della tossina, tcdA (tossina A) e tcdB (tossina B), e tre geni regolatori, uno dei quali (tcdC) codifica un regolatore negativo della trascrizione della tossina.[13][14]
La prima sequenza genomica completa di un ceppo di C.difficile è stata pubblicata per la prima volta nel 2005 dal Sanger Institute nel Regno Unito. Si trattava del ceppo 630, un ceppo virulento e resistente ai farmaci isolato in Svizzera nel 1982.
Batteriologia

Troviamo spore di Clostridioides difficili nel terreno, negli ospedali e nelle case per anziani. La forma attiva dei batteri si trova solo nel tratto intestinale . Al microscopio, dopo la colorazione di Gram , sono bacilli allungati con un'estremità leggermente gonfia. Il germe stesso è gram-positivo . La sua cultura è ottimale in ambiente basico di agar a 37 ° C . Quando le condizioni si fanno difficili, i batteri producono spore resistenti all'essiccazione, a temperature estreme e a molti prodotti chimici e disinfettanti.[15][16].
Le spore sono metabolicamente dormienti e altamente resistenti alle procedure di disinfezione standard, consentendo loro di persistere per lunghi periodi nell'ambiente. Le spore ingerite da ospiti sensibili possono riattivarsi (o germogliare) in risposta a specifici acidi biliari nell'intestino tenue e tornare a uno stile di vita attivo per produrre tossine e causare malattie. Le spore sono altamente trasmissibili e responsabili della contaminazione degli ambienti Il Clostridioides difficile è un germe della flora digestiva commensale. Rilevato anche nelle feci degli infanti è spesso asintomatico per l'incapacità della tossina, nei ceppi tossigenici, di legarsi ai recettori degli enterociti ancora immaturi. Alcune sue mutazioni emergenti sono multi-resistenti (si sono adattate alla maggior parte degli antibiotici) e in caso di disturbo della flora digestiva provocato dalla somministrazione di antibiotici, possono svilupparsi in modo significativo. La grande variabilità dei diversi ceppi rende difficile la loro caratterizzazione molecolare. Quando nel 2003 l'analisi molecolare ha portato all'identificazione di un ceppo di C.difficile caratterizzato come gruppo BI mediante analisi di endonucleasi di restrizione, NAP1 (nordamericano a campo pulsato) mediante elettroforesi su gel a campo pulsato e ribotipo 027, il "nuovo" ceppo è è stato chiamato " C.difficile BI / NAP1 / 027 ". La diversa terminologia riflette le tecniche predominanti utilizzate per la tipizzazione epidemiologica.[17][18][19]
Del C.difficile esistono sia ceppi tossigenici (cioè produttori di tossine) che non tossigenici, ma solo le forme tossigeniche sono causa di malattia negli esseri umani.
I batteri producono citotossine polipeptidiche ad alta massa molecolare, A e B. Alcuni ceppi producono solo una delle tossine, altri producono entrambe. La tossina A provoca una reazione infiammatoria che coinvolge l'ipersecrezione di liquido e la necrosi emorragica attraverso l'attivazione del rilascio di citochine da parte dei neutrofili. La citotossina B depolimerizza l'actina, la principale proteina del citoscheletro, e quindi aiuta nella distruzione dei tessuti. L'azione combinata delle tossine provoca necrosi dell'epitelio superficiale ed edema (gonfiore fluidico) nelle zone dell'intestino colpite. La proliferazione del C.difficile è normalmente contrastata dalla normale microflora intestinale, che si ritiene inibisca l'attaccamento del batterio e delle sue tossine alle pareti intestinali. L'alterazione dell'equilibrio microbico intestinale con la terapia antibiotica e la maggiore esposizione al batterio in ambiente ospedaliero consente al C.difficile di colonizzare individui suscettibili. Inoltre, è stato dimostrato che le concentrazioni subinibitorie di antibiotici promuovono una maggiore produzione di tossine da parte di C.difficile .[20][21][22][23][24][25]
Ceppi
Il C. difficile è tassonomicamente distinto da molti altri ben noti clostridi, con una struttura di popolazione diversificata che comprende centinaia di ceppi diversi sparsi in almeno 6 cladi filogenetici. Del C.difficile esistono sia ceppi tossigenici (cioè produttori di tossine) che non tossigenici, ma solo le forme tossigeniche sono causa di malattia negli esseri umani. La patogenicità dipende infatti dalla presenza delle tossine A (TcdA, enterotossina) e B (TcdB, citotossina). Sono stati identificati oltre 400 diversi ceppi tossigenici.[26]
Tutti i ceppi tossigenici esprimono la tossina TcdB, con o senza la tossina TcdA. La tossina B ha infatti attività citotossica più potente della tossina A. Entrambe le tossine sopravvivono nell’ambiente acido dello stomaco e sono in grado di legarsi alla superficie delle cellule epiteliali delle mucose intestinali dove vengono internalizzate e dove catalizzano la glicosilazione di alcune proteine citoplasmatiche, con successiva morte cellulare. Una terza tossina di cui non è chiara la patogenicità, la tossina binaria (CDT), è prodotta da alcuni ceppi di C.difficile. Questa tossina ha dimostrato di incrementare la virulenza di C.difficile attraverso l’adenosina difosfato-ribosilazione di actina, inducendo la formazione di protrusioni di microtubuli nelle cellule facilitando l’adesione del batterio sulla superficie cellulare. La tossina CDT può essere presente anche nei ceppi dove le tossine A e B sono assenti, riscontrati nei pazienti asintomatici.[27]
Esistono ceppi che producono quantità massive di tossine comparsi sin dal 2000. In particolare, C.difficile PCR-ribotipo 027 (BI/NAP1/027) è stato associato ad un aumento della gravità dei casi, delle ricorrenze e della mortalità. Il ceppo produce entrambe le tossine in gran quantità a causa di una mutazione (delezione) del gene regolatore tdcC e possiede, analogamente ad altre specie di Clostridi, la tossina binaria CDT. Oltre allo 027, anche altri ribotipi epidemici considerati ipervirulenti sono emersi, alcuni a diffusione internazionale, come lo 078 e lo 018.
Una varietà di approcci di tipizzazione molecolare sono stati sviluppati per studiare il C.difficile e CDI, tra cui: multilocus sequence typing (MLST), multilocus variable-number tandem-repeat analysis (MLVA), surface layer protein A gene sequence typing, amplificated fragment length polymorphism, elettroforesi su gel a campo pulsato (PFGE), analisi dell'endonucleasi di restrizione (REA), tossinotipizzazione (basata sui dati di sequenza delle tossine A e B) e ribotipizzazione PCR.[28] Quando nel 2005, l'analisi molecolare ha portato all'identificazione di un tipo di ceppo di C.difficile caratterizzato come gruppo BI mediante analisi di endonucleasi di restrizione , NAP1 di tipo a campo pulsato nordamericano mediante elettroforesi su gel a campo pulsato e ribotipo 027 mediante ribotipizzazione PCR multiplex è stato chiamato C.difficile BI/NAP1/027. La diversa terminologia riflette le tecniche predominanti utilizzate per la tipizzazione epidemiologica, anche se la ribotipizzazione PCR è quella più ampiamente accettata in Europa dove l'Anaerobe Reference Laboratory di Cardiff (UK) tiene la collezione dei ceppi e assegna la numerazione dei ribotipi PCR. In Nord America, PFGE è il metodo di digitazione preferito.[29]
Nel Nord America, a partire dal 2016, il ceppo NAP1 è stato sostituito da nuovi ceppi in alcune aree della British Columbia. Questi nuovi ceppi includono NAP2, NAP4, NAP9 e NAP11 e alcuni ceppi che non hanno una designazione NAP. La frequenza di questi nuovi ceppi è aumentata dal 2008 al 2013 in una regione studiata, sostituendo i batteri NAP1 originariamente più comuni e riconoscibili.
In generale, la maggior parte dei metodi di identificazione sono congruenti anche se non si ha una corrispondenza biunivoca tra le diverse tipizzazioni e solo le tecniche MLVA e REA consentono una sufficiente discriminazione tra diversi focolai.[30][31]
I principali database dove sono registrate le caratterizzazioni genetiche dei diversi ceppi di C.difficile sono: Enterobase[32] Public databases for molecular typing and microbial genome diversity (PubMLST).[33]
Alcuni ceppi, ad esempio RT017 RT078 RT027, possono vivere con basse concentrazioni di zucchero trealosio ; questi ceppi sono diventati più comuni dopo che il trealosio è stato introdotto come additivo alimentare nei primi anni 2000, aumentando così l'assunzione cibi addizionati di trealosio.[34][35]
Dal 2005, la morbilità e la mortalità derivanti da C. difficile sono aumentate costantemente in tutto il mondo in funzione dell'insorgenza di ceppi definiti ipervirulenti (in particolare il ribotipo 027). I meccanismi attualmente proposti che distinguono i ceppi ipervirulenti dai ceppi precedenti (meno virulenti) sono oggetto di controversia. Sono stati proposti: l'aumentato tasso di sporulazione, l'aumentata produzione di tossine , la capacità di sopravanzare i ceppi enedemici nell'intestino dell'ospite ma come questo patogeno sia diventato la principale causa di infezioni nosocomiali rimane sconosciuto e la classificazione di "ceppo ipervirulento" resta arbitraria.[36][37]Molti ceppi di C.difficile sono stati rilevati negli animali ed in alimenti ricavati da animali facendo emergere anche l'ipotesi che molte trasmissioni del C.difficile nell'uomo siano correlate con l'alimentazione di carne o pesce o interspecie.[38][39][40][41][42][43]
Genoma
Il pan-genoma del C.difficile è composto da un genoma centrale (quei geni presenti in tutti gli isolati) e da un genoma accessorio o adattativo (geni assenti da uno o più ceppi o unici per un particolare ceppo).
Una grande percentuale (11%) del genoma è costituita da elementi genetici mobili, principalmente sotto forma di trasposoni coniugativi.
L'analisi ha identificato quattro distinti cluster statisticamente supportati comprendenti un clade ipervirulento, una tossina A - B +clade e due cladi con isolati umani e animali. Le differenze genetiche tra i cladi hanno rivelato diverse isole genetiche relative alla virulenza e all'adattamento di nicchia, tra cui resistenza agli antibiotici, motilità, adesione e metabolismo enterico. Solo il 19,7% dei geni era condiviso da tutti i ceppi,
Il genoma di C.difficile è ampio e geneticamente diverso, mostrando livelli notevoli di plasticità e livelli bassissimi di conservazione tra i ceppi.
C. difficile possiede un genoma "aperto" con livelli estremi di plasticità, con accesso e scambio frequente con più ambienti ospiti e pool genetici batterici. Di conseguenza, poiché vengono sequenziati più ceppi di C.difficile da linee divergenti e diverse fonti animali e ambientali, questa stima aumenterà probabilmente.
La prima sequenza genomica completa di un ceppo di C.difficile è stata pubblicata per la prima volta nel 2005 dal Sanger Institute nel Regno Unito. Si trattava del ceppo 630, un ceppo virulento e resistente ai farmaci isolato in Svizzera nel 1982. Gli scienziati del Sanger Institute hanno sequenziato genomi di circa 30 isolati di C.difficile utilizzando tecnologie di sequenziamento di nuova generazione da 454 Life Sciences e Illumina .
I ricercatori della McGill University di Montreal hanno sequenziato il genoma del ceppo di C.difficile del Quebec altamente virulento nel 2005 utilizzando una tecnologia di sequenziamento ad altissima velocità. I test hanno comportato l'esecuzione di 400.000 reazioni di sequenziamento parallelo del DNA del genoma del batterio, che era stato frammentato per il sequenziamento. Queste sequenze sono state assemblate computazionalmente per formare una sequenza genomica completa.
Nel 2012, gli scienziati dell'Università di Oxford hanno sequenziato i genomi di C.difficile da 486 casi verificatisi in quattro anni nell'Oxfordshire utilizzando le tecnologie di sequenziamento di nuova generazione.
Nel 2006, Sebaihia e colleghi hanno descritto il primo genoma chiuso completamente sequenziato e annotato di C.difficile (ceppo 630; RT012).[21] Questo ceppo virulento, altamente trasmissibile e multiresistente di C.difficile è stato originariamente isolato nel 1982 da un paziente con PMC a Zurigo, Svizzera. Il sequenziamento e l'annotazione del ceppo 630 hanno rivelato un grande cromosoma circolare di 4.290.252 bp (4,3 Mb), 3.776 sequenze putative di codifica delle proteine (CDS) e un contenuto di GC del 29,06% . È stato anche identificato un plasmide circolare (pCD630) di 7.881 bp contenente 11 CDS .[21] Cinque anni dopo, Monot e colleghi[44] hanno riannotato il genoma del ceppo 630 utilizzando un approccio trascrittomico e proteomico combinato per aggiornare le funzioni putative di> 500 geni precedentemente putativi o sconosciuti. Da allora, molti altri genomi di dimensioni comprese tra 4,1 e 4,3 Mbp sono stati completamente sequenziati e annotati: CD37 (RT009; isolato negli Stati Uniti nel 1980), M68 (RT017; isolato in Irlanda nel 2006), CF5 (RT017; isolato in Belgio nel 1995), M120 (RT078; isolato nel Regno Unito nel 2007), G46 (RT027; isolato nel Regno Unito nel 2006), R20291(RT027; isolato nel Regno Unito nel 2006), 196 (RT027; isolato in Francia nel 1985), 2007855 (RT027; isolato negli Stati Uniti nel 2007) e BI1 (RT027; isolato negli Stati Uniti nel 1988).[45][46][47] I genomi di riferimento come quelli dei ceppi 630 e M120 svolgono un ruolo importante nella pipeline di analisi dei dati NGS. Comprendono una sequenza inequivocabile e contigua di nucleotidi noti che attraversano l'intero cromosoma e plasmidi (se presenti), fornendo quindi un riferimento di altissima qualità per la mappatura dei genomi in bozza.
In sintesi, gli studi MLST su diverse raccolte di ceppi di C. difficile suggeriscono che ci siano cinque lignaggi o cladi che possono generalmente essere suddivisi per i più importanti ribotipi PCR; 027, 017, 023, 078 e un ampio gruppo comprendente il resto dei ribotipi PCR che rappresenta un lignaggio interessante altamente divergente.
Epigenoma
C. difficile ha un epigenoma altamente diversificato, con 17 motivi di metilazione di alta qualità segnalati finora, la maggior parte relativi al tipo 6mA. È stato dimostrato che la metilazione in uno di questi motivi, CAAAA A, altamente conservato, influisce sulla sporulazione, un passaggio chiave nella trasmissione della malattia da C.difficile , nonché sulla lunghezza delle cellule, sulla formazione di biofilm e sulla colonizzazione dell'ospite.[48]
Batteriofagi
Un altro componente importante del mobiloma di C.difficile sono i batteriofagi (fagi). I fagi si sono evoluti con il C.difficile per periodi di tempo molto lunghi e l'infezione dei fagi è una parte intrinseca della storia naturale e della biologia del C.difficile . l'acquisizione di fagi e la loro perdita dal genoma di C.difficile sono eventi genetici significativi che hanno avuto un impatto sull'evoluzione dell'ospite.[49]
Almeno otto batteriofagi, principalmente temperati cioè lisogeni, sono stati isolati dal C.difficile , con dimensioni del genoma comprese tra circa 30 e circa 60 kb. Sia i ceppi di C.difficile di origine ambientale che quelli di origine clinica sono portatori di un insieme diversificato e prevalente di profagi .
Rilevanza clinica
Solo i ceppi di Clostridioides difficile che producono tossine sono di rilevanza clinica.
La rapida evoluzione della resistenza agli antibiotici nel C.difficile[50] e i conseguenti effetti sulla prevenzione e il trattamento delle infezioni da C. difficile sono motivo di preoccupazione per la salute pubblica. [51]
I fattori di virulenza del C.difficile sono: enterotossina, spore e ialuronidasi. Questo bacillo si trova normalmente nel microbiota umano per cui, se si utilizzano per lungo tempo antibiotici, questi possono distruggere anche quei batteri che tengono confinato il C.difficile il quale può prendere il sopravvento e provocare crampi addominali e malattie varie (colite pseudomembranosa). Queste malattie hanno un decorso benigno e autolimitanti tranne nel caso in cui ci sono complicanze che possono compromettere la parete intestinale con un possibile passaggio in circolo del bacillo, con conseguente sepsi e quindi morte dell'individuo.
L'isolamento può avvenire tramite la coprocoltura in terreni selettivi per C.difficile. L'enterotossina può essere saggiata con il metodo ELISA, mentre la citotossina con rivelazione di tossicità in colture cellulari.
Note
- ^ Paul A. Lawson, Diane M. Citron e Kerin L. Tyrrell, Reclassification of Clostridium difficile as Clostridioides difficile (Hall and O'Toole 1935) Prévot 1938, in Anaerobe, vol. 40, 2016-08, pp. 95–99, DOI:10.1016/j.anaerobe.2016.06.008. URL consultato il 22 gennaio 2021.
- ^ Peptoclostridium difficile - Genome - Assembly - NCBI, su www.ncbi.nlm.nih.gov. URL consultato il 23 gennaio 2021.
- ^ Evelyn Balsells, Ting Shi e Callum Leese, Global burden of Clostridium difficile infections: a systematic review and meta-analysis, in Journal of Global Health, vol. 9, n. 1, DOI:10.7189/jogh.09.010407. URL consultato il 23 gennaio 2021.
- ^ (EN) CDC, Nearly half a million Americans suffer from C. difficile infections in, su Centers for Disease Control and Prevention, 31 dicembre 2018. URL consultato il 23 gennaio 2021.
- ^ (EN) Clostridium difficile infections - Facts and surveillance, su European Centre for Disease Prevention and Control. URL consultato il 23 gennaio 2021.
- ^ Hall, Ivan C. e O'Toole. Elizabeth, Intestinal flora in newborn infants with a description of a new pathogenic anaerobe, Bacillus difficilis, in American Journal of Diseases of Children, vol. 49, n. 2, 1935, pp. 390–402.
- ^ X. Chen, K. Katchar; JD. Goldsmith; N. Nanthakumar; A. Cheknis; DN. Gerding; CP. Kelly, A mouse model of Clostridium difficile-associated disease., in Gastroenterology, vol. 135, n. 6, dicembre 2008, pp. 1984-92, DOI:10.1053/j.gastro.2008.09.002, PMID 18848941.
- ^ CP. Kelly, JT. LaMont, Clostridium difficile--more difficult than ever., in N Engl J Med, vol. 359, n. 18, ottobre 2008, pp. 1932-40, DOI:10.1056/NEJMra0707500, PMID 18971494.
- ^ MY. Hu, K. Katchar; L. Kyne; S. Maroo; S. Tummala; V. Dreisbach; H. Xu; DA. Leffler; CP. Kelly, Prospective derivation and validation of a clinical prediction rule for recurrent Clostridium difficile infection., in Gastroenterology, vol. 136, n. 4, aprile 2009, pp. 1206-14, DOI:10.1053/j.gastro.2008.12.038, PMID 19162027.
- ^ JT. Lamont, Theodore E. Woodward Award. How bacterial enterotoxins work: insights from in vivo studies., in Trans Am Clin Climatol Assoc, vol. 113, 2002, pp. 167-80; discussion 180-1, PMID 12053708.
- ^ a b c Stephen H. Gillespie e Peter M. Hawkey, Principles and Practice of Clinical Bacteriology, John Wiley and Sons, 1º luglio 2006, pp. 557–, ISBN 978-0-470-03532-0.
- ^ I. Brook, Isolation of toxin producing Clostridium difficile from two children with oxacillin- and dicloxacillin-associated diarrhea., in Pediatrics, vol. 65, n. 6, giugno 1980, pp. 1154-6, PMID 7375240.
- ^ T. Chopra, GJ. Alangaden; P. Chandrasekar, Clostridium difficile infection in cancer patients and hematopoietic stem cell transplant recipients., in Expert Rev Anti Infect Ther, vol. 8, n. 10, ottobre 2010, pp. 1113-9, DOI:10.1586/eri.10.95, PMID 20954878.
- ^ Patrizia Spigaglia e Paola Mastrantonio, Molecular Analysis of the Pathogenicity Locus and Polymorphism in the Putative Negative Regulator of Toxin Production (TcdC) among Clostridium difficile Clinical Isolates, in Journal of Clinical Microbiology, vol. 40, n. 9, 2002-9, pp. 3470–3475, DOI:10.1128/JCM.40.9.3470-3475.2002. URL consultato il 22 gennaio 2021.
- ^ Dale N. Gerding, Carlene A. Muto e Robert C. Owens, Measures to control and prevent Clostridium difficile infection, in Clinical Infectious Diseases: An Official Publication of the Infectious Diseases Society of America, 46 Suppl 1, 15 gennaio 2008, pp. S43–49, DOI:10.1086/521861. URL consultato il 22 gennaio 2021.
- ^ William A. Rutala, Maria F. Gergen e David J. Weber, Efficacy of different cleaning and disinfection methods against Clostridium difficile spores: importance of physical removal versus sporicidal inactivation, in Infection Control and Hospital Epidemiology, vol. 33, n. 12, 2012-12, pp. 1255–1258, DOI:10.1086/668434. URL consultato il 22 gennaio 2021.
- ^ Spigaglia, P., Barbanti, F., Mastrantonio, P., European Study Group on Clostridium difficile (ESGCD), Ackermann, G., Balmelli, C., … & Drudy, D. (2011). Multidrug resistance in European Clostridium difficile clinical isolates. Journal of Antimicrobial Chemotherapy, 66(10), 2227-2234.
- ^ Kuijper, E. J., van den Berg, R. J., Debast, S., Visser, C. E., Veenendaal, D., Troelstra, A., … & Notermans, D. W. (2006). Clostridium difficile ribotype 027, toxinotype III, the Netherlands. Emerging infectious diseases, 12(5), 827.
- ^ John G. Bartlett, Narrative Review: The New Epidemic of Clostridium difficile–Associated Enteric Disease, in Annals of Internal Medicine, vol. 145, n. 10, 21 novembre 2006, pp. 758–764, DOI:10.7326/0003-4819-145-10-200611210-00008. URL consultato il 21 gennaio 2021.
- ^ Daniel R. Knight, Briony Elliott e Barbara J. Chang, Diversity and Evolution in the Genome of Clostridium difficile, in Clinical Microbiology Reviews, vol. 28, n. 3, 2015-7, pp. 721–741, DOI:10.1128/CMR.00127-14. URL consultato il 21 gennaio 2021.
- ^ a b c Mohammed Sebaihia, Brendan W. Wren e Peter Mullany, The multidrug-resistant human pathogen Clostridium difficile has a highly mobile, mosaic genome, in Nature Genetics, vol. 38, n. 7, 2006-07, pp. 779–786, DOI:10.1038/ng1830. URL consultato il 21 gennaio 2021.
- ^ Donald C. Vinh, Strains and toxins of Clostridium, in CMAJ : Canadian Medical Association Journal, vol. 172, n. 3, 1º febbraio 2005, pp. 312–313, DOI:10.1503/cmaj.1041642. URL consultato il 21 gennaio 2021.
- ^ L. Clifford McDonald, George E. Killgore e Angela Thompson, An Epidemic, Toxin Gene–Variant Strain of Clostridium difficile, in New England Journal of Medicine, vol. 353, n. 23, 8 dicembre 2005, pp. 2433–2441, DOI:10.1056/NEJMoa051590. URL consultato il 21 gennaio 2021.
- ^ (EN) Rawish Fatima e Muhammad Aziz, The Hypervirulent Strain of Clostridium Difficile: NAP1/B1/027 - A Brief Overview, in Cureus, vol. 11, n. 1, 29 gennaio 2019, DOI:10.7759/cureus.3977. URL consultato il 21 gennaio 2021.
- ^ Gayatri Vedantam, Andrew Clark e Michele Chu, Clostridium difficile infection, in Gut Microbes, vol. 3, n. 2, 1º marzo 2012, pp. 121–134, DOI:10.4161/gmic.19399. URL consultato il 21 gennaio 2021.
- ^ (EN) Kate E. Dingle, David Griffiths e Xavier Didelot, Clinical Clostridium difficile: Clonality and Pathogenicity Locus Diversity, in PLOS ONE, vol. 6, n. 5, 19-mag-2011, pp. e19993, DOI:10.1371/journal.pone.0019993. URL consultato il 23 gennaio 2021.
- ^ (EN) C. Eckert, A. Emirian e A. Le Monnier, Prevalence and pathogenicity of binary toxin–positive Clostridium difficile strains that do not produce toxins A and B, in New Microbes and New Infections, vol. 3, 2015-01, pp. 12–17, DOI:10.1016/j.nmni.2014.10.003. URL consultato il 23 gennaio 2021.
- ^ European Centre for Disease Prevention and Control., Laboratory procedures for diagnosis and typing of human Clostridium difficile infection., Publications Office, 2018, DOI:10.2900/04291. URL consultato il 23 gennaio 2021.
- ^ The Continually Evolving Clostridium difficile Species, su Medscape. URL consultato il 23 gennaio 2021.
- ^ George Killgore, Angela Thompson e Stuart Johnson, Comparison of seven techniques for typing international epidemic strains of Clostridium difficile: restriction endonuclease analysis, pulsed-field gel electrophoresis, PCR-ribotyping, multilocus sequence typing, multilocus variable-number tandem-repeat analysis, amplified fragment length polymorphism, and surface layer protein A gene sequence typing, in Journal of Clinical Microbiology, vol. 46, n. 2, 2008-02, pp. 431–437, DOI:10.1128/JCM.01484-07. URL consultato il 24 gennaio 2021.
- ^ Fred C. Tenover, Thomas Åkerlund e Dale N. Gerding, Comparison of Strain Typing Results for Clostridium difficile Isolates from North America▿, in Journal of Clinical Microbiology, vol. 49, n. 5, 2011-5, pp. 1831–1837, DOI:10.1128/JCM.02446-10. URL consultato il 24 gennaio 2021.
- ^ EnteroBase, su enterobase.warwick.ac.uk. URL consultato il 24 gennaio 2021.
- ^ (EN) Clostridioides difficile, su PubMLST. URL consultato il 24 gennaio 2021.
- ^ J. Collins, C. Robinson e H. Danhof, Dietary trehalose enhances virulence of epidemic Clostridium difficile, in Nature, vol. 553, n. 7688, 18 gennaio 2018, pp. 291–294, DOI:10.1038/nature25178. URL consultato il 24 gennaio 2021.
- ^ (EN) David W. Eyre, Xavier Didelot e Anthony M. Buckley, Clostridium difficile trehalose metabolism variants are common and not associated with adverse patient outcomes when variably present in the same lineage, in EBioMedicine, vol. 43, 1º maggio 2019, pp. 347–355, DOI:10.1016/j.ebiom.2019.04.038. URL consultato il 24 gennaio 2021.
- ^ (EN) Michelle Merrigan, Anilrudh Venugopal e Michael Mallozzi, Human Hypervirulent Clostridium difficile Strains Exhibit Increased Sporulation as Well as Robust Toxin Production, in Journal of Bacteriology, vol. 192, n. 19, 1º ottobre 2010, pp. 4904–4911, DOI:10.1128/JB.00445-10. URL consultato il 24 gennaio 2021.
- ^ Laith Yakob, Thomas V. Riley e David L. Paterson, Mechanisms of hypervirulent Clostridium difficile ribotype 027 displacement of endemic strains: an epidemiological model, in Scientific Reports, vol. 5, 28 luglio 2015, DOI:10.1038/srep12666. URL consultato il 24 gennaio 2021.
- ^ (EN) Alexander Rodriguez-Palacios, Kevin Q Mo e Bhavan U. Shah, Global and Historical Distribution of Clostridioides difficile in the Human Diet (1981-2019): Systematic Review and Meta-Analysis of 21886 Samples Reveal Sources of Heterogeneity, High-Risk Foods, and Unexpected Higher Prevalence Towards the Tropic, Infectious Diseases (except HIV/AIDS), 22 novembre 2019, DOI:10.1101/19012450. URL consultato il 24 gennaio 2021.
- ^ Dallas G. Hoover e Alexander Rodriguez-Palacios, Transmission of Clostridium difficile in foods, in Infectious Disease Clinics of North America, vol. 27, n. 3, 2013-09, pp. 675–685, DOI:10.1016/j.idc.2013.05.004. URL consultato il 23 gennaio 2021.
- ^ (EN) K. Keel, J. S. Brazier e K. W. Post, Prevalence of PCR Ribotypes among Clostridium difficile Isolates from Pigs, Calves, and Other Species, in Journal of Clinical Microbiology, vol. 45, n. 6, 1º giugno 2007, pp. 1963–1964, DOI:10.1128/JCM.00224-07. URL consultato il 24 gennaio 2021.
- ^ Alexander W. W. Brown e Robert B. Wilson, Clostridium difficile colitis and zoonotic origins-a narrative review, in Gastroenterology Report, vol. 6, n. 3, 2018-08, pp. 157–166, DOI:10.1093/gastro/goy016. URL consultato il 23 gennaio 2021.
- ^ Alan M. Mc Govern, Niki F. Foster e Lynette A. Pereira, Human Clostridium difficile infection caused by a livestock-associated PCR ribotype 237 strain in Western Australia, in JMM case reports, vol. 3, n. 4, 2016-08, pp. e005062, DOI:10.1099/jmmcr.0.005062. URL consultato il 23 gennaio 2021.
- ^ (EN) Luis G Arroyo, Stephen A Kruth e Barbara M Willey, PCR ribotyping of Clostridium difficile isolates originating from human and animal sources, in Journal of Medical Microbiology, vol. 54, n. 2, 1º febbraio 2005, DOI:10.1099/jmm.0.45805-0#tab2. URL consultato il 23 gennaio 2021.
- ^ Marc Monot, Caroline Boursaux-Eude e Marie Thibonnier, Reannotation of the genome sequence of Clostridium difficile strain 630, in Journal of Medical Microbiology, vol. 60, Pt 8, 2011-08, pp. 1193–1199, DOI:10.1099/jmm.0.030452-0. URL consultato il 24 gennaio 2021.
- ^ Tom Gaulton, Raju Misra e Graham Rose, Complete Genome Sequence of the Hypervirulent Bacterium Clostridium difficile Strain G46, Ribotype 027, in Genome Announcements, vol. 3, n. 2, 26 marzo 2015, DOI:10.1128/genomeA.00073-15. URL consultato il 24 gennaio 2021.
- ^ Michael S. M. Brouwer, Elaine Allan e Peter Mullany, Draft genome sequence of the nontoxigenic Clostridium difficile strain CD37, in Journal of Bacteriology, vol. 194, n. 8, 2012-04, pp. 2125–2126, DOI:10.1128/JB.00122-12. URL consultato il 24 gennaio 2021.
- ^ Miao He, Mohammed Sebaihia e Trevor D. Lawley, Evolutionary dynamics of Clostridium difficile over short and long time scales, in Proceedings of the National Academy of Sciences of the United States of America, vol. 107, n. 16, 20 aprile 2010, pp. 7527–7532, DOI:10.1073/pnas.0914322107. URL consultato il 24 gennaio 2021.
- ^ Pedro H. Oliveira, John W. Ribis e Elizabeth M. Garrett, Epigenomic characterization of Clostridioides difficile finds a conserved DNA methyltransferase that mediates sporulation and pathogenesis, in Nature microbiology, vol. 5, n. 1, 2020-1, pp. 166–180, DOI:10.1038/s41564-019-0613-4. URL consultato il 22 gennaio 2021.
- ^ Katherine R. Hargreaves e Martha R. J. Clokie, Clostridium difficile phages: still difficult?, in Frontiers in Microbiology, vol. 5, 28 aprile 2014, DOI:10.3389/fmicb.2014.00184. URL consultato il 22 gennaio 2021.
- ^ Saeed S. Banawas, Clostridium difficile Infections: A Global Overview of Drug Sensitivity and Resistance Mechanisms, in BioMed Research International, vol. 2018, 21 febbraio 2018, DOI:10.1155/2018/8414257. URL consultato il 24 gennaio 2021.
- ^ Patrizia Spigaglia, Paola Mastrantonio e Fabrizio Barbanti, Antibiotic Resistances of Clostridium difficile, in Advances in Experimental Medicine and Biology, vol. 1050, 2018, pp. 137–159, DOI:10.1007/978-3-319-72799-8_9. URL consultato il 24 gennaio 2021.
Bibliografia
- Giampiero Carosi, Carosi - Pauluzzi e Sergio Pauluzzi, Malattie infettive, PICCIN, 2007, pp. 233–, ISBN 978-88-299-1844-7.
- (EN) Tracy Dale Wilkins, Clostridium difficile, Springer, 2000, ISBN 978-3-540-67291-3.
- (EN) Patrice Boquet e Emmanuel Lemichez, Bacterial virulence factors and Rho GTPases, Springer, 2005, pp. 117–, ISBN 978-3-540-23865-2.
- (EN) Ferruccio Mandler, Catalogo ragionato dei presidi diagnostici per la microbiologia clinica, PICCIN, 1999, pp. 62–, ISBN 978-88-299-1442-5.
- (EN) Stephen H. Gillespie e Peter M. Hawkey, Principles and Practice of Clinical Bacteriology, John Wiley and Sons, 1º luglio 2006, pp. 557–, ISBN 978-0-470-03532-0.
- (EN) James G. Fox, The Mouse in Biomedical Research: Diseases, Academic Press, 2007, pp. 357–, ISBN 978-0-12-369456-0.
- (EN) ICDDR, B, Diarrhoeal Diseases Research, ICDDR, B, settembre 1992, pp. 156–.
- (EN) Allison Tannis, Probiotic Rescue: How You Can Use Probiotics to Fight Cholesterol, Cancer Superbugs, Digestive Complaints and More, John Wiley and Sons, 3 aprile 2008, pp. 182–, ISBN 978-0-470-15475-5.
Riviste
- CH. Choi, SA. Jung; BI. Lee; KM. Lee; JS. Kim; DS. Han, [Diagnostic guideline of ulcerative colitis]., in Korean J Gastroenterol, vol. 53, n. 3, marzo 2009, pp. 145-60, PMID 19835217.
- E. van Nood, P. Speelman; EJ. Kuijper; JJ. Keller, Struggling with recurrent Clostridium difficile infections: is donor faeces the solution?, in Euro Surveill, vol. 14, n. 34, 2009, PMID 19712646.
- E. Palencia-Herrejón, B. Sánchez; I. Escobar; ML. Gómez-Lus, [Proton pump inhibitors and infection risk]., in Rev Esp Quimioter, vol. 24, n. 1, marzo 2011, pp. 4-12, PMID 21412664.
- AC. de Oliveira, QS. Damasceno, [Surfaces of the hospital environment as possible deposits of resistant bacteria: a review]., in Rev Esc Enferm USP, vol. 44, n. 4, dicembre 2010, pp. 1118-23, PMID 21337799.
- J. Freeman, MP. Bauer; SD. Baines; J. Corver; WN. Fawley; B. Goorhuis; EJ. Kuijper; MH. Wilcox, The changing epidemiology of Clostridium difficile infections., in Clin Microbiol Rev, vol. 23, n. 3, luglio 2010, pp. 529-49, DOI:10.1128/CMR.00082-09, PMID 20610822.
- D. Shah, MD. Dang; R. Hasbun; HL. Koo; ZD. Jiang; HL. DuPont; KW. Garey, Clostridium difficile infection: update on emerging antibiotic treatment options and antibiotic resistance., in Expert Rev Anti Infect Ther, vol. 8, n. 5, maggio 2010, pp. 555-64, DOI:10.1586/eri.10.28, PMID 20455684.
- C. Vaishnavi, Clinical spectrum pathogenesis of Clostridium difficile associated diseases., in Indian J Med Res, vol. 131, aprile 2010, pp. 487-99, PMID 20424299.
- H. Taslim, Clostridium difficile infection in the elderly., in Acta Med Indones, vol. 41, n. 3, luglio 2009, pp. 148-51, PMID 19752488.
- M. Bixquert Jiménez, Treatment of irritable bowel syndrome with probiotics. An etiopathogenic approach at last?, in Rev Esp Enferm Dig, vol. 101, n. 8, agosto 2009, pp. 553-64, PMID 19785495.
- C. Eckert, F. Barbut, [Clostridium-difficile-associated infections]., in Med Sci (Paris), vol. 26, n. 2, febbraio 2010, pp. 153-8, DOI:10.1051/medsci/2010262153, PMID 20188046.
Veterinaria
- Wilfried R. Kraft e Ulrich M. Dürr, Trattato di medicina e chirurgia del gatto, Elsevier srl, 2001, pp. 199–, ISBN 978-88-214-2525-7.
- (EN) Norman Edward Robinson e Kim A. Sprayberry, Current therapy in equine medicine, Elsevier Health Sciences, 2009, pp. 158–, ISBN 978-1-4160-5475-7.
Voci correlate
- Infezione da Clostridioides difficile
- Terreno selettivo per Clostridioides difficile
- Infezioni correlate all'assistenza
Altri progetti
 Wikimedia Commons contiene immagini o altri file su Clostridioides difficile
Wikimedia Commons contiene immagini o altri file su Clostridioides difficile Wikispecies contiene informazioni su Clostridioides difficile
Wikispecies contiene informazioni su Clostridioides difficile
