Batteriofago

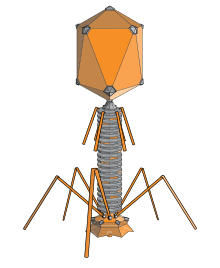
Un batteriòfago[2] o fago è un virus che infetta esclusivamente i batteri e sfrutta il loro apparato biosintetico per effettuare la replicazione virale. L'infezione virale del batterio ne causa la morte per lisi, ossia mediante rottura della membrana plasmatica dovuta all'accumulo della progenie nel citoplasma.
I batteriofagi sono composti da proteine che incapsulano un genoma a DNA o RNA; esso può avere strutture semplici o elaborate. I loro genomi possono codificare fino a quattro geni (ad esempio MS2) e fino a centinaia di geni. I fagi si replicano all'interno del batterio dopo l'iniezione del loro genoma nel citoplasma. Inoltre, i batteriofagi sono tra le entità più comuni e diverse nella biosfera; essi sono presenti ovunque si trovino batteri. Si stima che ci siano più di 1031 batteriofagi sul pianeta, più di ogni altro organismo terrestre, compresi i batteri, combinati.[3]
Una delle maggiori fonti naturali di fagi e altri virus è l'acqua di mare, dove sono stati trovati fino a 9x108 virioni per millilitro in tappeti microbici in superficie,[4] inoltre fino al 70% dei batteri marini può essere infetto da fagi.[5]
I fagi sono stati usati dalla fine del XX secolo come alternativa agli antibiotici sia nell'ex Unione Sovietica sia nell'Europa centrale, nonché in Francia.[6][7] Sono visti come una possibile terapia contro i ceppi multi-farmaco-resistenti di molti batteri (vedi terapia fagica).[8] D'altra parte, i fagi di Inoviridae hanno dimostrato di complicare i biofilm coinvolti nella polmonite e la fibrosi cistica e di proteggere i batteri dai farmaci destinati a debellare le malattie, promuovendo così un'infezione persistente.[9]
Struttura[modifica | modifica wikitesto]
I batteriofagi più complessi, come quelli della serie T (ad esempio il fago T2), hanno forma di spillo. La testa costituisce il capside e racchiude l'acido nucleico; al di sotto di essa vi è una sorta di collare, cui è attaccata una coda, la quale termina all'estremità basale con 5-6 filamenti detti fibre caudali, essi sono coperti dalla proteina virale, ovvero una sostanza che gli permette di riconoscere le cellule infette per poi iniettarci dentro il suo DNA o RNA.[10]
Replicazione[modifica | modifica wikitesto]

Il batteriofago attacca il batterio fissando le fibre caudali su un punto preciso della sua membrana cellulare. Con un meccanismo di contrazione inietta al suo interno il proprio acido nucleico, mentre l'involucro proteico rimane all'esterno.
Una volta penetrato, il genoma fagico può seguire due vie:[11]
- nel ciclo litico utilizzerà l'apparato di replicazione dell'ospite per produrre nuove particelle fagiche fino al raggiungimento del volume di scoppio, momento in cui la cellula si disgregherà per lisi;
- nel ciclo lisogeno il genoma fagico si integrerà in un punto specifico (attλ, nel caso del fago λ) del cromosoma batterico. In questo stato integrato il fago viene chiamato profago e, ogni qual volta il cromosoma batterico si replica, verrà replicato anche il genoma fagico. Il batterio che contiene il profago viene detto "lisogeno". Lo stato di profago viene mantenuto da una specifica proteina prodotta dal fago; l'allontanamento di questo repressore induce il passaggio verso il ciclo litico.
Classificazione[modifica | modifica wikitesto]
I batteriofagi si trovano abbondantemente nella biosfera, con diversi genomi e stili di vita. I fagi sono classificati dall'International Committee on Taxonomy of Viruses (ICTV) in base alla morfologia e all'acido nucleico.[12][13]
| Order | Famiglia | Morfologia | Acido nucleico | Esempi |
|---|---|---|---|---|
| Belfryvirales | Turriviridae | Avvolto, isometrico | Lineare dsDNA | |
| Caudovirales | Ackermannviridae | Coda contrattile non avvolta | Lineare dsDNA | |
| Myoviridae | Coda contrattile non avvolta | Lineare dsDNA | T4, Mu, P1, P2 | |
| Siphoviridae | Coda non avvolgente, non contrattile (lunga) | Lineare dsDNA | λ, T5, HK97, N15 | |
| Podoviridae | Coda non avvolgente, non contrattile (corta) | Lineare dsDNA | T7, T3, Φ29, P22 | |
| Halopanivirales | Sphaerolipoviridae | Avvolto, isometrico | Lineare dsDNA | |
| Haloruvirales | Pleolipoviridae | Avvolto, pleomorfo | Circolare ssDNA, circolare dsDNA, o lineare dsDNA | |
| Kalamavirales | Tectiviridae | Non avvolto, isometrico | Lineare dsDNA | |
| Levivirales | Leviviridae | Non avvolto, isometrico | Lineare ssRNA | MS2, Qβ |
| Ligamenvirales | Lipothrixviridae | Avvolto, a forma di bastoncino | Lineare dsDNA | Acidianus filamentous virus 1 |
| Rudiviridae | Non avvolto, a forma di bastoncino | Lineare dsDNA | Sulfolobus islandicus rod-shaped virus 1 | |
| Mindivirales | Cystoviridae | Avvolto, sferico | Segmentato dsRNA | |
| Petitvirales | Microviridae | Non avvolto, isometrico | Circolare ssDNA | ΦX174 |
| Tubulavirales | Inoviridae | Non avvolto, filamentoso | Circolare ssDNA | M13 |
| Vinavirales | Corticoviridae | Non avvolto, isometrico | Circolare dsDNA | PM2 |
| Unassigned | Ampullaviridae | Non avvolto, a forma di limone | Lineare dsDNA | |
| Bicaudaviridae | Nonenveloped, lemon-shaped | Circular dsDNA | ||
| Clavaviridae | Nonenveloped, rod-shaped | Circular dsDNA | ||
| Finnlakeviridae | dsDNA | FLiP[14] | ||
| Fuselloviridae | Non avvolto, a forma di bastoncino | Circolare DsDNA | ||
| Globuloviridae | Avvolto, isometrico | Lineare dsDNA | ||
| Guttaviridae | Non avvolto, ovoidale | Circolare dsDNA | ||
| Plasmaviridae | Avvolto, pleomorfo | Circolare dsDNA | ||
| Portogloboviridae | Avvolto, isometrico | Circolare dsDNA | ||
| Spiraviridae | Non avvolto, a forma di bastoncino | Circolare ssDNA | ||
| Tristromaviridae | Avvolto, a forma di bastoncino | Lineare dsDNA |
È stato suggerito che membri della famiglia Picobirnaviridae infettino i batteri ma non i mammiferi.[15] Un'altra famiglia proposta è quella degli "Autolykiviridae" (dsDNA).[16]
Note[modifica | modifica wikitesto]
- ^ Victor Padilla-Sanchez, Structural Model of Bacteriophage T4, in WikiJournal of Science, vol. 4, n. 1, 2021, p. 5, DOI:10.15347/WJS/2021.005. URL consultato il 5 agosto 2021.
- ^ Batteriofago, in Treccani.it – Vocabolario Treccani on line, Roma, Istituto dell'Enciclopedia Italiana.
- ^ a b McGrath S and van Sinderen D (editors)., Bacteriophage: Genetics and Molecular Biology, 1st, Caister Academic Press, 2007, ISBN 978-1-904455-14-1.
- ^ K. E. Wommack e R. R. Colwell, Virioplankton: Viruses in Aquatic Ecosystems, in Microbiology and Molecular Biology Reviews, vol. 64, n. 1, 2000, pp. 69-114, DOI:10.1128/MMBR.64.1.69-114.2000, PMC 98987, PMID 10704475.
- ^ Prescott, L. (1993). Microbiology, Wm. C. Brown Publishers, ISBN 0-697-01372-3
- ^ BBC Horizon (1997): The Virus that Cures – Documentary about the history of phage medicine in Russia and the West
- ^ Borrell, Brendan (August 2012). "Science talk: Phage factor". Scientific American. pp. 80–83.
- ^ E. C. Keen, Phage Therapy: Concept to Cure, in Frontiers in Microbiology, vol. 3, 2012, p. 238, DOI:10.3389/fmicb.2012.00238, PMC 3400130, PMID 22833738.
- ^ Bacteria and bacteriophages collude in the formation of clinically frustrating biofilms, su phys.org.
- ^ Al-Shayeb B, Sachdeva R, Chen LX, Ward F, Munk P, Devoto A, Castelle CJ, Olm MR, Bouma-Gregson K, Amano Y, He C, Méheust R, Brooks B, Thomas A, Lavy A, Matheus-Carnevali P, Sun C, Goltsman DSA, Borton MA, Sharrar A, Jaffe AL, Nelson TC, Kantor R, Keren R, Lane KR, Farag IF, Lei S, Finstad K, Amundson R, Anantharaman K, Zhou J, Probst AJ, Power ME, Tringe SG, Li WJ, Wrighton K, Harrison S, Morowitz M, Relman DA, Doudna JA, Lehours AC, Warren L, Cate JHD, Santini JM, Banfield JF, Clades of huge phages from across Earth's ecosystems, in Nature, vol. 578, n. 7795, febbraio 2020, pp. 425-431, DOI:10.1038/s41586-020-2007-4, PMC 7162821, PMID 32051592.
- ^ Mason, Kenneth A., Jonathan B. Losos, Susan R. Singer, Peter H Raven, and George B. Johnson. (2011). Biology, p. 533. McGraw-Hill, New York. ISBN 978-0-07-893649-4.
- ^ Tolstoy I, Kropinski AM, Brister JR, Bacteriophage Taxonomy: An Evolving Discipline, in Methods Mol. Biol., vol. 1693, 2018, pp. 57-71, DOI:10.1007/978-1-4939-7395-8_6, PMID 29119432.
- ^ Adriaenssens EM, Sullivan MB, Knezevic P, van Zyl LJ, Sarkar BL, Dutilh BE, Alfenas-Zerbini P, Łobocka M, Tong Y, Brister JR, Moreno Switt AI, Klumpp J, Aziz RK, Barylski J, Uchiyama J, Edwards RA, Kropinski AM, Petty NK, Clokie MRJ, Kushkina AI, Morozova VV, Duffy S, Gillis A, Rumnieks J, Kurtböke İ, Chanishvili N, Goodridge L, Wittmann J, Lavigne R, Jang HB, Prangishvili D, Enault F, Turner D, Poranen MM, Oksanen HM, Krupovic M, Taxonomy of prokaryotic viruses: 2018-2019 update from the ICTV Bacterial and Archaeal Viruses Subcommittee, in Arch. Virol., vol. 165, n. 5, maggio 2020, pp. 1253-1260, DOI:10.1007/s00705-020-04577-8, PMID 32162068.
- ^ Elina Laanto, Sari Mäntynen, Luigi De Colibus, Jenni Marjakangas, Ashley Gillum, David I. Stuart, Janne J. Ravantti, Juha Huiskonen, Lotta-Riina Sundberg: Virus found in a boreal lake links ssDNA and dsDNA viruses. In: Proceedings of the National Academy of Sciences 114(31), July 2017, doi:10.1073/pnas.1703834114
- ^ Krishnamurthy SR, Wang D, Extensive conservation of prokaryotic ribosomal binding sites in known and novel picobirnaviruses, in Virology, vol. 516, 2018, pp. 108-114, DOI:10.1016/j.virol.2018.01.006, PMID 29346073.
- ^ Kathryn M. Kauffman, Fatima A. Hussain, Joy Yang, Philip Arevalo, Julia M. Brown, William K. Chang, David VanInsberghe, Joseph Elsherbini, Radhey S. Sharma, Michael B. Cutler, Libusha Kelly, Martin F. Polz: A major lineage of non-tailed dsDNA viruses as unrecognized killers of marine bacteria. In: Nature Vol. 554, pp. 118–122. January 24th, 2018. doi:10.1038/nature25474
Bibliografia[modifica | modifica wikitesto]
- Bacteriophages - StatPearls - NCBI Bookshelf, su ncbi.nlm.nih.gov.
Voci correlate[modifica | modifica wikitesto]
Altri progetti[modifica | modifica wikitesto]
 Wikizionario contiene il lemma di dizionario «batteriofago»
Wikizionario contiene il lemma di dizionario «batteriofago» Wikimedia Commons contiene immagini o altri file su batteriofago
Wikimedia Commons contiene immagini o altri file su batteriofago
Collegamenti esterni[modifica | modifica wikitesto]
- batteriofago, su Treccani.it – Enciclopedie on line, Istituto dell'Enciclopedia Italiana.
- (EN) bacteriophage, su Enciclopedia Britannica, Encyclopædia Britannica, Inc.
| Controllo di autorità | Thesaurus BNCF 20367 · LCCN (EN) sh85010874 · GND (DE) 4125508-2 · BNF (FR) cb119790710 (data) · J9U (EN, HE) 987007284497505171 |
|---|
