Titolazione virale
La titolazione virale o saggio virale o conta virale è il conteggio, fatto in laboratorio, del numero di particelle virali di un dato virus in esame presenti in un campione biologico. Viene utilizzata nei campi della ricerca microbiologica, della diagnostica e della produzione di vaccini antivirali, tutte situazioni che richiedono la conoscenza della quantità di virus in analisi o uso.
Possiamo distinguere le tecniche di titolazione virale in tecniche quantitative e qualitative. I metodi di titolazione quantitative mirano a verificare la presenza di un virus in un dato campione e ad identificare il numero di particelle virali in esso presenti, mentre quelle qualitative mirano a verificare la presenza o l'assenza di determinati effetti del virus in un substrato cellulare.
Un'ulteriore distinzione può essere fatta tra tecniche tradizionali e moderne. Le prime, già in uso da decenni ma generalmente lente e faticose, sono distinte in tecniche di titolazione fisica, biologica e sierologica. Le seconde, nuove e da poco in uso, sono tecniche che riducono il tempo necessario alla titolazione, con una elevata specificità e sensibilità nella rilevazione del virus, ma che attualmente hanno un costo elevato.
Metodi tradizionali[modifica | modifica wikitesto]
I metodi tradizionali comprendono tecniche di titolazione fisica, biologica e sierologica. La titolazione fisica, quantitativa, richiede l'uso di attrezzature complesse e costose e risulta fondamentale nella ricerca della presenza o assenza di virus e loro identificazione in un materiale contaminato. La titolazione biologica, sia quantitativa che qualitativa, si basa sull'osservazione e la valutazione degli effetti biologici specifici prodotti dal virus in esame in un campione biologico, come colture cellulari, colture batteriche, cavie o embrioni di pollo. La titolazione sierologica, qualitativa, sfrutta la capacità dei virus di interagire con i componenti del sangue, in particolare con gli anticorpi e i globuli rossi. La prima sfrutta la specificità antigenica degli anticorpi per i componenti del virione, mentre la seconda sfrutta la capacità di alcuni virus di produrre l'emoagglutinina.
Microscopia[modifica | modifica wikitesto]

Una nuova ed ignota specie virale, che presenti caratteristiche nuove o anomale, ma comunque capace di agire da patogeno, potrebbe non essere rilevabile con i normali metodi di ricerca. In tal caso l'unico metodo certo per rilevare la presenza del virus è l'osservazione diretta delle particelle virali. Tuttavia a causa delle ridotte dimensioni dei virioni (10-300 nm), inferiori al massimo ingrandimento possibile con un microscopio ottico, è necessario l'utilizzo di microscopi più potenti, ingombranti e costosi, come il microscopio elettronico (a scansione o a trasmissione) ed il microscopio a fluorescenza. Tali microscopi permettono di osservare direttamente particelle virali in sospensione all'interno di una goccia di soluzione e di apprezzare preliminarmente le caratteristiche morfologiche del virione (presenza del pericapside, di fibre, di peplomeri o altre strutture esterne, simmetria del capside, forma e dimensione del virione). Il problema principale di questa metodica fisica è che il campo di osservazione è limitato e solo una parte delle particelle virali contenute nel campione risultano visibili e contabili. Per ovviare a ciò, la soluzione di particelle virali a concentrazione ignota viene miscelata ad una soluzione a concentrazione nota di particelle di lattice o polistirene, le cui dimensioni molto superiori a quelle del virus le rendono facilmente riconoscibili. A questo punto un conteggio proporzionale delle particelle virali e delle particelle di lattice permette di risalire alla concentrazione iniziale della soluzione virale.
Saggio delle placche[modifica | modifica wikitesto]
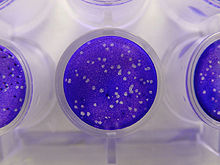
Questo metodo di titolazione biologica quantitativo si basa sull'idea che, infettando con un virus animale una coltura cellulare o con un batteriofago una coltura batterica, la grandezza del focolaio di infezione sia proporzionale al numero di particelle virali che lo ha causato, ossia che maggiore è il numero di particelle virali infettanti inoculate più evidenti e numerosi saranno gli effetti citopatici rilevabili sulla coltura. Questo metodo è utile per rilevare e saggiare i virus in grado di lisare la cellula ospite e quelle che la circondano, producendo le tipiche placche di lisi.
Per la conta si procede infettando varie colture inoculando a ciascuna di esse un volume della soluzione a concentrazione ignota e avendo cura di agitare la provetta per spandere la soluzione su tutta la superficie. Prima dell'inoculo del virus va eliminato il terreno dalla coltura cellulare poiché questo contiene una grande quantità di liquido che influirebbe sulla diffusione delle particelle virali e quindi la si incuba a 37 °C per 45 minuti-1 ora, tempo che permette l'adsorbimento delle particelle virali ed è per questo detto tempo di adsorbimento. Teoricamente, se ogni virus presente nel volume di soluzione infettasse una sola cellula, basterebbe contare il numero di cellule lisate, ossia che sono state infettate, per conoscere il numero di particelle virali in essa presenti e ricavare facilmente la concentrazione virale secondo la formula
.
Tuttavia ciò non è possibile poiché normalmente all'interno dei campioni virali estratti da colture o animali infetti è presente un numero molto elevato di particelle virali, cosicché solitamente più di una infetta contemporaneamente una stessa cellula. Inoltre i nuovi virioni liberati dopo le infezioni primarie andrebbero a produrre per diffusione nuove infezioni su altre cellule anche a grande distanza, rendendo impossibile risalire al numero originale di particelle virali. Per ovviare a questi difetti vengono utilizzati due accorgimenti:
- vengono utilizzate soluzioni molto diluite della soluzione virale originale, in maniera che l'inoculo contenga un numero di particelle virali notevolmente più piccolo rispetto al numero di cellule contenute nella coltura e quindi risulti molto improbabile che due particelle virali infettino la stessa cellula. Inoltre agitare la provetta e spandere la soluzione su tutta la superficie della coltura contribuisce ulteriormente a rendere meno probabile che le particelle virali infettino la stessa cellula o cellule vicine tra loro;
- vengono aggiunte sostanze gelatinose (solitamente gel di agar) alla coltura che la rendono semisolida e impediscono alle nuove particelle virali di infettare cellule lontane dalla prima cellula infettata. Ne consegue che le infezioni secondarie si verificheranno solo nelle vicinanze della sede di infezione primaria e che per ogni particella virale iniziale otterrò una ed una sola placca di lisi sulla coltura. A questo punto le placche possono essere colorate con vari metodi e contate.
Dato che la concentrazione della soluzione virale originale è ignota non posso sapere quanto diluirla per ottenere inoculi adatti alla conta delle placche. Procedo quindi per tentativi infettando varie colture monostrato con un volume costante di soluzioni a diluizione sempre maggiore.
Genericamente, se considero una soluzione a concentrazione iniziale e ne prelevo un volume che diluisco con un volume di solvente, la nuova concentrazione della soluzione diluita sarà
.
Se poi prelevo nuovamente dalla soluzione a concentrazione lo stesso volume e lo diluisco sempre con un volume di solvente , la nuova concentrazione della soluzione sarà
o, sostituendo ,
.
In generale la concentrazione della soluzione ad -esima diluizione della soluzione iniziale a concentrazione sarà
.
Nella pratica si è soliti prelevare un volume = 0,1 ml e diluirlo con un volume = 0,9 ml in modo che
e quindi la concentrazione della soluzione ad -esima diluizione sia
.
Da ognuna delle soluzioni diluite nell'ordine di viene quindi prelevato un volume che viene inoculato alla coltura per produrre le placche di lisi; le soluzioni a concentrazione maggiore produrranno i difetti descritti, rendendo impossibile la conta, mentre quelli a concentrazione adeguata permetteranno di contare le placche e risalire al numero originario di particelle virali presenti nell'inoculo a concentrazione . Solitamente vengono prodotte colture infettate con diluizioni fino a o e tra queste vengono scelte per calcolare la concentrazione iniziale quelle che riportano un numero di placche compreso tra 50 e 70. Ciò perché, per ragioni statistiche, campioni diluiti troppo o troppo poco, e di conseguenza con un numero troppo piccolo o troppo grande di placche, ridurrebbero la sensibilità del metodo utilizzato. Per lo stesso motivo più colture sono infettate con inoculi a stessa diluizione, in modo da avere dei gruppi di controllo con cui confrontare i dati. In questo caso il numero reale di placche sarà considerato il numero medio di placche per coltura.
Quindi, posto il numero medio di placche per coltura prodotte dall'inoculo di un volume di una soluzione diluita volte, la concentrazione di particelle virali di tale soluzione è
.
Ne consegue che la concentrazione iniziale è
.
Poiché nella pratica si è soliti inoculare volumi di soluzione virale diluita nell'ordine del ml e di esprimere il numero di particelle virali in unità formanti placche (plaque forming units, pfu), inteso come l'equivalente numero di virioni necessari a formare una placca, la concentrazione di particelle virali può essere espressa in unità formanti placche per millilitro (pfu/ml).
Saggio di messa a fuoco (FFA)[modifica | modifica wikitesto]
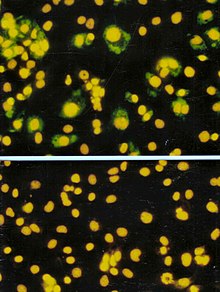
Il saggio di messa a fuoco (focus forming assay, FFA) è una variante del saggio delle placche che invece di basarsi sulla lisi cellulare per rilevare la formazione delle placche impiega tecniche di immunoistochimica che utilizzano anticorpi marcati in maniera fluorescente specifici per un determinato antigene virale per rilevare cellule ospiti infettate e particelle virali libere prima che si formino le placche. L'FFA è utile per quantificare le classi di virus che non lisano le membrane cellulari che altrimenti non sarebbero rilevabili con il normale saggio delle placche. Come il saggio delle placche, i monostrati di cellule ospiti sono infettati da varie diluizioni del campione di virus ed incubati per un periodo di incubazione relativamente breve (24-72 ore) con un terreno di coltura semisolido che limita la diffusione del virus, creando gruppi localizzati (focolai) di cellule infettate. Le piastre vengono successivamente analizzate mediante anticorpi fluorescenti specifici per un determinato antigene virale e il microscopio a fluorescenza viene utilizzato per contare e quantificare il numero di focolai. Il saggio FFA in genere dà risultati in meno tempo rispetto al normale saggio delle placche o al calcolo della dose infettante citopatica (DITC50), ma può essere più costoso in termini di reagenti e apparecchiature richiesti. Il tempo di completamento del test dipende anche dall'estensione dell'area che l'operatore sta contando. Un'area più ampia richiederà più tempo ma può fornire una rappresentazione più accurata del campione. I risultati del saggio FFA sono espressi come unità di messa a fuoco su millilitro (FFU/mL).[1]
Saggio della diluizione limite[modifica | modifica wikitesto]
Il saggio della diluizione limite è un metodo di titolazione biologica quantitativo. Si parte dalla valutazione di un tampone biologico che rappresenta il totale di virus presenti, quindi bisogna praticare una serie di diluizioni per ridurre il numero totale di virus e portarlo a concentrazione nota. Le diluizioni saranno esponenziali su base logaritmica (partendo, quindi, da fino ad almeno alla ); ciò significa che si partirà da un rapporto di 1:10 (1 parte di virus e 9 parti di diluente) e si arriverà a un rapporto di 1:10000000. È consigliabile allestire più pozzetti (circa 4), in maniera tale da avere un risultato quanto più preciso possibile. In ogni pozzetto si inserirà 50 μl di terreno, si diluirà il virus in maniera esponenziale e dopo un po' di tempo si aggiungeranno 100 μl di cellule. In seguito, si andranno ad osservare gli effetti citopatici (se presenti). Il titolo virale è la più alta diluizione del virus (cioè il più piccolo numero di particelle virali) in grado di infettare il 50% delle unità inoculate e corrisponde a 1 dose infettante citopatica (DITC50).
Saggi proteici[modifica | modifica wikitesto]
Saggio di emoagglutinazione[modifica | modifica wikitesto]

Molti virus con envelope sono dotati di glicoproteine di superficie (peplomeri) note come emoagglutinine. Queste sono importanti recettori virali capaci di legare residui di acido sialico presenti sulla membrana cellulare e quindi di promuovere sia il legame alla cellula bersaglio che l'emoagglutinazione.
Quando si inocula un virus emoagglutinante in una coltura cellulare, durante il ciclo replicativo le emoagglutinine vengono esposte sulla superficie delle cellule infette per essere incluse, assieme a parte della membrana cellulare, nel virione maturo. Quindi se si aggiungono globuli rossi di una appropriata specie animale (solitamente di coniglio o di cavallo, ma anche di topi geneticamente ricombinati umanizzati) alle colture in cui il virus si sta replicando, queste si legheranno tenacemente alla superficie delle cellule infette, grazie alla particolare ricchezza di acido sialico della superficie dei globuli rossi. Si è soliti definire questa proprietà delle colture infette di trattenere i globuli rossi emoadsorbimento.
Sfruttando l'emoadsorbimento è quindi possibile rilevare la presenza di virus emoadsorbenti nella coltura inoculata già alcuni giorni prima della comparsa dell'effetto citopatico. L'identificazione può essere confermata mediante test di inibizione dell'emoagglutinazione, di inibizione dell'emoadsorbimento o immunofluorescenza.
Saggio con anticorpi antivirali specifici[modifica | modifica wikitesto]
Gli anticorpi sono in grado di riconoscere in modo altamente specifico gli antigeni virali e pertanto possono essere usati per l'identificazione e la quantificazione delle particelle virali presenti in un campione. Le tecniche basate su anticorpi antivirali più utilizzate sono l'immunofluorescenza e i test di inibizione.
Saggio dell'acido bicinconinico[modifica | modifica wikitesto]
Il saggio dell'acido bicinconinico si basa su una misurazione colorimetrica ed è il test di quantificazione delle proteine più comune. Nel saggio dell'acido bicinconinico i legami peptidici di una proteina riducono quantitativamente Cu2+ a Cu1+, che produce un colore blu chiaro. L'acido bicinconinico (BCA) chela il Cu1+ con un rapporto di 2:1 risultante in specie più intensamente colorate che assorbono a 562 nm. L'assorbanza di un campione a 562 nm viene utilizzata per determinare la concentrazione di proteine sfuse nel campione. I risultati del test vengono confrontati con le curve standard note dopo l'analisi con uno spettrofotometro o un lettore di piastre.[2] Il tempo totale del test va da 30 minuti a un'ora. Sebbene questo test sia veloce, manca di specificità poiché conta tutte le proteine; la preparazione del virus da quantificare deve quindi contenere livelli molto bassi di proteine delle cellule ospiti.
Saggio di immunodiffusione radiale singola (SRID)[modifica | modifica wikitesto]
Il saggio di immunodiffusione radiale singola (SRID) è un saggio proteico che rileva la quantità di antigene virale specifico mediante immunodiffusione in un terreno di coltura semisolido (ad esempio agar). Il terreno contiene un antisiero specifico per l'antigene target e l'antigene viene posizionato al centro del disco. Quando l'antigene si diffonde nel mezzo crea un anello precipitato che cresce fino al raggiungimento dell'equilibrio. Il tempo di analisi può variare da 10 ore a giorni a seconda del tempo di raggiungimento dell'avvenuta reazione antigene-anticorpo. Il diametro della zona dall'anello è correlato linearmente al logaritmo della concentrazione proteica e comparato con i diametri della zona per standard proteici noti per la quantificazione.[3]
Metodi moderni[modifica | modifica wikitesto]
Rilevazione di impulsi resistivi sintonizzabili (TRPS)[modifica | modifica wikitesto]

La rilevazione di impulsi resistivi sintonizzabili (tunable resistive pulse sensing, TRPS) è un metodo che consente misurazioni ad alta velocità di singole particelle virali di virus, in quanto guidate attraverso un nanoporo di diametro regolabile, una alla volta.[4] La tecnica ha il vantaggio di determinare simultaneamente la dimensione e la concentrazione delle particelle virali in soluzione ad alta risoluzione. Questo metodo può essere usato per valutare la stabilità del campione e il contributo degli aggregati, nonché la concentrazione totale di particelle virali (vp/mL).[5]
La misurazione basata su TRPS avviene in un tampone ionico e non è richiesta alcuna precolorazione dei campioni prima dell'analisi, quindi la tecnica è più rapida di quelle che richiedono un pretrattamento con coloranti fluorescenti, con un tempo totale di preparazione e misurazione inferiore a 10 minuti per campione.
Citometria a flusso[modifica | modifica wikitesto]
Esistono alcuni tipi di citometri a flusso che possono essere utilizzati per la quantificazione dei virus. Un contatore di virus quantifica il numero di particelle virali intatte in un campione sfruttando la fluorescenza per rilevare proteine e acidi nucleici colocalizzati. I campioni vengono colorati con due coloranti, uno specifico per le proteine e uno specifico per gli acidi nucleici, e analizzati mentre fluiscono attraverso un raggio laser. La quantità di particelle virali che producono eventi simultanei su ciascuno dei due distinti canali di fluorescenza viene determinata, insieme alla portata misurata del campione, per calcolare una concentrazione di particelle virali (vp/mL).[6] I risultati sono simili in quantità assoluta a quelli ottenuti con un microscopio elettronico a trasmissione. Il dosaggio ha un intervallo di lavoro lineare di 105-109 vp/mL e un tempo di analisi di circa 10 minuti con un breve tempo di preparazione del campione.
Reazione a catena quantitativa della polimerasi (qPCR)[modifica | modifica wikitesto]
La PCR quantitativa utilizza il meccanismo della reazione a catena della polimerasi per amplificare il DNA o l'RNA virali per produrre concentrazioni sufficientemente elevate per il rilevamento e la quantificazione mediante fluorescenza. In generale, la quantificazione mediante qPCR si basa su diluizioni seriali di standard di concentrazione nota analizzati in parallelo con campioni sconosciuti per calibrazione e riferimento. Il rilevamento quantitativo può essere ottenuto utilizzando un'ampia varietà di strategie di rilevamento della fluorescenza, comprese sonde specifiche per sequenza o coloranti fluorescenti non specifici come il SYBR Green.[7] Sonde specifiche per la sequenza, come TaqMan, Molecular Beacons o Scorpion, si legano solo al DNA della sequenza appropriata prodotta durante la reazione. La metodica amplifica tutto l'acido nucleico bersaglio, compreso quello originato da particelle virali infettive intatte o da particelle virali difettose e quello libero in soluzione. Per la quantificazione virale il rapporto tra virioni interi e copie di acido nucleico è raramente 1:1. Questo perché durante la replicazione virale, l'acido nucleico e le proteine virali non sono sempre prodotti in rapporto 1:1 e il processo di assemblaggio virale produce virioni completi, capsidi vuoti e/o eccesso di genomi virali liberi. I vantaggi della qPCR comprendono tempi di risposta rapidi (1-4 ore) e notevole sensibilità che permette il rilevamento di una concentrazione di virus molto più bassa rispetto ad altri metodi.
ELISA[modifica | modifica wikitesto]

L'ELISA è una variante moderna dei saggi proteici che utilizza un anticorpo specifico legato ad un enzima per rilevare la presenza di una quantità sconosciuta di antigene (in questo caso un virus) in un campione. L'avvenuta reazione antigene-anticorpo (ovvero la formazione di un immunocomplesso) viene rilevata e/o quantificata attraverso la capacità dell'enzima di convertire un reagente in un segnale rilevabile che può essere utilizzato per calcolare la concentrazione dell'antigene nel campione.[8] La perossidasi di rafano (HRP) è un enzima comunemente utilizzato nei saggi ELISA grazie alla sua capacità di amplificare il segnale e aumentare la sensibilità del dosaggio. Esistono molte tipologie di saggi ELISA, classificati come diretti, indiretti, competitivi o sandwich.[9] Questa tecnica richiede meno lavoro rispetto ai metodi tradizionali e può richiedere dalle 4 alle 24 ore in base al tempo di incubazione degli anticorpi.
Note[modifica | modifica wikitesto]
- ^ Flint S.J., Enquist W., Racaniello V.R., Skalka A.M. (2009), Virological Methods, in Principles of Virology, ASM Press, ISBN 978-1-55581-443-4.
- ^ Pierce Protein Biology.
- ^ Rodda S.J., Gallichio H.A., Hampson A.W. (1981), The Single Radial Immunodiffusion Assay Highlights Small Antigenic Differences Among Influenza Virus Hemagglutinins, in Journal of Clinical Microbiology, vol. 14, 5ª pubblicazione.
- ^ Stephen J. Sowerby, Murray F. Broom, George B. Petersen, Dynamically resizable nanometre-scale apertures for molecular sensing, in Sensors and Actuators B, vol. 123, 1ª edizione, 2007, pp. 325-330.
- ^ G. Seth Roberts, Sam Yu, Qinglu Zeng, Leslie C.L. Chan, Will Anderson, Aaron H. Colby, Mark W. Grinstaff, Steven Reid, Robert Vogel, Tunable Pores for Measuring Concentrations of Synthetic and Biological Nanoparticle Dispersions, in Biosensors and Bioelectronics, vol. 31, 2012, pp. 17-25.
- ^ Stoffel C.L., Finch R., Christensen K., Edwards D., Rowlen K.L. (2005), Rapid Determination of Baculovirus Titer by a Dual Channel Virus Counter, in American Biotechnology Laboratory, vol. 37, 22ª pubblicazione, pp. 24-25.
- ^ PCR/Real-Time PCR Protocols.
- ^ Kemeny D.M., Challacombe S.J. (1988), ELISA and Other Solid Phase Immunoassays: Theoretical and Practical Aspects, John Wiley and Sons, ISBN 0-471-90982-3.
- ^ Kuby J., Kindt T.J., Goldsby R.A., Osborne B.A. (2007), Kuby Immunology 6th edition, W.H. Freeman and Company, ISBN 978-1-4292-0211-4.























