Proteine intrinseche di membrana
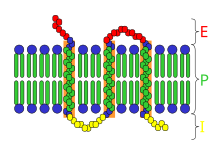
Una proteina di membrana intrinseca (conosciuta anche come proteina di membrana integrale - IMP) è un tipo di proteina di membrana che è permanentemente collegata alla membrana biologica. Tutte le proteine transmembrana sono proteine di membrana intrinseche, ma non tutte le proteine di membrana intrinseche sono proteine transmembrana.[1] Le proteine di membrana intrinseche comprendono una frazione significativa delle proteine codificate nel genoma di un organismo.[2] Le proteine che attraversano la membrana sono circondate da lipidi "anulari" che costituiscono una sorta di guscio lipidico anulare. I lipidi anulari sono quelle molecole lipidiche che sono in contatto diretto con una proteina di membrana. Tali proteine possono essere separate dalle membrane biologiche usando solo detersivi, solventi non polari o talvolta agenti denaturanti.
Struttura[modifica | modifica wikitesto]

La membrana è rappresentata in marrone chiaro.
Tramite metodi di cristallografia a raggi X o spettroscopia di risonanza magnetica nucleare sono state determinate nella loro risoluzione atomica le strutture tridimensionali di ~ 160 diverse proteine di membrana intrinseche. Lo studio di queste proteine si è rivelato di particolare complessità soprattutto a causa delle difficoltà associate all'estrazione e alla cristallizzazione delle stesse. Inoltre, le strutture di molti domini proteici solubili in acqua di proteine di membrana intrinseche sono disponibili nel Protein Data Bank (PDB). Le loro eliche che le ancorano alla membrana sono state rimosse per facilitare l'estrazione e la cristallizzazione. È quindi possibile ricercare una particolare proteine di membrana integrale nel PPB (il quale si basa su una classificazione di ontologia genetica). Le proteine di membrana integrale possono essere divisi in due gruppi:
- Proteine polipropiche intrinseche (proteine transmembrana)
- Proteine monotopiche intrinseche
Proteine polipeptiche integrali (proteine transmembrana)[modifica | modifica wikitesto]
Il tipo più comune di proteine di membrana integrale è la proteina transmembrana (TM), che copre l'intera membrana biologica. Le proteine di membrana ‘’a passaggio singolo’’ attraversano la membrana solo una volta, mentre le proteine di membrana ‘’a più passaggi’’ si intrecciano tra loro e si incrociano più volte. Le proteine transmembrana a passaggio singolo possono essere classificate come: tipo I (posizionate in modo tale che il loro carbossile-terminale sia rivolto verso il citosol) o tipo II (posizionate con la porzione amino-terminale verso il citosol). Le proteine di tipo III hanno domini transmembrana multipli in un singolo polipeptide, mentre il tipo IV è costituito da diversi polipeptidi assemblati insieme in un canale attraverso la membrana. Le proteine di tipo V sono invece ancorate al doppio strato lipidico attraverso lipidi legati in modo covalente. Infine le proteine di tipo VI presentano sia un dominio transmembrana che degli ancoraggi lipidici.[3]
Proteine monotopiche integrali[modifica | modifica wikitesto]
Le proteine monotopiche integrali sono associate alla membrana da un lato ma non si estendono completamente attraverso il doppio strato lipidico.
Determinazione della struttura proteica[modifica | modifica wikitesto]
La Protein Structure Initiative (PSI), finanziata dall'Istituto Nazionale Americano di Scienze Mediche Generali (NIGMS), parte del National Institutes of Health (NIH), ha tra i suoi scopi quello di determinare le strutture proteiche tridimensionali e di sviluppare tecniche per l'uso in biologia strutturale, incluso per proteine di membrana. Il modello di omologia può essere utilizzato per costruire un modello di risoluzione atomica della proteina integrale "target" a partire dalla sua sequenza di aminoacidi e una struttura tridimensionale sperimentale di una proteina omologa correlata. Questa procedura è stata ampiamente utilizzata per i ligandi dei recettori accoppiati a proteine G (o GPCR) e i loro complessi.[4]
Funzione[modifica | modifica wikitesto]
Le proteine intrinseche di membrana comprendono trasportatori, proteine adattatrici, canali, recettori, enzimi, domini di ancoraggio strutturali della membrana, proteine coinvolte nell'accumulo e trasduzione di energia e proteine responsabili dell'adesione cellulare. La classificazione dei trasportatori può essere trovata nella classificazione TC nota anche con il nome di Transporter Classification Database.[5] Un esempio della relazione tra le proteine intrinseche di membrana (in questo caso il pigmento batterico fotoassorbente, la batteriorodopsina) e la membrana formata dal doppio strato di fosfolipidi è illustrato di seguito. In questo caso la proteina di membrana integrale copre il doppio strato di fosfolipidi per sette volte. La parte della proteina che è incorporata nelle regioni idrofobiche del doppio strato è alfa elicoidale e composta da aminoacidi prevalentemente idrofobici. L'estremità terminale amminica della proteina è nel citosol mentre la regione terminale C si trova all'esterno della cellula. Una membrana che contiene questa particolare proteina è in grado di funzionare nella fotosintesi.[6]
Note[modifica | modifica wikitesto]
- ^ Steven R. Goodman, Medical cell biology, Academic Press, 2008, pp. 37–, ISBN=978-0-12-370458-0. URL consultato il 30 aprile 2018.
- ^ Wallin E, von Heijne G, Genome-wide analysis of integral membrane proteins from eubacterial, archaean, and eukaryotic organisms, in Protein Science, vol. 7, n. 4, 1998, pp. 1029–38, DOI:10.1002/pro.5560070420, PMC 2143985, PMID 9568909.
- ^ David L. Nelson, Michael Cox, Principles of Biochemistry, 7ª edizione, WH Freeman, 2017, ISBN 978-1-319-10824-3.
- ^ Marquer C, Fruchart-Gaillard C, Letellier G, Marcon E, Mourier G, Zinn-Justin S, Ménez A, Servent D, Gilquin B, Structural model of ligand-G protein-coupled receptor (GPCR) complex based on experimental double mutant cycle data: MT7 snake toxin bound to dimeric hM1 muscarinic receptor, in J. Biol. Chem., vol. 286, n. 36, Settembre 2011, pp. 31661–75, DOI:10.1074/jbc.M111.261404, PMC 3173127, PMID 21685390. URL consultato il 30 aprile 2018.
- ^ Saier MH, Yen MR, Noto K, Tamang DG, Elkan C, The Transporter Classification Database: recent advances, in Nucleic Acids Res., vol. 37, Database issue, Gennaio 2009, pp. D274–8, DOI:10.1093/nar/gkn862, PMC 2686586, PMID 19022853. URL consultato il 30 aprile 2018.
- ^ Integral membrane proteins, su academic.brooklyn.cuny.edu. URL consultato il 30 aprile 2015 (archiviato dall'url originale il 1º febbraio 2015).
Altri progetti[modifica | modifica wikitesto]
 Wikimedia Commons contiene immagini o altri file su Proteine intrinseche di membrana
Wikimedia Commons contiene immagini o altri file su Proteine intrinseche di membrana
Collegamenti esterni[modifica | modifica wikitesto]
- (EN) intrinsic protein, su Enciclopedia Britannica, Encyclopædia Britannica, Inc.