Utente:Anonymus07*5/Traduzione eucariotica
La traduzione eucariotica è il processo biologico attraverso il quale l'RNA messaggero viene tradotto in proteine negli eucarioti. È composto da quattro fasi: inizio, allungamento, terminazione e riciclo.
Inizio[modifica | modifica wikitesto]

Avvio dipendente dal cappuccio[modifica | modifica wikitesto]
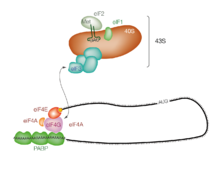
L'inizio della traduzione di solito implica l'interazione di alcune proteine chiave, i fattori di iniziazione, con un'etichetta speciale legata all'estremità 5' di una molecola di mRNA, il cappuccio 5' , nonché con l' UTR 5' . Queste proteine si legano alla subunità ribosomiale piccola (40S) e mantengono in posizione l'mRNA. eIF3 è associato alla subunità ribosomiale 40S e svolge un ruolo nel mantenere la subunità ribosomiale grande ( 60S ) dal legame prematuro. eIF3 interagisce anche con il complesso eIF4F , che consiste IN altri tre fattori di inizio: eIF4A , eIF4E ed eIF4G . eIF4G è una proteina di impalcatura che si associa direttamente sia a eIF3 che agli altri due componenti. eIF4E è la proteina legante il cappuccio. Il legame del cap da parte di eIF4E è spesso considerato il passo limitante della velocità di inizio dipendente dal cap e la concentrazione di eIF4E è un nesso normativo del controllo traslazionale. Alcuni virus fendono una porzione di eIF4G che lega eIF4E, impedendo così alla traduzione dipendente da cap per dirottare il meccanismo host a favore dei messaggi virali (cap-indipendent). eIF4A è una RNA elicasi ATP-dipendente che aiuta il ribosoma risolvendo alcune strutture secondarie formate lungo il trascritto dell'mRNA. La proteina legante il poli (A) (PABP) si associa anche con eIF4Fcomplesso tramite eIF4G e lega la coda poli-A della maggior parte delle molecole di mRNA eucariotico. Questa proteina è stata implicata nel giocare un ruolo nella circolarizzazione dell'mRNA durante la traduzione. Questo complesso di preiniziazione 43S (43S PIC) accompagnato dai fattori proteici si muove lungo la catena dell'mRNA verso la sua estremità 3 ', in un processo noto come "scansione", per raggiungere il codone iniziale (tipicamente AUG). Negli eucarioti e negli archei , l' amminoacido codificato dal codone iniziale è la metionina . Il tRNA iniziatore a carica Met (Met-tRNA i Met ) viene portato al sito P della piccola subunità ribosomiale dafattore di iniziazione eucariotica 2 (eIF2) . Idrolizza GTP e segnala la dissociazione di diversi fattori dalla subunità ribosomiale piccola, portando infine all'associazione della subunità grande (o subunità 60S ). Il ribosoma completo ( 80S ) inizia quindi l'allungamento di traslazione. La regolazione della sintesi proteica è in parte influenzata dalla fosforilazione di eIF2 (tramite la subunità α), che fa parte del complesso ternario eIF2-GTP-Met-tRNA i Met (eIF2-TC). Quando un gran numero di eIF2 viene fosforilato, la sintesi proteica viene inibita. Ciò si verifica durante la fame di aminoacidi o dopo l'infezione virale. Tuttavia, una piccola frazione di questo fattore di iniziazione è naturalmente fosforilata. Un altro regolatore è4EBP , che si lega al fattore di inizio eIF4E e inibisce le sue interazioni con eIF4G , prevenendo così l'inizio dipendente dal cap. Per contrastare gli effetti del 4EBP, i fattori di crescita fosforilano il 4EBP, riducendone l'affinità per eIF4E e consentendo la sintesi proteica. Mentre la sintesi proteica è regolata globalmente modulando l'espressione dei fattori chiave di inizio e il numero di ribosomi, i singoli mRNA possono avere velocità di traduzione diverse a causa della presenza di elementi di sequenza regolatrice. Ciò si è dimostrato importante in una varietà di contesti, tra cui la meiosi del lievito e la risposta dell'etilene nelle piante. Inoltre, recenti lavori su lieviti e esseri umani suggeriscono che la divergenza evolutiva nelle sequenze di regolazione cis può influire sulla regolazione della traduzione. Inoltre, elicasi di RNA come DHX29 e Ded1 / DDX3 possono partecipare al processo di inizio della traduzione, specialmente per mRNA con 5'UTR strutturati.
Avvio indipendente dal cappuccio[modifica | modifica wikitesto]
L'esempio meglio studiato di inizio della traduzione indipendente dal cappuccio negli eucarioti utilizza il sito di ingresso dei ribosomi interni (IRES). A differenza della traduzione dipendente da cap, la traduzione indipendente da cap non richiede un cap da 5 'per avviare la scansione dall'estremità 5' dell'mRNA fino al codone iniziale. Il ribosoma può localizzarsi nel sito di inizio mediante legame diretto, fattori di inizio e / o ITAF (fattori trans-attivi IRES) aggirando la necessità di scansionare l'intero 5 'UTR . Questo metodo di traduzione è importante in condizioni che richiedono la traduzione di mRNA specifici durante lo stress cellulare, quando la traduzione complessiva è ridotta. Gli esempi includono fattori che rispondono all'apoptosi e risposte indotte dallo stress.
Allungamento[modifica | modifica wikitesto]
L'allungamento dipende dai fattori di allungamento eucariotico . Alla fine della fase di iniziazione, l'mRNA è posizionato in modo che il codone successivo possa essere tradotto durante la fase di allungamento della sintesi proteica. Il tRNA iniziatore occupa il sito P nel ribosomae il sito A è pronto per ricevere un amminoacil-tRNA. Durante l'allungamento della catena, ogni amminoacido aggiuntivo viene aggiunto alla catena polipeptidica nascente in un microciclo a tre fasi. I passaggi in questo microciclo sono (1) posizionare l'amminoacil-tRNA corretto nel sito N del ribosoma, che viene portato in quel sito da eIF2, (2) formare il legame peptidico e (3) spostare l'mRNA di un parente codone al ribosoma. A differenza dei batteri, in cui l'inizio della traduzione avviene non appena viene sintetizzata l'estremità 5 'di un mRNA, negli eucarioti non è possibile un accoppiamento così stretto tra trascrizione e traduzione perché la trascrizione e la traduzione sono effettuate in compartimenti separati della cellula (il nucleo e citoplasma ). I precursori dell'mRNA eucariotico devono essere elaborati nel filenucleo (p. es., capping, poliadenilazione , splicing) prima che vengano esportati nel citoplasma per la traduzione. La traduzione può anche essere influenzata dalla pausa ribosomiale , che può innescare un attacco endonucleolitico del tRNA, un processo chiamato decadimento senza via dell'mRNA. La pausa ribosomiale aiuta anche il ripiegamento co-traduzionale del polipeptide nascente sul ribosoma e ritarda la traduzione delle proteine mentre codifica il tRNA. Questo può innescare il frameshifting ribosomiale.
Terminazione[modifica | modifica wikitesto]
La fine dell'allungamento dipende dai fattori di rilascio eucariotico . Il processo è simile a quello della terminazione batterica , ma a differenza della terminazione batterica, esiste un fattore di rilascio universale , eRF1, che riconosce tutti e tre i codoni di stop. Al termine, il ribosoma viene smontato e il polipeptide completato viene rilasciato. eRF3 è una GTPasi dipendente dal ribosoma che aiuta eRF1 a rilasciare il polipeptide completato. Il genoma umano codifica per alcuni geni il cui codone di arresto dell'mRNA presenta sorprendentemente perdite: in questi geni, la terminazione della traduzione è inefficiente a causa delle speciali basi di RNA nelle vicinanze del codone di arresto. La terminazione che perde in questi geni porta alla lettura traslazionalefino al 10% dei codoni di stop di questi geni. Alcuni di questi geni codificano domini proteici funzionali nella loro estensione readthrough in modo che possano sorgere nuove isoforme proteiche . Questo processo è stato definito "readthrough traslazionale funzionale".
Note[modifica | modifica wikitesto]
Voci correlate[modifica | modifica wikitesto]
Collegamenti esterni[modifica | modifica wikitesto]
Categoria:Eucarioti Categoria:Proteine Categoria:RNA Categoria:Traduzione